valeurs de réglage de ntree et mtry pour le modèle de régression de la forêt aléatoire
j'utilise R package randomForest pour faire une régression sur certaines données biologiques. Ma taille de données de formation est 38772 X 201.
je me demandais---ce serait une bonne valeur pour le nombre d'arbres <!-Et le nombre de variables par niveau mtry? Est-il une formule approximative pour trouver de telles valeurs de paramètre?
Chaque ligne de mes données d'entrée est de 200 caractères représentant la séquence d'acides aminés, et je veux construire un modèle de régression à utiliser une telle séquence afin de prédire les distances entre les protéines.
5 réponses
La valeur par défaut pour mtry est tout à fait raisonnable, donc il n'y a pas vraiment besoin de coucher avec elle. Il y a une fonction tuneRF pour optimiser ce paramètre. Cependant, soyez conscient que cela peut causer de la partialité.
Il n'y a pas d'optimisation pour le nombre de bootstrap réplique. Je commence souvent avec ntree=501 et ensuite tracer l'objet forestier au hasard. Cela vous montrera la convergence d'erreurs basée sur l'erreur OOB. Vous voulez assez d'arbres pour stabiliser l'erreur, mais pas tant que vous sur corrélez l'ensemble, qui mène à l'overfit.
Voici la mise en garde: les interactions de variables se stabilisent à un rythme plus lent que l'erreur, donc, si vous avez un grand nombre de variables indépendantes, vous avez besoin de plus de répliques. Je garderais un nombre impair pour briser les liens.
Pour les dimensions de votre problème, je commencerais ntree=1501. Je recommande également de consulter l'une des méthodes de sélection des variables publiées afin de réduire le nombre de vos variables indépendantes.
La réponse courte est non.
randomForest la fonction a bien sûr des valeurs par défaut pour les deux ntree et mtry. La valeur par défaut de mtry est souvent (mais pas toujours) raisonnable, alors que généralement les gens voudront augmenter ntree il est par défaut de 500 tout à fait un peu.
la valeur "correcte" pour ntree généralement n'est pas très préoccupant, car il sera tout à fait évident avec un peu de bricolage que les prédictions du modèle ne changeront pas beaucoup après un certain nombre d'arbres.
vous pouvez passer (lire: gaspiller) beaucoup de temps à bricoler avec des choses comme mtry (et sampsize et maxnodes et nodesize etc.), probablement pour un certain bénéfice, mais d'après mon expérience pas beaucoup. Cependant, chaque ensemble de données sera différent. Parfois, vous pouvez voir une grande différence, parfois pas du tout.
signe le paquet a une fonction très générale train cela vous permet de faire une recherche de grille simple sur les valeurs des paramètres comme mtry pour une grande variété de modèles. Ma seule mise en garde serait que faire cela avec des ensembles de données assez grands est susceptible d'obtenir la consommation de temps assez rapidement, alors attention à cela.
Aussi, j'ai oublié que le ranfomForest le paquet lui-même a un tuneRF fonction spécifique à la recherche de la valeur" optimale " pour mtry.
puisse ce livre aider ? Limiter le Nombre d'Arbres dans les Forêts Aléatoires
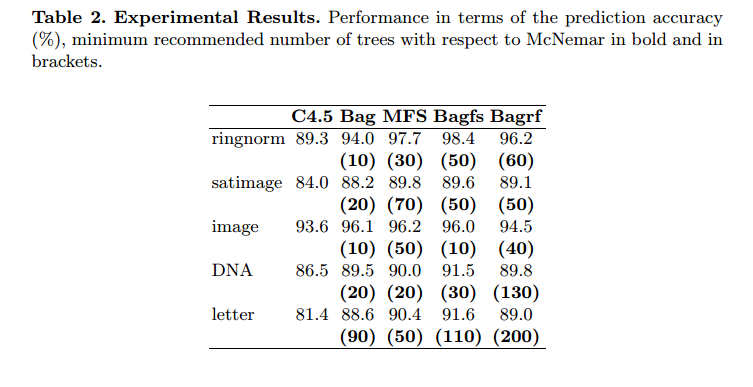
Abstrait. L'objectif de ce papier est de proposer une procédure simple qui a priori détermine un nombre minimum de classificateurs pour combiner afin pour obtenir un niveau de précision de prédiction similaire à celui obtenu avec le combinaison d'ensembles plus grands. La procédure est basée sur le McNemar test de signification non paramétrique. Connaître a priori le minimum la taille de l'ensemble Classificateur donnant la meilleure précision de prédiction, constitue un gain de temps et de mémoire surtout pour les bases de données énormes et applications en temps réel. Ici, nous avons appliqué cette procédure à quatre plusieurs systèmes de classificateur avec C4.5 arbre de décision Les sous-espaces aléatoires, leur combinaison nous avons étiqueté 'Bagfs', et Breiman's Forêts aléatoires) et cinq grandes bases de données de référence. Il vaut la peine de remarquer que la procédure proposée peut être facilement étendue à d'autres base apprendre des algorithmes qu'un arbre de décision aussi bien. Les résultats expérimentaux a montré qu'il est possible de limiter considérablement le nombre d'arbres. Nous a également montré que le nombre minimum d'arbres requis pour obtenir la meilleure précision de prédiction peut varier d'une combinaison de classificateurs méthode à l'autre
ils n'utilisent jamais plus de 200 arbres.
j'utilise le code ci-dessous pour vérifier que j'ai jouer avec ntree et mtry (modifier les paramètres):
results_df <- data.frame(matrix(ncol = 8))
colnames(results_df)[1]="No. of trees"
colnames(results_df)[2]="No. of variables"
colnames(results_df)[3]="Dev_AUC"
colnames(results_df)[4]="Dev_Hit_rate"
colnames(results_df)[5]="Dev_Coverage_rate"
colnames(results_df)[6]="Val_AUC"
colnames(results_df)[7]="Val_Hit_rate"
colnames(results_df)[8]="Val_Coverage_rate"
trees = c(50,100,150,250)
variables = c(8,10,15,20)
for(i in 1:length(trees))
{
ntree = trees[i]
for(j in 1:length(variables))
{
mtry = variables[j]
rf<-randomForest(x,y,ntree=ntree,mtry=mtry)
pred<-as.data.frame(predict(rf,type="class"))
class_rf<-cbind(dev$Target,pred)
colnames(class_rf)[1]<-"actual_values"
colnames(class_rf)[2]<-"predicted_values"
dev_hit_rate = nrow(subset(class_rf, actual_values ==1&predicted_values==1))/nrow(subset(class_rf, predicted_values ==1))
dev_coverage_rate = nrow(subset(class_rf, actual_values ==1&predicted_values==1))/nrow(subset(class_rf, actual_values ==1))
pred_prob<-as.data.frame(predict(rf,type="prob"))
prob_rf<-cbind(dev$Target,pred_prob)
colnames(prob_rf)[1]<-"target"
colnames(prob_rf)[2]<-"prob_0"
colnames(prob_rf)[3]<-"prob_1"
pred<-prediction(prob_rf$prob_1,prob_rf$target)
auc <- performance(pred,"auc")
dev_auc<-as.numeric(auc@y.values)
pred<-as.data.frame(predict(rf,val,type="class"))
class_rf<-cbind(val$Target,pred)
colnames(class_rf)[1]<-"actual_values"
colnames(class_rf)[2]<-"predicted_values"
val_hit_rate = nrow(subset(class_rf, actual_values ==1&predicted_values==1))/nrow(subset(class_rf, predicted_values ==1))
val_coverage_rate = nrow(subset(class_rf, actual_values ==1&predicted_values==1))/nrow(subset(class_rf, actual_values ==1))
pred_prob<-as.data.frame(predict(rf,val,type="prob"))
prob_rf<-cbind(val$Target,pred_prob)
colnames(prob_rf)[1]<-"target"
colnames(prob_rf)[2]<-"prob_0"
colnames(prob_rf)[3]<-"prob_1"
pred<-prediction(prob_rf$prob_1,prob_rf$target)
auc <- performance(pred,"auc")
val_auc<-as.numeric(auc@y.values)
results_df = rbind(results_df,c(ntree,mtry,dev_auc,dev_hit_rate,dev_coverage_rate,val_auc,val_hit_rate,val_coverage_rate))
}
}
un truc sympa que j'utilise est de commencer par prendre la racine carrée du nombre de prédicteurs et de brancher cette valeur pour "mtry". Il est généralement autour de la même valeur que tunerf funtion dans la forêt aléatoire serait choisir.