Équations SVM du paquet e1071 r?
je suis intéressé pour tester les performances des SVM à classer plusieurs individus en quatre groupes/classes. En utilisant la fonction svmtrain LibSVM de MATLAB, je suis en mesure d'obtenir les trois équations utilisées pour classer ces individus parmi les 4 groupes, sur la base des valeurs de cette équation. Un tel programme pourrait être comme suit:
All individuals (N)*
|
Group 1 (n1) <--- equation 1 ---> (N-n1)
|
(N-n1-n2) <--- equation 2 ---> Group 2 (n2)
|
Group 3 (n3) <--- equation 3 ---> Group 4(n4)
*N = n1+n2+n3+n4
y a-t-il un moyen d'obtenir ces équations en utilisant la fonction svm dans le paquet e1071 r?
1 réponses
svme1071 utilise la stratégie" un contre un " pour la classification multiclasse (c'est-à-dire la classification binaire entre toutes les paires, suivie du vote). Donc, pour gérer cette configuration hiérarchique, vous avez probablement besoin de faire une série de classificateurs binaires manuellement, comme group 1 vs. all, puis group 2 vs. whatever is left, etc.. En outre, la base svm la fonction n'accorde pas les hyperparamètres, donc vous voudrez typiquement utiliser un wrapper comme tunee1071, ou train dans le excellent caret package.
quoi qu'il en soit, pour classer les nouveaux individus en R, Vous n'avez pas à entrer des nombres dans une équation manuellement. Plutôt, vous utilisez le predict fonction générique, qui a des méthodes pour différents modèles comme SVM. Pour les objets de modèle comme celui-ci, vous pouvez aussi généralement utiliser les fonctions génériques plot et summary. Voici un exemple de L'idée de base en utilisant un SVM linéaire:
require(e1071)
# Subset the iris dataset to only 2 labels and 2 features
iris.part = subset(iris, Species != 'setosa')
iris.part$Species = factor(iris.part$Species)
iris.part = iris.part[, c(1,2,5)]
# Fit svm model
fit = svm(Species ~ ., data=iris.part, type='C-classification', kernel='linear')
# Make a plot of the model
dev.new(width=5, height=5)
plot(fit, iris.part)
# Tabulate actual labels vs. fitted labels
pred = predict(fit, iris.part)
table(Actual=iris.part$Species, Fitted=pred)
# Obtain feature weights
w = t(fit$coefs) %*% fit$SV
# Calculate decision values manually
iris.scaled = scale(iris.part[,-3], fit$x.scale[[1]], fit$x.scale[[2]])
t(w %*% t(as.matrix(iris.scaled))) - fit$rho
# Should equal...
fit$decision.values
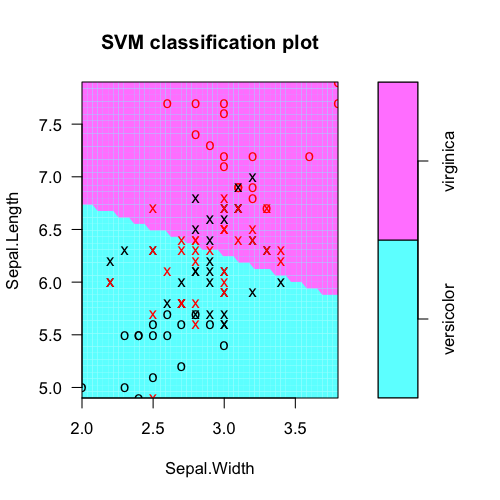
tabuler les étiquettes de classe réelles vs les prédictions du modèle:
> table(Actual=iris.part$Species, Fitted=pred)
Fitted
Actual versicolor virginica
versicolor 38 12
virginica 15 35
extraire les poids Caractéristiques de svm objet modèle (pour la sélection des fonctionnalités, etc.). Ici,Sepal.Length est évidemment plus utile.
> t(fit$coefs) %*% fit$SV
Sepal.Length Sepal.Width
[1,] -1.060146 -0.2664518
pour comprendre d'où viennent les valeurs de décision, nous pouvons les calculer manuellement comme le produit de points des poids de caractéristique et des vecteurs de caractéristique pré-traités, moins le décalage d'interception rho. (On entend par "prétraitement" la possibilité d'une transformation centrée/à l'échelle et/ou du noyau en cas D'utilisation de RBF SVM., etc.)
> t(w %*% t(as.matrix(iris.scaled))) - fit$rho
[,1]
51 -1.3997066
52 -0.4402254
53 -1.1596819
54 1.7199970
55 -0.2796942
56 0.9996141
...
Ce doit être égal à ce qui est calculé en interne:
> head(fit$decision.values)
versicolor/virginica
51 -1.3997066
52 -0.4402254
53 -1.1596819
54 1.7199970
55 -0.2796942
56 0.9996141
...