comment tracer et annoter les dendrogrammes de regroupement hiérarchique dans scipy / matplotlib
j'utilise dendrogram de scipy pour tracer le groupement hiérarchique en utilisant matplotlib comme suit:
mat = array([[1, 0.5, 0.9],
[0.5, 1, -0.5],
[0.9, -0.5, 1]])
plt.subplot(1,2,1)
plt.title("mat")
dist_mat = mat
linkage_matrix = linkage(dist_mat,
"single")
print "linkage2:"
print linkage(1-dist_mat, "single")
dendrogram(linkage_matrix,
color_threshold=1,
labels=["a", "b", "c"],
show_leaf_counts=True)
plt.subplot(1,2,2)
plt.title("1 - mat")
dist_mat = 1 - mat
linkage_matrix = linkage(dist_mat,
"single")
dendrogram(linkage_matrix,
color_threshold=1,
labels=["a", "b", "c"],
show_leaf_counts=True)
mes questions sont les suivantes: Premièrement, pourquoi mat et 1-mat donnent des regroupements identiques ici? et deuxièmement, Comment puis-je annoter la distance le long de chaque branche de l'arbre en utilisant dendrogram pour que les distances entre les paires de noeuds puissent être comparées?
enfin il semble que le drapeau show_leaf_counts est ignoré, est-il un moyen de l'allumer, de sorte que le nombre d'objets dans chaque catégorie est indiqué? grâce. 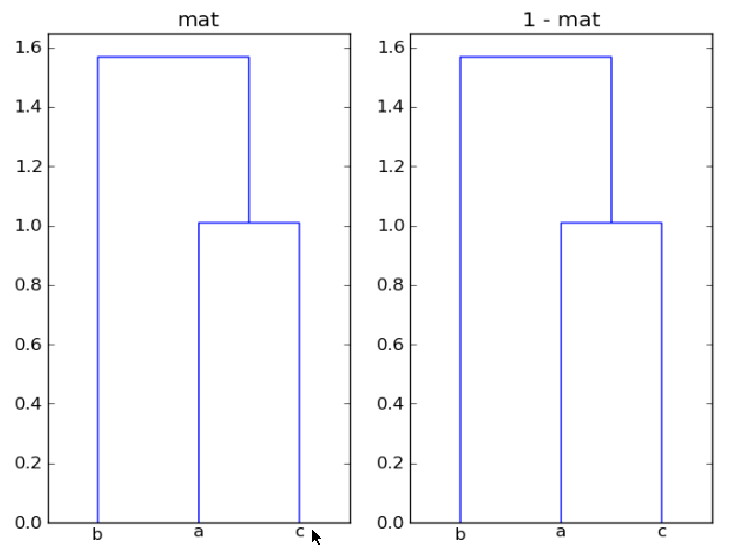
2 réponses
l'entrée à linkage() est soit un tableau n x m, représentant n points dans
m-dimensions de l'espace, ou un tableau à une dimension contenant les condensé matrice de distance . Dans votre exemple, mat est de 3 x 3, donc vous regroupez
3-d points. Le regroupement est basé sur la distance entre ces points.
pourquoi le tapis et le 1-mat donnent des regroupements identiques ici?
les tableaux mat et 1-mat produisent le même clustering parce que le clustering
est basée sur les distances entre les points, et ni une réflexion ( -mat )
pas plus qu'une traduction ( mat + offset ) de l'ensemble des données ne change le relatif
les distances entre les points.
Comment puis-je annoter la distance le long de chaque branche de l'arbre à l'aide de dendrogramme afin que les distances entre les paires de noeuds puissent être comparées?
dans le code ci-dessous, I
montrer comment vous pouvez utiliser les données retournées par dendrogramme pour étiqueter l'horizontale
segments du diagramme avec la distance correspondante. Les valeurs associées
avec les touches icoord et dcoord donner les coordonnées x et y de chaque
trois segments inversés-U de la figure. Dans augmented_dendrogram cette donnée
est utilisé pour ajouter une étiquette de la distance (c.-à-d. la valeur y) de chaque horizontale
segment de ligne à dendrogramme.
from scipy.cluster.hierarchy import dendrogram
import matplotlib.pyplot as plt
def augmented_dendrogram(*args, **kwargs):
ddata = dendrogram(*args, **kwargs)
if not kwargs.get('no_plot', False):
for i, d in zip(ddata['icoord'], ddata['dcoord']):
x = 0.5 * sum(i[1:3])
y = d[1]
plt.plot(x, y, 'ro')
plt.annotate("%.3g" % y, (x, y), xytext=(0, -8),
textcoords='offset points',
va='top', ha='center')
return ddata
pour votre tableau mat , le dendrogramme augmenté est
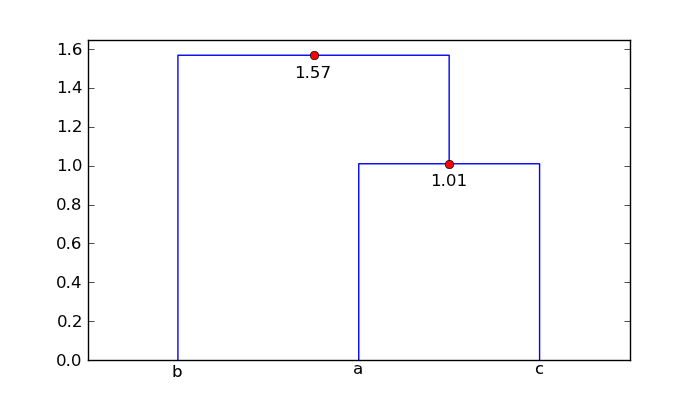
ainsi, les points " a " et " c "sont à 1,01 unité d'écart et le point" b " est à 1,57 unité d'écart. le cluster ['a','c'].
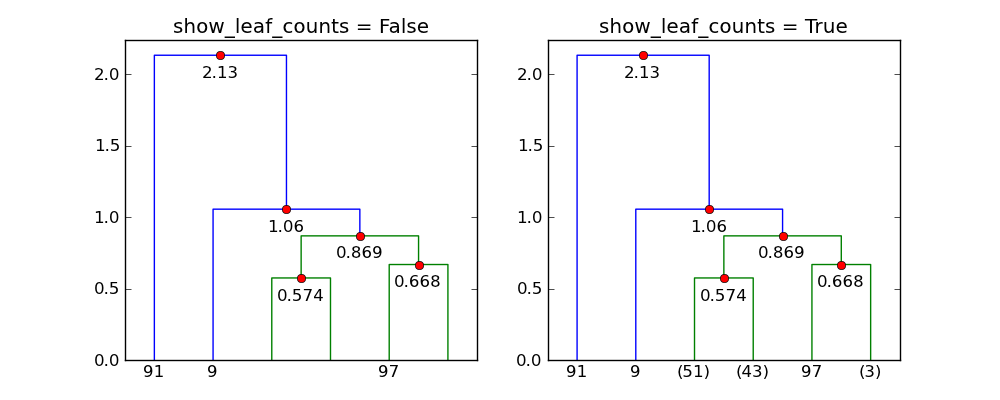
Il semble que show_leaf_counts est ignoré, il est un moyen pour l'activer
de sorte que le nombre d'objets dans chaque catégorie est indiqué?
le drapeau show_leaf_counts s'applique seulement lorsque toutes les données originales
les points sont représentés sous forme de feuilles. Par exemple, lorsque trunc_mode = "lastp" ,
seuls les derniers noeuds p sont affichés.
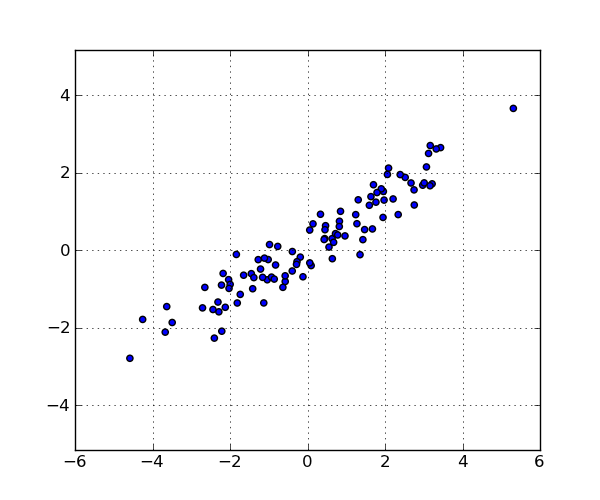
voici un exemple avec 100 points:
import numpy as np
from scipy.cluster.hierarchy import linkage
import matplotlib.pyplot as plt
from augmented_dendrogram import augmented_dendrogram
# Generate a random sample of `n` points in 2-d.
np.random.seed(12312)
n = 100
x = np.random.multivariate_normal([0, 0], np.array([[4.0, 2.5], [2.5, 1.4]]),
size=(n,))
plt.figure(1, figsize=(6, 5))
plt.clf()
plt.scatter(x[:, 0], x[:, 1])
plt.axis('equal')
plt.grid(True)
linkage_matrix = linkage(x, "single")
plt.figure(2, figsize=(10, 4))
plt.clf()
plt.subplot(1, 2, 1)
show_leaf_counts = False
ddata = augmented_dendrogram(linkage_matrix,
color_threshold=1,
p=6,
truncate_mode='lastp',
show_leaf_counts=show_leaf_counts,
)
plt.title("show_leaf_counts = %s" % show_leaf_counts)
plt.subplot(1, 2, 2)
show_leaf_counts = True
ddata = augmented_dendrogram(linkage_matrix,
color_threshold=1,
p=6,
truncate_mode='lastp',
show_leaf_counts=show_leaf_counts,
)
plt.title("show_leaf_counts = %s" % show_leaf_counts)
plt.show()
ce sont les points dans l'ensemble de données:
avec p=6 et trunc_mode="lastp" , dendrogram montre seulement le " haut"
du dendrogramme. Ce qui suit montre l'effet de show_leaf_counts .
je pense qu'il y a quelques malentendus quant à l'utilisation de la fonction que vous essayez d'utiliser. Voici un extrait de code entièrement fonctionnel pour illustrer mes points:
import matplotlib.pyplot as plt
from scipy.cluster.hierarchy import dendrogram, linkage
from numpy import array
import numpy as np
mat = array([184, 222, 177, 216, 231,
45, 123, 128, 200,
129, 121, 203,
46, 83,
83])
dist_mat = mat
linkage_matrix = linkage(dist_mat, 'single')
print linkage_matrix
plt.figure(101)
plt.subplot(1, 2, 1)
plt.title("ascending")
dendrogram(linkage_matrix,
color_threshold=1,
truncate_mode='lastp',
labels=array(['a', 'b', 'c', 'd', 'e', 'f']),
distance_sort='ascending')
plt.subplot(1, 2, 2)
plt.title("descending")
dendrogram(linkage_matrix,
color_threshold=1,
truncate_mode='lastp',
labels=array(['a', 'b', 'c', 'd', 'e', 'f']),
distance_sort='descending')
def make_fake_data():
amp = 1000.
x = []
y = []
for i in range(0, 10):
s = 20
x.append(np.random.normal(30, s))
y.append(np.random.normal(30, s))
for i in range(0, 20):
s = 2
x.append(np.random.normal(150, s))
y.append(np.random.normal(150, s))
for i in range(0, 10):
s = 5
x.append(np.random.normal(-20, s))
y.append(np.random.normal(50, s))
plt.figure(1)
plt.title('fake data')
plt.scatter(x, y)
d = []
for i in range(len(x) - 1):
for j in range(i+1, len(x) - 1):
d.append(np.sqrt(((x[i]-x[j])**2 + (y[i]-y[j])**2)))
return d
mat = make_fake_data()
plt.figure(102)
plt.title("Three Clusters")
linkage_matrix = linkage(mat, 'single')
print "three clusters"
print linkage_matrix
dendrogram(linkage_matrix,
truncate_mode='lastp',
color_threshold=1,
show_leaf_counts=True)
plt.show()
tout d'abord, le calcul m -> m - 1 n'a pas vraiment changé votre résultat puisque la matrice de distance, qui décrit essentiellement les distances relatives entre toutes les paires uniques, n'a pas changé dans votre cas spécifique. (Dans mon exemple de code ci-dessus, toutes les distances sont euclidiennes donc toutes sont positifs et cohérents à partir de points sur un plan 2d.)
pour votre deuxième question, vous avez probablement besoin de déployer votre propre routine d'annotation pour faire ce que vous voulez, car je ne pense pas que le dendromgramme le supporte nativement...
pour la dernière question, show_leaf_counts ne semble fonctionner que lorsque vous essayez d'Afficher des noeuds de feuilles non-singleton avec l'option truncate_mode='lastp'. En gros une les feuilles sont repliées de tellement proches qu'ils ne sont pas faciles à voir. Donc, vous avez une option d'affichage d'une feuille, mais ont une option de montrer (entre parenthèses) combien sont bottes dans cette feuille.
Espérons que cette aide.