Un exemple d'utilisation de fixations python pour la bibliothèque SVM, LIBSVM
j'ai grand besoin d'un exemple de tâche de classification utilisant LibSVM en python. Je ne sais pas à quoi devrait ressembler la saisie et quelle fonction est responsable de la formation et quelle fonction doit être testée. Merci
8 réponses
LIBSVM lit les données d'un tuple contenant deux listes. La première liste contient les classes et la deuxième liste contient les données d'entrée. créer un ensemble de données simple avec deux classes possibles vous devez également spécifier le noyau que vous souhaitez utiliser en créant svm_parameter.
>> from libsvm import *
>> prob = svm_problem([1,-1],[[1,0,1],[-1,0,-1]])
>> param = svm_parameter(kernel_type = LINEAR, C = 10)
## training the model
>> m = svm_model(prob, param)
#testing the model
>> m.predict([1, 1, 1])
les exemples de code énumérés ici ne fonctionnent pas avec LibSVM 3.1, donc j'ai plus ou moins porté l'exemple de mossplix :
from svmutil import *
svm_model.predict = lambda self, x: svm_predict([0], [x], self)[0][0]
prob = svm_problem([1,-1], [[1,0,1], [-1,0,-1]])
param = svm_parameter()
param.kernel_type = LINEAR
param.C = 10
m=svm_train(prob, param)
m.predict([1,1,1])
cet exemple montre une classe unique SVM classifier ; il est à peu près aussi simple que possible tout en montrant le flux de travail LIBSVM complet.
Step 1 : Import NumPy & LIBSVM
import numpy as NP
from svm import *
Étape 2: générer des données synthétiques: pour cet exemple, 500 points dans un limite (note: Pas mal de real les ensembles de données sont fournis sur le LIBSVM site web )
Data = NP.random.randint(-5, 5, 1000).reshape(500, 2)
Étape 3: maintenant, choisissez une limite de décision non linéaire pour une classe unique Classificateur:
rx = [ (x**2 + y**2) < 9 and 1 or 0 for (x, y) in Data ]
Étape 4: ensuite, séparez arbitrairement les données w / R / T ceci limite de la décision:
-
Classe I : ceux qui se trouvent sur ou dans un cercle arbitraire
-
Classe II : tous les points à l'extérieur de la limite de décision (cercle)
la construction du modèle SVM commence ici; toutes les étapes avant celle-ci étaient juste pour préparer quelques données synthétiques.
Étape 5 : construire la Description du problème en appelant svm_problem , en passant dans la fonction de limite de décision et la data , puis lier ce résultat à une variable.
px = svm_problem(rx, Data)
Étape 6: sélectionner une fonction du noyau pour la cartographie non linéaire
pour cet exemple, j'ai choisi RBF (fonction de base radiale) comme fonction de mon noyau
pm = svm_parameter(kernel_type=RBF)
Étape 7: Train le classificateur, en appelant svm_model , en passant dans le description du problème (px) & noyau (h)
v = svm_model(px, pm)
Étape 8: enfin, tester le classificateur formé en appelant prédire sur l'objet modèle formé ('v')
v.predict([3, 1])
# returns the class label (either '1' or '0')
pour l'exemple ci-dessus, j'ai utilisé la version 3.0 de LIBSVM (la version stable actuelle à l'époque cette réponse a été affichée).
enfin, w/r/t la partie de votre question concernant le choix de fonction du noyau , les machines vectorielles de Support sont et non spécifiques à un noyau particulier fonction -- par exemple, j'aurais pu choisir un noyau différent (gaussien, polynomial, etc.).
LIBSVM inclut toutes les fonctions du noyau les plus couramment utilisées-ce qui est d'une grande aide parce que vous pouvez voir toutes les alternatives plausibles et d'en choisir une pour utilisation dans votre modèle, est juste une question d'appeler svm_parameter et passer dans une valeur pour kernel_type (une abréviation de trois lettres pour le noyau choisi).
enfin, la fonction du noyau que vous choisissez pour la formation doit correspondre à la fonction du noyau utilisée contre les données de test.
vous pourriez envisager d'utiliser
http://scikit-learn.sourceforge.net /
qui a une grande liaison python de libsvm et devrait être facile à installer""
ajoute à @shinNoNoir:
param.kernel_type représente le type de fonction du noyau que vous voulez utiliser, 0: linéaire 1: polynôme 2: RBF 3: Sigmoid
ont également à l'esprit que, svm_problem(y,x) : ici y est les étiquettes de classe et x est les instances de classe et x et y ne peuvent être que des listes,des tuples et des dictionnaires.(pas de tableau numpy)
SVM via SciKit-learn:
from sklearn.svm import SVC
X = [[0, 0], [1, 1]]
y = [0, 1]
model = SVC().fit(X, y)
tests = [[0.,0.], [0.49,0.49], [0.5,0.5], [2., 2.]]
print(model.predict(tests))
# prints [0 0 1 1]
pour plus de détails ici: http://scikit-learn.org/stable/modules/svm.html#svm
param = svm_parameter('-s 0 -t 2 -d 3 -c '+str(C)+' -g '+str(G)+' -p '+str(self.epsilon)+' -n '+str(self.nu))
je ne sais pas pour les versions antérieures, mais dans LibSVM 3.xx la méthode svm_parameter('options') prend juste un argument .
Dans mon cas C , G , p et nu sont les valeurs dynamiques. Vous faites des changements selon votre code.
options:
-s svm_type : set type of SVM (default 0)
0 -- C-SVC (multi-class classification)
1 -- nu-SVC (multi-class classification)
2 -- one-class SVM
3 -- epsilon-SVR (regression)
4 -- nu-SVR (regression)
-t kernel_type : set type of kernel function (default 2)
0 -- linear: u'*v
1 -- polynomial: (gamma*u'*v + coef0)^degree
2 -- radial basis function: exp(-gamma*|u-v|^2)
3 -- sigmoid: tanh(gamma*u'*v + coef0)
4 -- precomputed kernel (kernel values in training_set_file)
-d degree : set degree in kernel function (default 3)
-g gamma : set gamma in kernel function (default 1/num_features)
-r coef0 : set coef0 in kernel function (default 0)
-c cost : set the parameter C of C-SVC, epsilon-SVR, and nu-SVR (default 1)
-n nu : set the parameter nu of nu-SVC, one-class SVM, and nu-SVR (default 0.5)
-p epsilon : set the epsilon in loss function of epsilon-SVR (default 0.1)
-m cachesize : set cache memory size in MB (default 100)
-e epsilon : set tolerance of termination criterion (default 0.001)
-h shrinking : whether to use the shrinking heuristics, 0 or 1 (default 1)
-b probability_estimates : whether to train a SVC or SVR model for probability estimates, 0 or 1 (default 0)
-wi weight : set the parameter C of class i to weight*C, for C-SVC (default 1)
-v n: n-fold cross validation mode
-q : quiet mode (no outputs)
Source de la documentation: https://www.csie.ntu.edu.tw / ~ cjlin/libsvm /
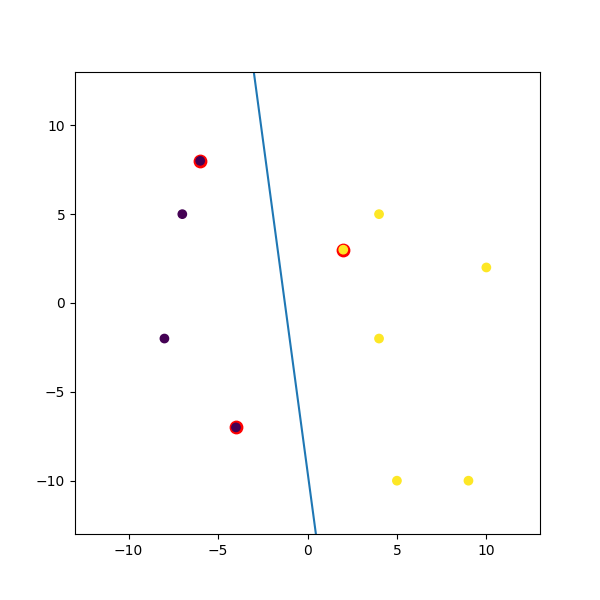
voici un exemple factice que j'ai masqué:
import numpy
import matplotlib.pyplot as plt
from random import seed
from random import randrange
import svmutil as svm
seed(1)
# Creating Data (Dense)
train = list([randrange(-10, 11), randrange(-10, 11)] for i in range(10))
labels = [-1, -1, -1, 1, 1, -1, 1, 1, 1, 1]
options = '-t 0' # linear model
# Training Model
model = svm.svm_train(labels, train, options)
# Line Parameters
w = numpy.matmul(numpy.array(train)[numpy.array(model.get_sv_indices()) - 1].T, model.get_sv_coef())
b = -model.rho.contents.value
if model.get_labels()[1] == -1: # No idea here but it should be done :|
w = -w
b = -b
print(w)
print(b)
# Plotting
plt.figure(figsize=(6, 6))
for i in model.get_sv_indices():
plt.scatter(train[i - 1][0], train[i - 1][1], color='red', s=80)
train = numpy.array(train).T
plt.scatter(train[0], train[1], c=labels)
plt.plot([-5, 5], [-(-5 * w[0] + b) / w[1], -(5 * w[0] + b) / w[1]])
plt.xlim([-13, 13])
plt.ylim([-13, 13])
plt.show()