Pourquoi mon modèle CIFAR 100 CNN prédit-il principalement deux classes?
j'essaie actuellement d'obtenir un score décent (précision> 40%) avec Keras sur CIFAR 100. Cependant, je suis confronté à un comportement étrange d'un modèle CNN: il tend à prédire certaines classes (2 - 5) beaucoup plus souvent que d'autres:
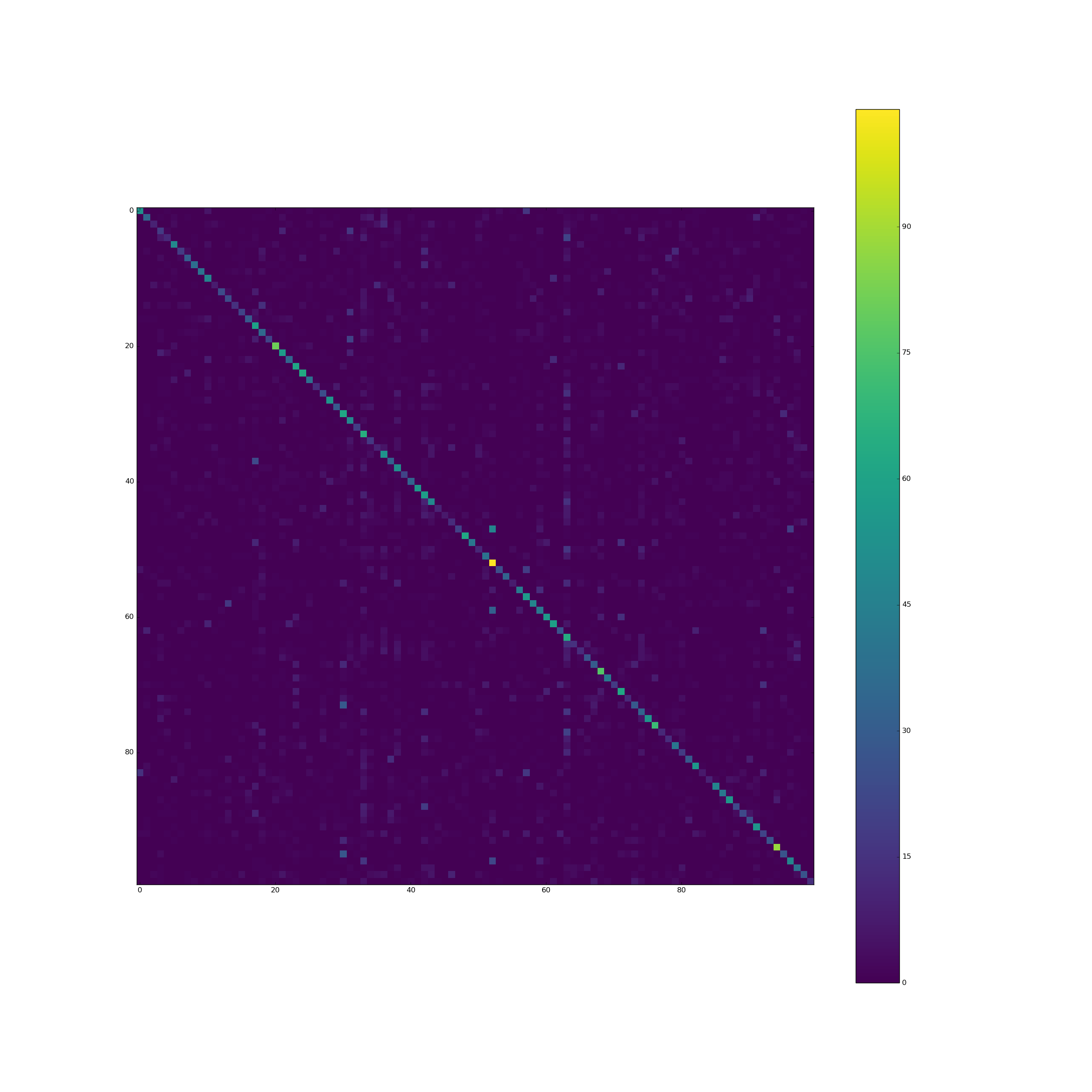
le pixel à la position (i, j) contient le nombre d'éléments du jeu de validation de la classe i prédits comme étant de la classe J. Ainsi la diagonale contient les classifications correctes, tout le reste est une erreur. Les deux barres verticales indiquent que le modèle prédit souvent ces classes, bien que ce ne soit pas le cas.
CIFAR 100 est parfaitement équilibré: toutes les 100 classes ont 500 échantillons de formation.
pourquoi le modèle a-t-il tendance à prédire certaines classes beaucoup plus souvent que d'autres? Comment cela peut-il être fixé?
le code
en cours d'Exécution, cela prend une alors.
#!/usr/bin/env python
from __future__ import print_function
from keras.datasets import cifar100
from keras.preprocessing.image import ImageDataGenerator
from keras.models import Sequential
from keras.layers import Dense, Dropout, Activation, Flatten
from keras.layers import Convolution2D, MaxPooling2D
from keras.utils import np_utils
from sklearn.model_selection import train_test_split
import numpy as np
batch_size = 32
nb_classes = 100
nb_epoch = 50
data_augmentation = True
# input image dimensions
img_rows, img_cols = 32, 32
# The CIFAR10 images are RGB.
img_channels = 3
# The data, shuffled and split between train and test sets:
(X, y), (X_test, y_test) = cifar100.load_data()
X_train, X_val, y_train, y_val = train_test_split(X, y,
test_size=0.20,
random_state=42)
# Shuffle training data
perm = np.arange(len(X_train))
np.random.shuffle(perm)
X_train = X_train[perm]
y_train = y_train[perm]
print('X_train shape:', X_train.shape)
print(X_train.shape[0], 'train samples')
print(X_val.shape[0], 'validation samples')
print(X_test.shape[0], 'test samples')
# Convert class vectors to binary class matrices.
Y_train = np_utils.to_categorical(y_train, nb_classes)
Y_test = np_utils.to_categorical(y_test, nb_classes)
Y_val = np_utils.to_categorical(y_val, nb_classes)
model = Sequential()
model.add(Convolution2D(32, 3, 3, border_mode='same',
input_shape=X_train.shape[1:]))
model.add(Activation('relu'))
model.add(Convolution2D(32, 3, 3))
model.add(Activation('relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Convolution2D(64, 3, 3, border_mode='same'))
model.add(Activation('relu'))
model.add(Convolution2D(64, 3, 3))
model.add(Activation('relu'))
model.add(MaxPooling2D(pool_size=(2, 2)))
model.add(Dropout(0.25))
model.add(Flatten())
model.add(Dense(1024))
model.add(Activation('tanh'))
model.add(Dropout(0.5))
model.add(Dense(nb_classes))
model.add(Activation('softmax'))
model.compile(loss='categorical_crossentropy',
optimizer='adam',
metrics=['accuracy'])
X_train = X_train.astype('float32')
X_val = X_val.astype('float32')
X_test = X_test.astype('float32')
X_train /= 255
X_val /= 255
X_test /= 255
if not data_augmentation:
print('Not using data augmentation.')
model.fit(X_train, Y_train,
batch_size=batch_size,
nb_epoch=nb_epoch,
validation_data=(X_val, y_val),
shuffle=True)
else:
print('Using real-time data augmentation.')
# This will do preprocessing and realtime data augmentation:
datagen = ImageDataGenerator(
featurewise_center=False, # set input mean to 0 over the dataset
samplewise_center=False, # set each sample mean to 0
featurewise_std_normalization=False, # divide inputs by std of the dataset
samplewise_std_normalization=False, # divide each input by its std
zca_whitening=False, # apply ZCA whitening
rotation_range=0, # randomly rotate images in the range (degrees, 0 to 180)
width_shift_range=0.1, # randomly shift images horizontally (fraction of total width)
height_shift_range=0.1, # randomly shift images vertically (fraction of total height)
horizontal_flip=True, # randomly flip images
vertical_flip=False) # randomly flip images
# Compute quantities required for featurewise normalization
# (std, mean, and principal components if ZCA whitening is applied).
datagen.fit(X_train)
# Fit the model on the batches generated by datagen.flow().
model.fit_generator(datagen.flow(X_train, Y_train,
batch_size=batch_size),
samples_per_epoch=X_train.shape[0],
nb_epoch=nb_epoch,
validation_data=(X_val, Y_val))
model.save('cifar100.h5')
code de visualisation
#!/usr/bin/env python
"""Analyze a cifar100 keras model."""
from keras.models import load_model
from keras.datasets import cifar100
from sklearn.model_selection import train_test_split
import numpy as np
import json
import io
import matplotlib.pyplot as plt
try:
to_unicode = unicode
except NameError:
to_unicode = str
n_classes = 100
def plot_cm(cm, zero_diagonal=False):
"""Plot a confusion matrix."""
n = len(cm)
size = int(n / 4.)
fig = plt.figure(figsize=(size, size), dpi=80, )
plt.clf()
ax = fig.add_subplot(111)
ax.set_aspect(1)
res = ax.imshow(np.array(cm), cmap=plt.cm.viridis,
interpolation='nearest')
width, height = cm.shape
fig.colorbar(res)
plt.savefig('confusion_matrix.png', format='png')
# Load model
model = load_model('cifar100.h5')
# Load validation data
(X, y), (X_test, y_test) = cifar100.load_data()
X_train, X_val, y_train, y_val = train_test_split(X, y,
test_size=0.20,
random_state=42)
# Calculate confusion matrix
y_val_i = y_val.flatten()
y_val_pred = model.predict(X_val)
y_val_pred_i = y_val_pred.argmax(1)
cm = np.zeros((n_classes, n_classes), dtype=np.int)
for i, j in zip(y_val_i, y_val_pred_i):
cm[i][j] += 1
acc = sum([cm[i][i] for i in range(100)]) / float(cm.sum())
print("Validation accuracy: %0.4f" % acc)
# Create plot
plot_cm(cm)
# Serialize confusion matrix
with io.open('cm.json', 'w', encoding='utf8') as outfile:
str_ = json.dumps(cm.tolist(),
indent=4, sort_keys=True,
separators=(',', ':'), ensure_ascii=False)
outfile.write(to_unicode(str_))
harengs rouges
tanh
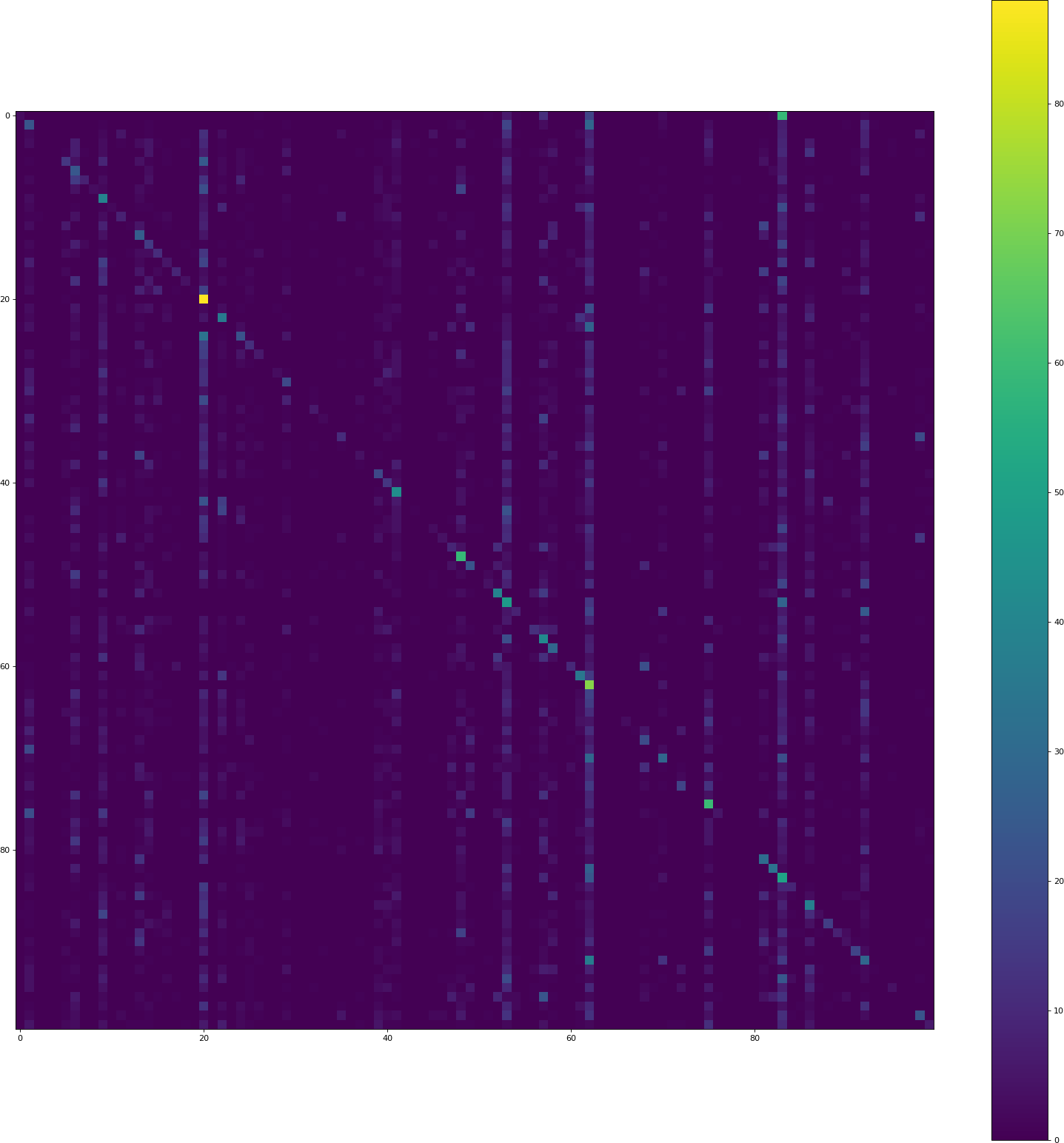
j'ai remplacé tanh par relu . Le histoire csv semble ok, mais la visualisation a le même problème:
veuillez noter que la précision de validation ici est seulement 3.44%.
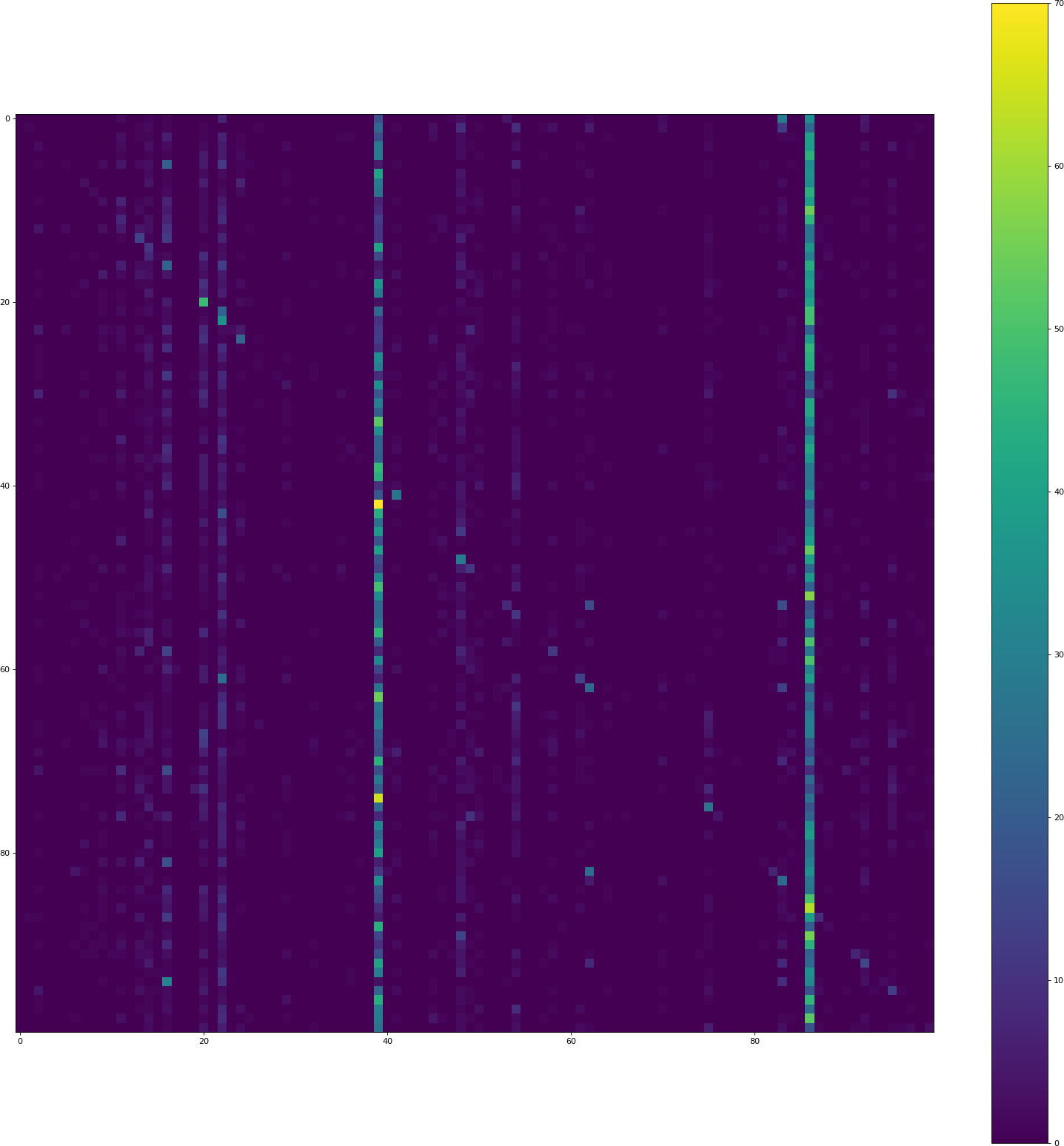
Décrochage + tanh + bordure en mode
suppression de l'abandon, remplacement de tanh par relu, mise en mode border partout: historique csv
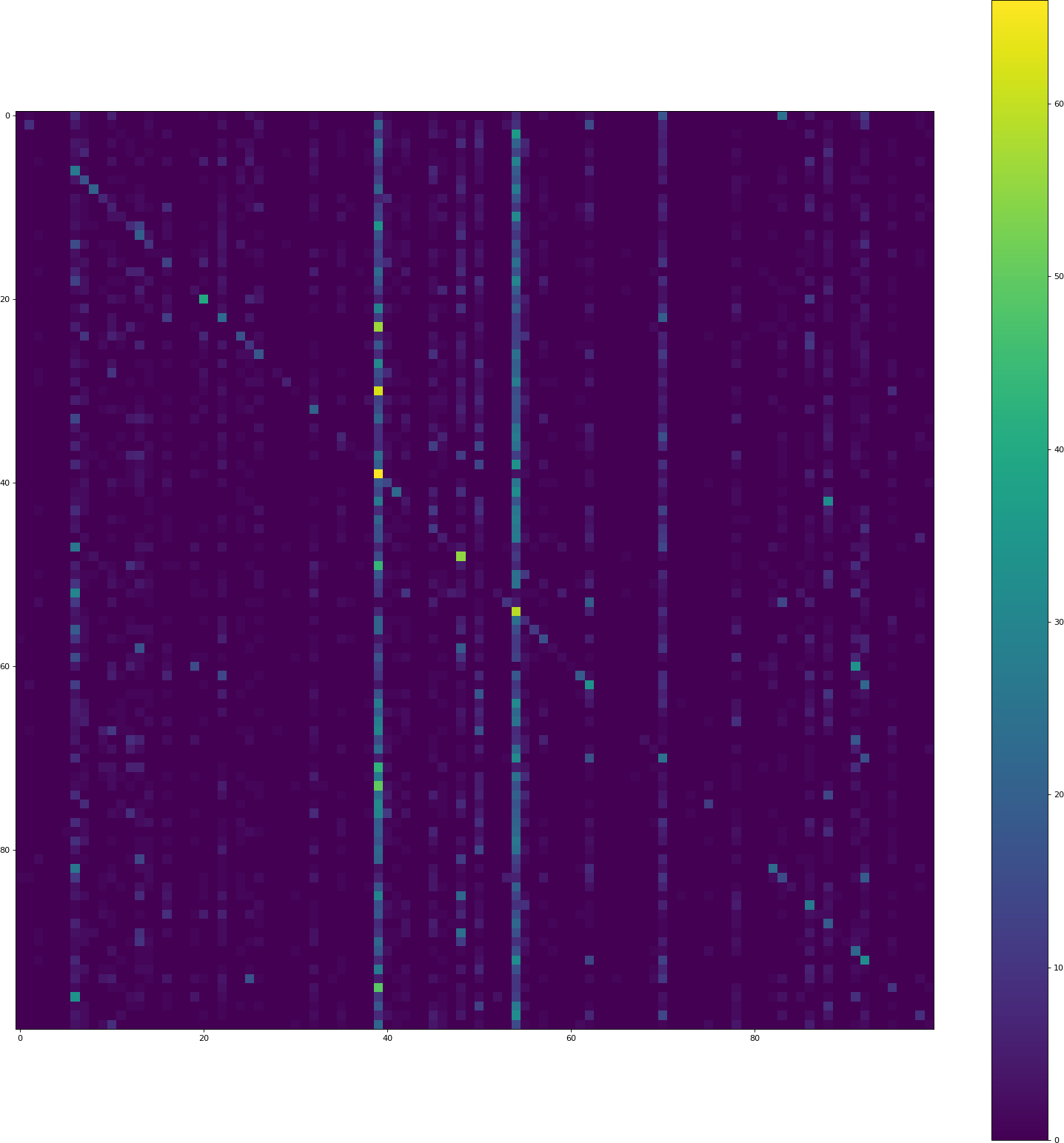
le code de visualisation donne encore une précision beaucoup plus faible (8.50% cette fois) que le code de formation de keras.
Q &A
Voici un résumé des commentaires:
- les données sont également réparties entre les classes. Il n'y a donc pas de "surentraînement" de ces deux classes.
- l'augmentation de données est utilisée, mais sans l'augmentation de données le problème persiste.
- La visualisation n'est pas le problème.
4 réponses
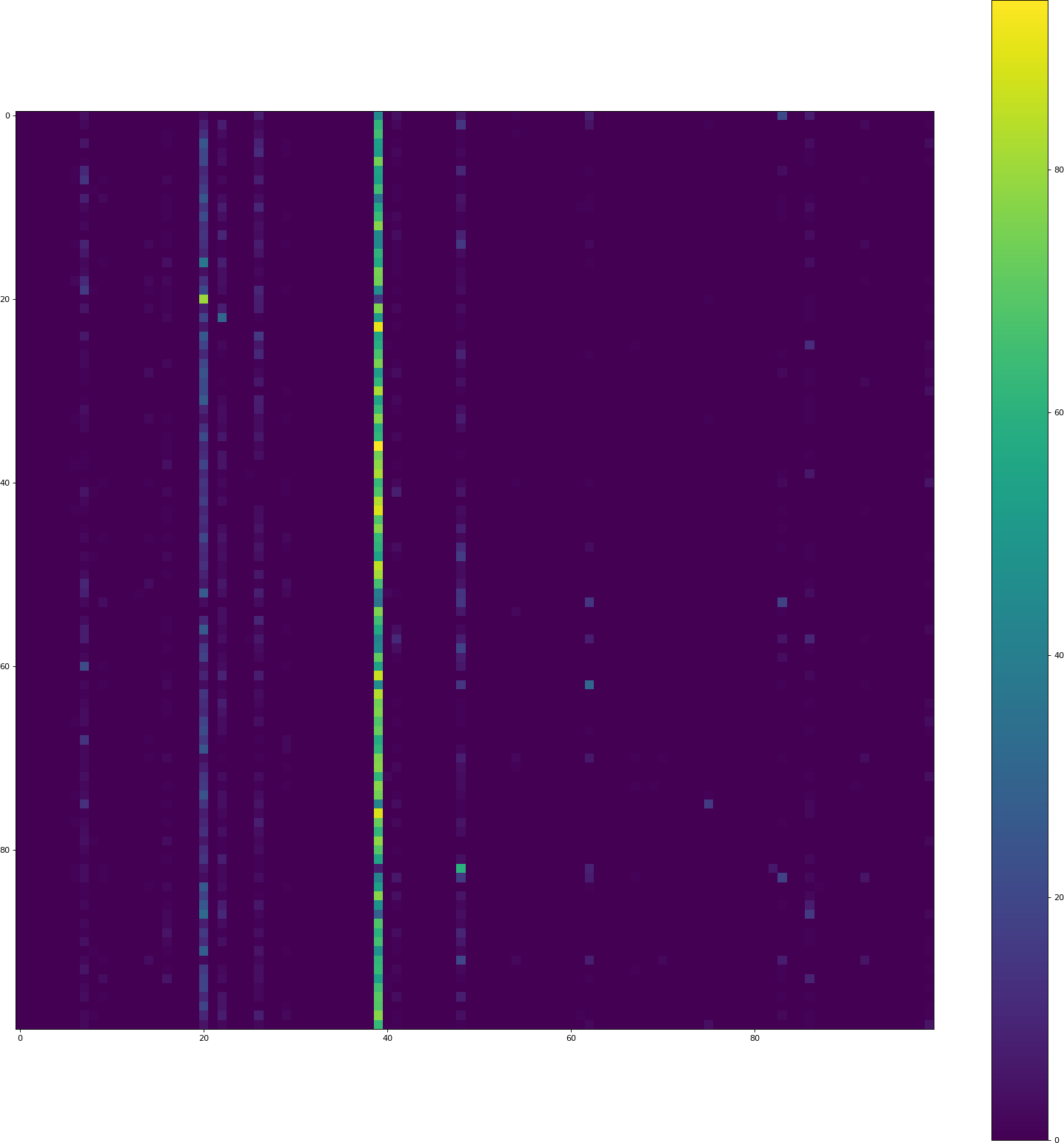
si vous obtenez une bonne précision lors de la formation et de la validation, mais pas lors des tests, assurez-vous de faire exactement le même prétraitement sur votre ensemble de données dans les deux cas. Ici vous avez lors de la formation:
X_train /= 255
X_val /= 255
X_test /= 255
mais pas de tel code pour prédire votre matrice de confusion. Ajout au test:
X_val /= 255.
donne la matrice de confusion d'apparence agréable suivante:
je n'ai pas un bon feeling avec cette partie du code:
model.add(Dense(1024))
model.add(Activation('tanh'))
model.add(Dropout(0.5))
model.add(Dense(nb_classes))
model.add(Activation('softmax'))
le modèle restant est plein de relus , mais ici il y a un tanh .
tanh parfois disparaît ou explose (satureates à -1 et 1), ce qui pourrait conduire à votre overimportance de classe 2.
keras-exemple cifar 10 utilise essentiellement la même architecture( les tailles de la couche dense peuvent être différentes), mais utilise également un relu là (pas de tanh du tout). Il en va de même pour ce code cifar 100 externe basé sur keras .
Une partie importante du problème était que mon ~/.keras/keras.json était
{
"image_dim_ordering": "th",
"epsilon": 1e-07,
"floatx": "float32",
"backend": "tensorflow"
}
J'ai donc dû remplacer image_dim_ordering par tf . Cela conduit à
et une précision de 12.73%. De toute évidence, il y a toujours un problème puisque le "historique de validation a donné une précision de 45,1%.
-
Je ne vous vois pas faire du mean-centering, même en datagen. Je suppose que c'est la cause principale. Pour faire signifie centrage utilisant
ImageDataGenerator, mettrefeaturewise_center = 1. Une autre façon est de soustraire la moyenne D'ImageNet de chaque pixel RVB. Le vecteur moyen à soustraire est[103.939, 116.779, 123.68]. -
faites toutes les activations
relus, sauf si vous avez une raison spécifique d'avoir un seultanh. -
Supprimer deux abandons de 0.25 et voir ce qui se passe. Si vous voulez appliquer des dropouts à la couche de convolution, il est préférable d'utiliser
SpatialDropout2D. Il est en quelque sorte retiré de la documentation en ligne de Keras mais vous pouvez le trouver dans le source . -
vous avez deux couches
convavecsameet deux avecvalid. Il n'y a rien de mal à cela, mais il serait plus simple de garder tousconvcouches avecsameet contrôler votre taille juste basée sur max-poolings.