Quelle disposition dois-je utiliser pour obtenir des bords ne se chevauchant pas dans igraph?
j'essaie de construire des graphiques en utilisant des données arborescentes, où les noeuds se divisent typiquement en >2 arêtes. J'ai essayé différentes mises en page, et je vois que la mise en page.reingold.le paramètre tilford générera des graphiques arborescents avec des données non bifurquées. Toutefois, les résultats ne sont pas particulièrement attrayants. Je préférerais utiliser quelque chose comme la disposition.lgl ou de mise en page.kamada.kawai puisque ceux-ci produisent des structures plus radiales. Je ne vois pas comment changer les paramètres en R de telle sorte que ces arbres n'ont pas mais les bords se chevauchent. Est-ce possible?
j'ai importé un simple fichier de données au format Pajek, avec 355 noeuds et 354 arêtes. Je l'imprime actuellement en utilisant:
plot.igraph(g,vertex.size=3,vertex.label=NA,layout=layout.lgl)
cela me donne une sortie comme celle-ci, qui est agréable, mais a toujours des bords qui se chevauchent. J'ai lu que vous pouvez corriger cela manuellement en utilisant tkplot, ou un autre programme comme cytoscape, cependant j'ai pas mal de ceux-ci à construire, et la taille d'entre eux rend la correction manuelle un tracas.
Beaucoup grâce.

3 réponses
layout.reingold.tilford a un paramètre appelé circular. Mettre ce paramètre à TRUE convertira le plan final en un plan radial en traitant la coordonnée X comme l'angle (après ré-échelle appropriée) et la coordonnée Y comme le rayon. Ironiquement, cela ne garantit pas qu'il n'y aura pas de traversées de bord à la fin, mais cela fonctionne bien si vos sous-arbres ne sont pas excessivement larges par rapport au reste du graphique:
> g <- barabasi.game(100, directed=F)
> layout <- layout.reingold.tilford(g, circular=T)
> plot(g, layout=layout)
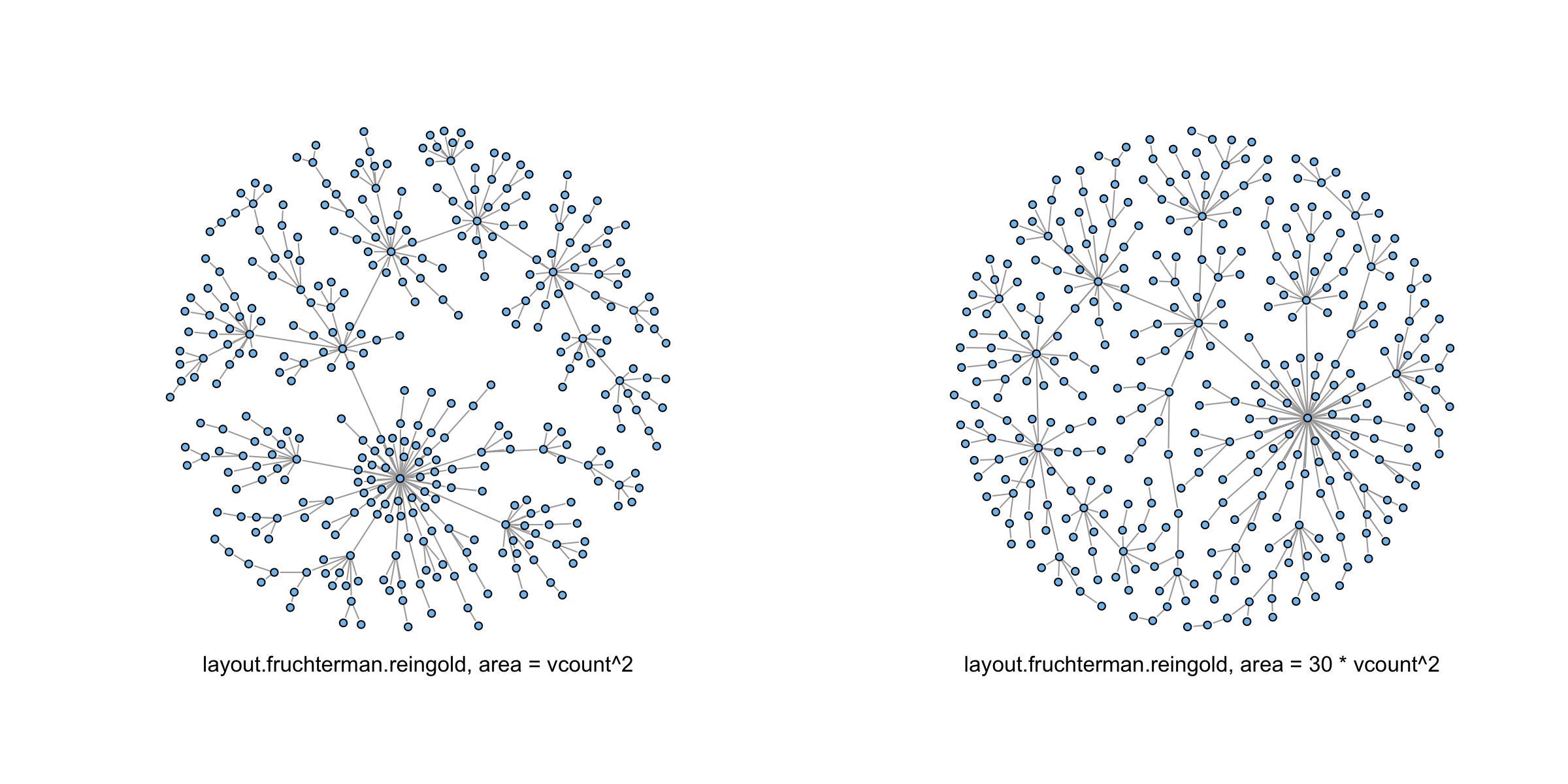
Vous pouvez essayer layout.fruchterman.reingold(). Il semble faire un bon travail pour empêcher les bords de traverser. Je l'ai testé avec une version à 355 noeuds du graphe barabasi suggéré par @Tamás.
library(igraph)
g = barabasi.game(355, directed=FALSE)
png("plot1.png", height=6, width=12, units="in", res=200)
par(mfrow=c(1, 2))
plot.igraph(g,vertex.size=3,vertex.label=NA,
layout=layout.fruchterman.reingold(g, niter=10000))
mtext("layout.fruchterman.reingold, area = vcount^2", side=1)
plot.igraph(g,vertex.size=3,vertex.label=NA,
layout=layout.fruchterman.reingold(g, niter=10000, area=30*vcount(g)^2))
mtext("layout.fruchterman.reingold, area = 30 * vcount^2", side=1)
dev.off()
je veux juste ajouter un commentaire mais ma réputation est trop basse. La méthode que @bdemarest a posté ne fonctionne pas sur igraph version > 0.7. La nouvelle version ne supporte pas le area paramètre, donc je ne peux pas obtenir le même effet. Et obtenir l'ancienne version à construire m'a pris un certain temps, donc j'ai pensé que je partagerais quelques idées. Vous pouvez installer manuellement igraph 0.7 partir de la source si vous le télécharger à partir de igraph nightly builds. Sur ma machine (Mac OS 10.10), j'ai rencontré quelques problèmes de construction c', en raison de gfortran, donc j'ai trouvé ce lien qui a résolu le problème. Hope qui aide toute personne qui veut créer des graphiques similaires dans R.