Quelle est la différence entre pandas ACF et statsmodel ACF?
Je calcule la fonction D'autocorrélation pour les retours d'actions. Pour ce faire, j'ai testé deux fonctions, l' autocorr fonction intégrée dans Pandas, et le acf fonction fournie par statsmodels.tsa. Ceci est fait dans le suivant MWE:
import pandas as pd
from pandas_datareader import data
import matplotlib.pyplot as plt
import datetime
from dateutil.relativedelta import relativedelta
from statsmodels.tsa.stattools import acf, pacf
ticker = 'AAPL'
time_ago = datetime.datetime.today().date() - relativedelta(months = 6)
ticker_data = data.get_data_yahoo(ticker, time_ago)['Adj Close'].pct_change().dropna()
ticker_data_len = len(ticker_data)
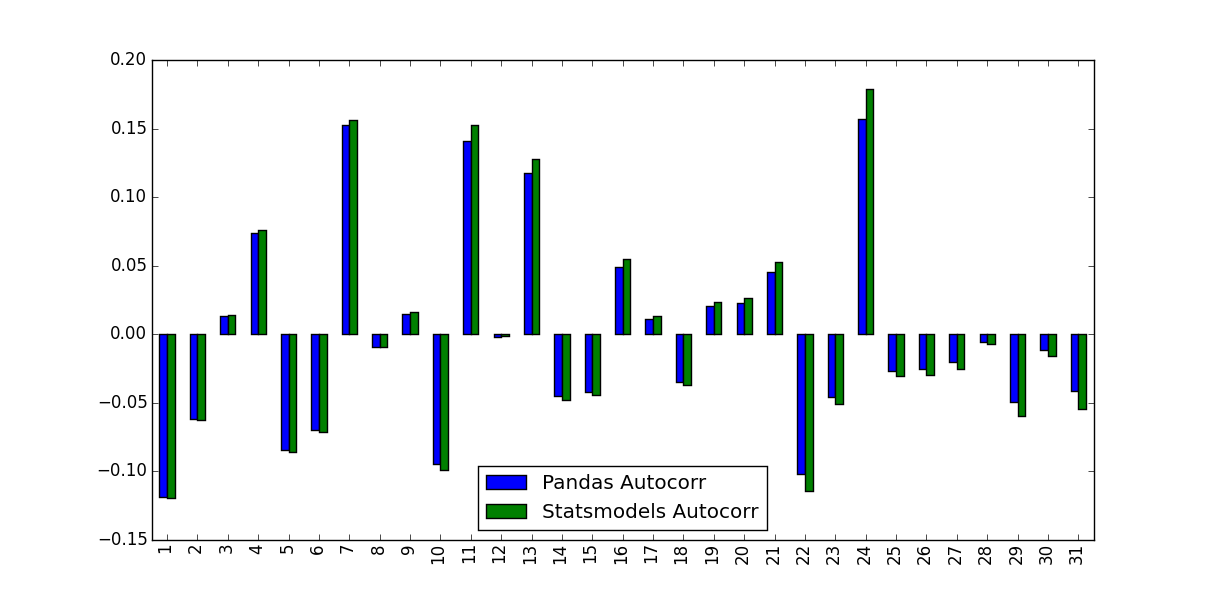
ticker_data_acf_1 = acf(ticker_data)[1:32]
ticker_data_acf_2 = [ticker_data.autocorr(i) for i in range(1,32)]
test_df = pd.DataFrame([ticker_data_acf_1, ticker_data_acf_2]).T
test_df.columns = ['Pandas Autocorr', 'Statsmodels Autocorr']
test_df.index += 1
test_df.plot(kind='bar')
ce que j'ai remarqué, c'est que les valeurs prédites n'étaient pas identiques:
quelles sont les raisons de cette différence et quelles sont les valeurs à utiliser?
2 réponses
la différence entre la version Pandas et la version Statsmodels réside dans la soustraction moyenne et la normalisation / Division de la variance:
autocorrne fait rien de plus que de passer des sous-séries de la série originale ànp.corrcoef. Dans cette méthode, la moyenne de l'échantillon et la variance de l'échantillon de ces sous-séries sont utilisées pour déterminer le coefficient de corrélationacf, au contraire, utilise la moyenne globale de l'échantillon de la série et la variance de l'échantillon pour déterminer la corrélation coefficient.
les différences peuvent devenir plus petites pour les séries chronologiques plus longues, mais sont assez grandes pour les séries plus courtes.
par rapport à Matlab, les Pandas autocorr la fonction correspond probablement à faire des Matlabs xcorr (cross-corr) avec la série (décalée) elle-même, au lieu deautocorr, qui calcule l'autocorrélation de l'échantillon (en devinant à partir du docs; Je ne peux pas valider cela parce que je n'ai pas accès à Matlab).
voir ce MWE pour clarification:
import numpy as np
import pandas as pd
from statsmodels.tsa.stattools import acf
import matplotlib.pyplot as plt
plt.style.use("seaborn-colorblind")
def autocorr_by_hand(x, lag):
# Slice the relevant subseries based on the lag
y1 = x[:(len(x)-lag)]
y2 = x[lag:]
# Subtract the subseries means
sum_product = np.sum((y1-np.mean(y1))*(y2-np.mean(y2)))
# Normalize with the subseries stds
return sum_product / ((len(x) - lag) * np.std(y1) * np.std(y2))
def acf_by_hand(x, lag):
# Slice the relevant subseries based on the lag
y1 = x[:(len(x)-lag)]
y2 = x[lag:]
# Subtract the mean of the whole series x to calculate Cov
sum_product = np.sum((y1-np.mean(x))*(y2-np.mean(x)))
# Normalize with var of whole series
return sum_product / ((len(x) - lag) * np.var(x))
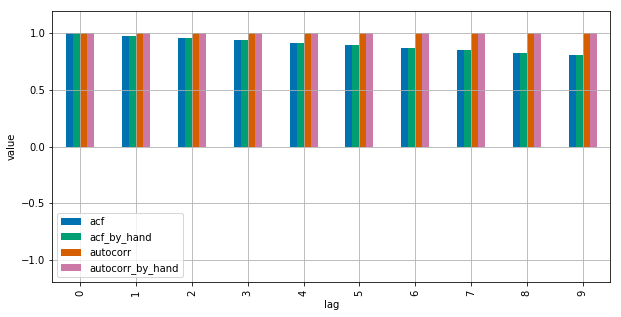
x = np.linspace(0,100,101)
results = {}
nlags=10
results["acf_by_hand"] = [acf_by_hand(x, lag) for lag in range(nlags)]
results["autocorr_by_hand"] = [autocorr_by_hand(x, lag) for lag in range(nlags)]
results["autocorr"] = [pd.Series(x).autocorr(lag) for lag in range(nlags)]
results["acf"] = acf(x, unbiased=True, nlags=nlags-1)
pd.DataFrame(results).plot(kind="bar", figsize=(10,5), grid=True)
plt.xlabel("lag")
plt.ylim([-1.2, 1.2])
plt.ylabel("value")
plt.show()
Statsmodels utilise np.correlate pour optimiser ceci, mais c'est essentiellement comme ça que ça marche.
Comme suggéré dans les commentaires, le problème peut être diminué, mais pas complètement résolu, en fournissant unbiased=Truestatsmodels fonction. À l'aide d'un aléatoire d'entrée:
import statistics
import numpy as np
import pandas as pd
from statsmodels.tsa.stattools import acf
DATA_LEN = 100
N_TESTS = 100
N_LAGS = 32
def test(unbiased):
data = pd.Series(np.random.random(DATA_LEN))
data_acf_1 = acf(data, unbiased=unbiased, nlags=N_LAGS)
data_acf_2 = [data.autocorr(i) for i in range(N_LAGS+1)]
# return difference between results
return sum(abs(data_acf_1 - data_acf_2))
for value in (False, True):
diffs = [test(value) for _ in range(N_TESTS)]
print(value, statistics.mean(diffs))
Sortie:
False 0.464562410987
True 0.0820847168593