Diagrammes de Venn avec R? [fermé]
sont d'autres paquets pour faire des diagrammes Venn dans R en plus du paquet limma.
quelqu'un a des conseils?
En voici notes sur le fait de faire des diagrammes de Venn avec les paquets limma.
8 réponses
Duncan Murdoch a un paquet venn, qui n'est pas sur l'CRAN. (astuce chapeau Gabor Grothendieck)
Vous pouvez également lire à ce sujet dans le "Journal d'un Logiciel Statistique".
Il y a Vennerable paquet sur la R-forge.
source("http://bioconductor.org/biocLite.R")
biocLite(c("graph", "RBGL", "gtools", "xtable"))
install.packages("Vennerable", repos="http://R-Forge.R-project.org")
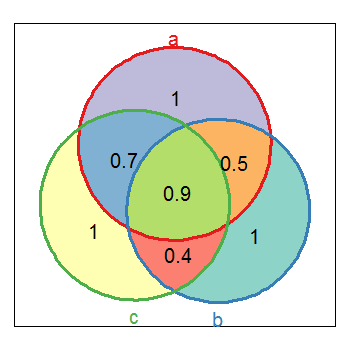
la fonction venn dans le paquet gplots est également utile si vous avez besoin de créer le diagramme de Venn des ensembles 4/5.
j'utilise deux fonctions personnalisées que pour l'astuce. Mon implémentation de venndia trace le diagramme de venn et retourne les listes de chevauchements entre A et B (et C). Voir le code ci-dessous.
Avec ces, vous pouvez
vd <- venndia(A=LETTERS[1:15], B=LETTERS[5:20], getdata=TRUE)
qui va à la fois tracer et retourner les données. vous pouvez désactiver renvoyer les données en faisant
venndia(A=LETTERS[1:15], B=LETTERS[5:20])
depuis getdata est FALSE par défaut. /Daniel
circle <- function(x, y, r, ...) {
ang <- seq(0, 2*pi, length = 100)
xx <- x + r * cos(ang)
yy <- y + r * sin(ang)
polygon(xx, yy, ...)
}
venndia <- function(A, B, C, getdata=FALSE, ...){
cMissing <- missing(C)
if(cMissing){ C <- c() }
unionAB <- union(A, B)
unionAC <- union(A, C)
unionBC <- union(B, C)
uniqueA <- setdiff(A, unionBC)
uniqueB <- setdiff(B, unionAC)
uniqueC <- setdiff(C, unionAB)
intersAB <- setdiff(intersect(A, B), C)
intersAC <- setdiff(intersect(A, C), B)
intersBC <- setdiff(intersect(B, C), A)
intersABC <- intersect(intersect(A, B), intersect(B, C))
nA <- length(uniqueA)
nB <- length(uniqueB)
nC <- length(uniqueC)
nAB <- length(intersAB)
nAC <- length(intersAC)
nBC <- length(intersBC)
nABC <- length(intersABC)
par(mar=c(2, 2, 0, 0))
plot(-10, -10, ylim=c(0, 9), xlim=c(0, 9), axes=FALSE, ...)
circle(x=3, y=6, r=3, col=rgb(1,0,0,.5), border=NA)
circle(x=6, y=6, r=3, col=rgb(0,.5,.1,.5), border=NA)
circle(x=4.5, y=3, r=3, col=rgb(0,0,1,.5), border=NA)
text( x=c(1.2, 7.7, 4.5), y=c(7.8, 7.8, 0.8), c("A", "B", "C"), cex=3, col="gray90" )
text(
x=c(2, 7, 4.5, 4.5, 3, 6, 4.5),
y=c(7, 7, 2 , 7 , 4, 4, 5),
c(nA, nB, nC, nAB, nAC, nBC, nABC),
cex=2
)
if(getdata){
list(A=uniqueA, B=uniqueB, C=uniqueC,
AB=intersAB , AC=intersAC , BC=intersBC ,
ABC=intersABC
)
}
}
cela arrive très tard mais cela pourrait être utile pour d'autres qui cherchent une réponse: VennDiagram, on CRAN ici.
install.packages("VennDiagram")
library(VennDiagram)
peaks1 = RangedData(IRanges(start = c(967654, 2010897, 2496704),
end = c(967754, 2010997, 2496804), names = c("Site1", "Site2", "Site3")),
space = c("1", "2", "3"), strand=as.integer(1),feature=c("a","b","f"))
peaks2 = RangedData(IRanges(start = c(967659, 2010898,2496700,3075866,3123260),
end = c(967869, 2011108, 2496920, 3076166, 3123470),
names = c("t1", "t2", "t3", "t4", "t5")),
space = c("1", "2", "3", "1", "2"), strand = c(1, 1, -1,-1,1), feature=c("a","b","c","d","a"))
makeVennDiagram(RangedDataList(peaks1,peaks2, peaks1, peaks2), NameOfPeaks=c("TF1", "TF2","TF3", "TF4"),
totalTest=100,useFeature=TRUE, main="Venn Diagram",
col = "transparent",fill = c("cornflowerblue", "green", "yellow", "darkorchid1"),
alpha = 0.50,label.col = c("orange", "white", "darkorchid4", "white", "white", "white", "white", "white", "darkblue", "white", "white", "white", "white", "darkgreen", "white"), cat.col = c("darkblue", "darkgreen", "orange", "darkorchid4"))
Voici une référence à une autre version pour les données à 3 variables: http://elliotnoma.wordpress.com/2011/02/09/venn-diagram/
le code est également disponible dans l'emballage colorfulVennPlot: http://cran.r-project.org/web/packages/colorfulVennPlot/index.html
je recommande le paquet VennDiagram: http://cran.r-project.org/web/packages/VennDiagram/VennDiagram.pdf
sur pake 19 vous trouverez 10 pakes de très bons exemples (à la fois avancés et simplifiés). Jusqu'à présent, je n'ai rien trouvé qu'il ne peut pas faire que j'ai besoin de faire.