utilisation de la fonction stat et de facet wrap together in ggplot2 in R
j'essaie de représenter graphiquement les données de type réseau avec ggplot2 puis de superposer une distribution normale sur les données de l'échantillon pour illustrer à quel point les données sous-jacentes sont anormales. Je voudrais avoir le dist normal sur le dessus pour avoir la même moyenne et stdev que le panneau.
voici un exemple:
library(ggplot2)
#make some example data
dd<-data.frame(matrix(rnorm(144, mean=2, sd=2),72,2),c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
#This works
pg <- ggplot(dd) + geom_density(aes(x=Predicted_value)) + facet_wrap(~State_CD)
print(pg)
tous fonctionne très bien et produit un joli trois panneau graphique des données. Comment ajouter le dist normal sur le dessus? Il semble que j'utiliserais stat_function, mais ceci échoue:
#this fails
pg <- ggplot(dd) + geom_density(aes(x=Predicted_value)) + stat_function(fun=dnorm) + facet_wrap(~State_CD)
print(pg)
il semble que la fonction stat_function ne s'entend pas avec la fonctionnalité facet_wrap. Comment je fais pour que ces deux-là jouent gentiment?
------------MODIFIER---------
j'ai essayé d'intégrer des idées à partir de deux des réponses ci-dessous et je ne suis toujours pas là:
en utilisant une combinaison des deux réponses je peux Hacker ensemble ceci:
library(ggplot)
library(plyr)
#make some example data
dd<-data.frame(matrix(rnorm(108, mean=2, sd=2),36,2),c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
DevMeanSt <- ddply(dd, c("State_CD"), function(df)mean(df$Predicted_value))
colnames(DevMeanSt) <- c("State_CD", "mean")
DevSdSt <- ddply(dd, c("State_CD"), function(df)sd(df$Predicted_value) )
colnames(DevSdSt) <- c("State_CD", "sd")
DevStatsSt <- merge(DevMeanSt, DevSdSt)
pg <- ggplot(dd, aes(x=Predicted_value))
pg <- pg + geom_density()
pg <- pg + stat_function(fun=dnorm, colour='red', args=list(mean=DevStatsSt$mean, sd=DevStatsSt$sd))
pg <- pg + facet_wrap(~State_CD)
print(pg)
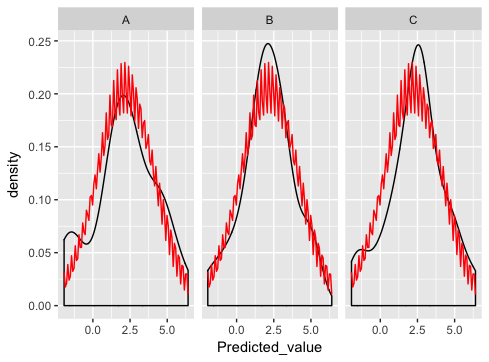
qui est vraiment très proche... sauf que quelque chose ne va pas avec le dist normal traçage:
ce que je fais mal?
4 réponses
stat_function est conçu pour superposer la même fonction dans chaque panneau. (Il n'y a aucun moyen évident de faire correspondre les paramètres de la fonction avec les différents panneaux).
dd <- data.frame(
predicted = rnorm(72, mean = 2, sd = 2),
state = rep(c("A", "B", "C"), each = 24)
)
grid <- with(dd, seq(min(predicted), max(predicted), length = 100))
normaldens <- ddply(dd, "state", function(df) {
data.frame(
predicted = grid,
density = dnorm(grid, mean(df$predicted), sd(df$predicted))
)
})
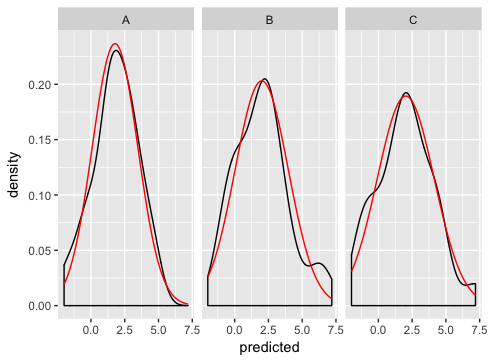
ggplot(dd, aes(predicted)) +
geom_density() +
geom_line(aes(y = density), data = normaldens, colour = "red") +
facet_wrap(~ state)
je pense que vous avez besoin de fournir plus d'informations. Cela semble fonctionner:
pg <- ggplot(dd, aes(Predicted_value)) ## need aesthetics in the ggplot
pg <- pg + geom_density()
## gotta provide the arguments of the dnorm
pg <- pg + stat_function(fun=dnorm, colour='red',
args=list(mean=mean(dd$Predicted_value), sd=sd(dd$Predicted_value)))
## wrap it!
pg <- pg + facet_wrap(~State_CD)
pg
nous fournissons la même moyenne et le même paramètre sd pour chaque panneau. L'obtention de moyens spécifiques au panneau et d'écarts-types est laissée au lecteur comme un exercice* ;)
' * ' en d'autres termes, Je ne sais pas comment faire...
je pense que votre meilleur pari est de tracer la ligne manuellement avec geom_line.
dd<-data.frame(matrix(rnorm(144, mean=2, sd=2),72,2),c(rep("A",24),rep("B",24),rep("C",24)))
colnames(dd) <- c("x_value", "Predicted_value", "State_CD")
dd$Predicted_value<-dd$Predicted_value*as.numeric(dd$State_CD) #make different by state
##Calculate means and standard deviations by level
means<-as.numeric(by(dd[,2],dd$State_CD,mean))
sds<-as.numeric(by(dd[,2],dd$State_CD,sd))
##Create evenly spaced evaluation points +/- 3 standard deviations away from the mean
dd$vals<-0
for(i in 1:length(levels(dd$State_CD))){
dd$vals[dd$State_CD==levels(dd$State_CD)[i]]<-seq(from=means[i]-3*sds[i],
to=means[i]+3*sds[i],
length.out=sum(dd$State_CD==levels(dd$State_CD)[i]))
}
##Create normal density points
dd$norm<-with(dd,dnorm(vals,means[as.numeric(State_CD)],
sds[as.numeric(State_CD)]))
pg <- ggplot(dd, aes(Predicted_value))
pg <- pg + geom_density()
pg <- pg + geom_line(aes(x=vals,y=norm),colour="red") #Add in normal distribution
pg <- pg + facet_wrap(~State_CD,scales="free")
pg
si vous ne voulez pas générer la ligne de distribution normale-graphe "à la main", utilisez toujours stat_function, et afficher les graphiques côte à côte -- alors vous pouvez envisager d'utiliser la fonction "multiplot" publiée sur "Cookbook for R" comme une alternative à facet_wrap. Vous pouvez copier le code multiplot à votre projet de là.
après avoir copié le code, faites ce qui suit:
# Some fake data (copied from hadley's answer)
dd <- data.frame(
predicted = rnorm(72, mean = 2, sd = 2),
state = rep(c("A", "B", "C"), each = 24)
)
# Split the data by state, apply a function on each member that converts it into a
# plot object, and return the result as a vector.
plots <- lapply(split(dd,dd$state),FUN=function(state_slice){
# The code here is the plot code generation. You can do anything you would
# normally do for a single plot, such as calling stat_function, and you do this
# one slice at a time.
ggplot(state_slice, aes(predicted)) +
geom_density() +
stat_function(fun=dnorm,
args=list(mean=mean(state_slice$predicted),
sd=sd(state_slice$predicted)),
color="red")
})
# Finally, present the plots on 3 columns.
multiplot(plotlist = plots, cols=3)