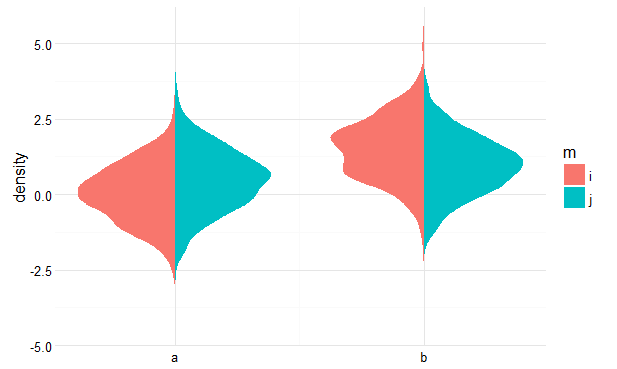Split violin plot avec ggplot2
<!-Je voudrais créer un graphe de densité de violon divisé en utilisant ggplot, comme le quatrième exemple sur cette page de la documentation seaborn.
Ici est quelques données:
set.seed(20160229)
my_data = data.frame(
y=c(rnorm(1000), rnorm(1000, 0.5), rnorm(1000, 1), rnorm(1000, 1.5)),
x=c(rep('a', 2000), rep('b', 2000)),
m=c(rep('i', 1000), rep('j', 2000), rep('i', 1000))
)
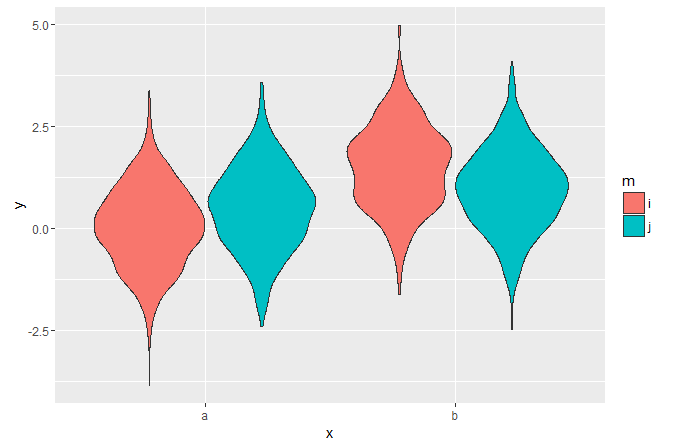
je peux parcelle esquivé les violons comme ceci:
library('ggplot2')
ggplot(my_data, aes(x, y, fill=m)) +
geom_violin()
j'ai trouvé un base r solution graphique mais la fonction est assez longue et je veux mettre en évidence les modes de distribution, qui sont faciles à ajouter comme couches supplémentaires dans ggplot mais seront plus difficiles à faire si j'ai besoin de comprendre comment éditer cette fonction.
29
demandé sur
Mike Wise
2016-03-01 10:49:38
2 réponses
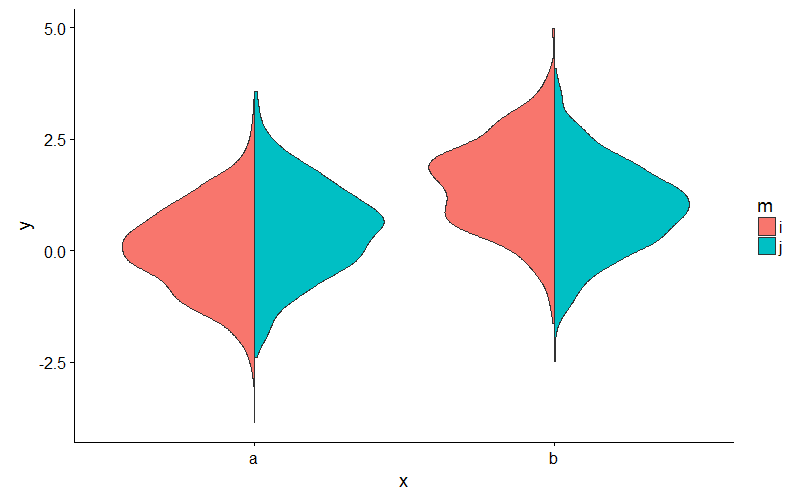
Ou, pour éviter de jongler avec les densités, vous pouvez prolonger comme ceci:
GeomSplitViolin <- ggproto("GeomSplitViolin", GeomViolin,
draw_group = function(self, data, ..., draw_quantiles = NULL) {
data <- transform(data, xminv = x - violinwidth * (x - xmin), xmaxv = x + violinwidth * (xmax - x))
grp <- data[1, "group"]
newdata <- plyr::arrange(transform(data, x = if (grp %% 2 == 1) xminv else xmaxv), if (grp %% 2 == 1) y else -y)
newdata <- rbind(newdata[1, ], newdata, newdata[nrow(newdata), ], newdata[1, ])
newdata[c(1, nrow(newdata) - 1, nrow(newdata)), "x"] <- round(newdata[1, "x"])
if (length(draw_quantiles) > 0 & !scales::zero_range(range(data$y))) {
stopifnot(all(draw_quantiles >= 0), all(draw_quantiles <=
1))
quantiles <- ggplot2:::create_quantile_segment_frame(data, draw_quantiles)
aesthetics <- data[rep(1, nrow(quantiles)), setdiff(names(data), c("x", "y")), drop = FALSE]
aesthetics$alpha <- rep(1, nrow(quantiles))
both <- cbind(quantiles, aesthetics)
quantile_grob <- GeomPath$draw_panel(both, ...)
ggplot2:::ggname("geom_split_violin", grid::grobTree(GeomPolygon$draw_panel(newdata, ...), quantile_grob))
}
else {
ggplot2:::ggname("geom_split_violin", GeomPolygon$draw_panel(newdata, ...))
}
})
geom_split_violin <- function(mapping = NULL, data = NULL, stat = "ydensity", position = "identity", ...,
draw_quantiles = NULL, trim = TRUE, scale = "area", na.rm = FALSE,
show.legend = NA, inherit.aes = TRUE) {
layer(data = data, mapping = mapping, stat = stat, geom = GeomSplitViolin,
position = position, show.legend = show.legend, inherit.aes = inherit.aes,
params = list(trim = trim, scale = scale, draw_quantiles = draw_quantiles, na.rm = na.rm, ...))
}
et utilisez le nouveau geom_split_violin comme ceci:
ggplot(my_data, aes(x, y, fill = m)) + geom_split_violin()
24
répondu
jan-glx
2018-10-04 20:56:20
vous pouvez y parvenir en calculant vous-même les densités à l'avance, puis en traçant des polygones. Voir ci-dessous pour une idée générale. Il ne devrait pas être trop difficile d'écrire cela dans une fonction.
Obtenir des densités
library(dplyr)
pdat <- my_data %>%
group_by(x, m) %>%
do(data.frame(loc = density(.$y)$x,
dens = density(.$y)$y))
densité de retournement et de décalage pour les groupes
pdat$dens <- ifelse(pdat$m == 'i', pdat$dens * -1, pdat$dens)
pdat$dens <- ifelse(pdat$x == 'b', pdat$dens + 1, pdat$dens)
Complot
ggplot(pdat, aes(dens, loc, fill = m, group = interaction(m, x))) +
geom_polygon() +
scale_x_continuous(breaks = 0:1, labels = c('a', 'b')) +
ylab('density') +
theme_minimal() +
theme(axis.title.x = element_blank())
Résultat
37
répondu
Axeman
2016-03-01 09:37:03