régression sigmoïde avec scipy, numpy, python, etc.
j'ai deux variables (x et y) qui ont une relation quelque peu sigmoïdale l'une avec l'autre, et j'ai besoin de trouver une sorte d'équation de prédiction qui me permettra de prédire la valeur de y, étant donné n'importe quelle valeur de X. Mon équation de prédiction doit montrer la relation quelque peu sigmoïdale entre les deux variables. Par conséquent, je ne peux pas me contenter d'une équation de régression linéaire qui produit une ligne. J'ai besoin de voir l'progressive, curviligne changement de pente se produit à la fois le droit et à gauche du graphique des deux variables.
j'ai commencé à utiliser numpy.polyfit après googling régression curvilinéaire et python, mais qui m'a donné les résultats terribles, vous pouvez voir si vous exécutez le code ci-dessous. quelqu'un peut-il me montrer comment réécrire le code ci-dessous pour obtenir le type d'équation de régression sigmoïdale que je veux?
si vous exécutez le code ci-dessous, vous pouvez voir qu'il donne une parabole orientée vers le bas, ce qui n'est pas ce que la relation entre mes variables devrait ressembler. Au lieu de cela, il devrait y avoir plus d'une relation sigmoïdale entre mes deux variables, mais avec une correspondance étroite avec les données que j'utilise dans le code ci-dessous. Les données dans le code ci-dessous sont des moyens d'une étude de recherche de grand-échantillon, de sorte qu'ils pack Plus de puissance statistique que leurs cinq points de données pourrait suggérer. Je ne dispose pas des données réelles de l'étude de recherche sur grand échantillon, mais j'ai les moyens ci-dessous et leurs écarts-types(que je ne montre pas). Je préférerait simplement tracer une fonction simple avec les données moyennes énumérées ci-dessous, mais le code pourrait devenir plus complexe si la complexité offrirait des améliorations substantielles.
Comment puis-je changer mon code pour montrer la meilleure correspondance d'une fonction sigmoïdale, de préférence en utilisant scipy, numpy et python? Voici la version actuelle de mon code, qui doit être corrigée:
import numpy as np
import matplotlib.pyplot as plt
# Create numpy data arrays
x = np.array([821,576,473,377,326])
y = np.array([255,235,208,166,157])
# Use polyfit and poly1d to create the regression equation
z = np.polyfit(x, y, 3)
p = np.poly1d(z)
xp = np.linspace(100, 1600, 1500)
pxp=p(xp)
# Plot the results
plt.plot(x, y, '.', xp, pxp, '-')
plt.ylim(140,310)
plt.xlabel('x')
plt.ylabel('y')
plt.grid(True)
plt.show()
MODIFIER ci-DESSOUS: (Re-cadré de la question)
votre réponse, et sa vitesse, sont très impressionnantes. Merci, unutbu. Mais, afin de produire des résultats plus valides, je dois reformuler mes valeurs de données. Cela signifie que les valeurs de x sont rejouées en pourcentage de la valeur de X max, tandis que les valeurs de y sont rejouées en pourcentage des valeurs de x dans les données originales. J'ai essayé de le faire avec votre code, et est venu avec le code suivant:
import numpy as np
import matplotlib.pyplot as plt
import scipy.optimize
# Create numpy data arrays
'''
# Comment out original data
#x = np.array([821,576,473,377,326])
#y = np.array([255,235,208,166,157])
'''
# Re-calculate x values as a percentage of the first (maximum)
# original x value above
x = np.array([1.000,0.702,0.576,0.459,0.397])
# Recalculate y values as a percentage of their respective x values
# from original data above
y = np.array([0.311,0.408,0.440,0.440,0.482])
def sigmoid(p,x):
x0,y0,c,k=p
y = c / (1 + np.exp(-k*(x-x0))) + y0
return y
def residuals(p,x,y):
return y - sigmoid(p,x)
p_guess=(600,200,100,0.01)
(p,
cov,
infodict,
mesg,
ier)=scipy.optimize.leastsq(residuals,p_guess,args=(x,y),full_output=1,warning=True)
'''
# comment out original xp to allow for better scaling of
# new values
#xp = np.linspace(100, 1600, 1500)
'''
xp = np.linspace(0, 1.1, 1100)
pxp=sigmoid(p,xp)
x0,y0,c,k=p
print('''
x0 = {x0}
y0 = {y0}
c = {c}
k = {k}
'''.format(x0=x0,y0=y0,c=c,k=k))
# Plot the results
plt.plot(x, y, '.', xp, pxp, '-')
plt.ylim(0,1)
plt.xlabel('x')
plt.ylabel('y')
plt.grid(True)
plt.show()
pouvez-vous me montrer comment corriger cette révision code?
NOTE: en relançant les données, j'ai essentiellement fait tourner le sigmoïde 2d (x,y) autour de l'axe z de 180 degrés. De plus, le 1.000 n'est pas vraiment un maximum des valeurs de X. Au lieu de cela, 1.000 est une moyenne de la gamme de valeurs de différents participants à l'essai dans une condition d'essai maximale.
SECOND EDIT BELOW:
Merci, ubuntu. J'ai lu attentivement votre code et j'ai regardé les aspects de la TI dans la documentation de scipy. Puisque votre nom semble apparaître comme un auteur de la documentation scipy, j'espère que vous pouvez répondre aux questions suivantes:
1.) Est-ce que leastsq() appelle residuals(), qui renvoie alors la différence entre le vecteur y d'entrée et le vecteur y retourné par la fonction sigmoid ()? Dans l'affirmative, comment tient-il compte de la différence de longueur entre le vecteur y d'entrée et le vecteur y retourné par la fonction sigmoid ()?
2.) Il on dirait que je peux appeler leastsq () pour n'importe quelle équation mathématique, tant que j'accède à cette équation mathématique par une fonction de résidus, qui à son tour appelle la fonction mathématique. Est-ce vrai?
3.) De plus, je remarque que p_guess a le même nombre d'éléments que P. Cela signifie-t-il que les quatre éléments de p_guess correspondent respectivement aux valeurs retournées par x0, y0, c et k?
4.) Est le p qui est envoyé comme argument aux fonctions residuals() et sigmoid() même p qui sera produit par leastsq(), et la fonction leastsq() utilise ce p en interne avant de le retourner?
5.) Peut-p et p_guess avoir n'importe quel nombre d'éléments, en fonction de la complexité de l'équation utilisée comme un modèle, aussi longtemps que le nombre d'éléments de p est égal au nombre d'éléments dans p_guess?
4 réponses
en utilisant scipy.optimiser.leastsq:
import numpy as np
import matplotlib.pyplot as plt
import scipy.optimize
def sigmoid(p,x):
x0,y0,c,k=p
y = c / (1 + np.exp(-k*(x-x0))) + y0
return y
def residuals(p,x,y):
return y - sigmoid(p,x)
def resize(arr,lower=0.0,upper=1.0):
arr=arr.copy()
if lower>upper: lower,upper=upper,lower
arr -= arr.min()
arr *= (upper-lower)/arr.max()
arr += lower
return arr
# raw data
x = np.array([821,576,473,377,326],dtype='float')
y = np.array([255,235,208,166,157],dtype='float')
x=resize(-x,lower=0.3)
y=resize(y,lower=0.3)
print(x)
print(y)
p_guess=(np.median(x),np.median(y),1.0,1.0)
p, cov, infodict, mesg, ier = scipy.optimize.leastsq(
residuals,p_guess,args=(x,y),full_output=1,warning=True)
x0,y0,c,k=p
print('''\
x0 = {x0}
y0 = {y0}
c = {c}
k = {k}
'''.format(x0=x0,y0=y0,c=c,k=k))
xp = np.linspace(0, 1.1, 1500)
pxp=sigmoid(p,xp)
# Plot the results
plt.plot(x, y, '.', xp, pxp, '-')
plt.xlabel('x')
plt.ylabel('y',rotation='horizontal')
plt.grid(True)
plt.show()
les rendements
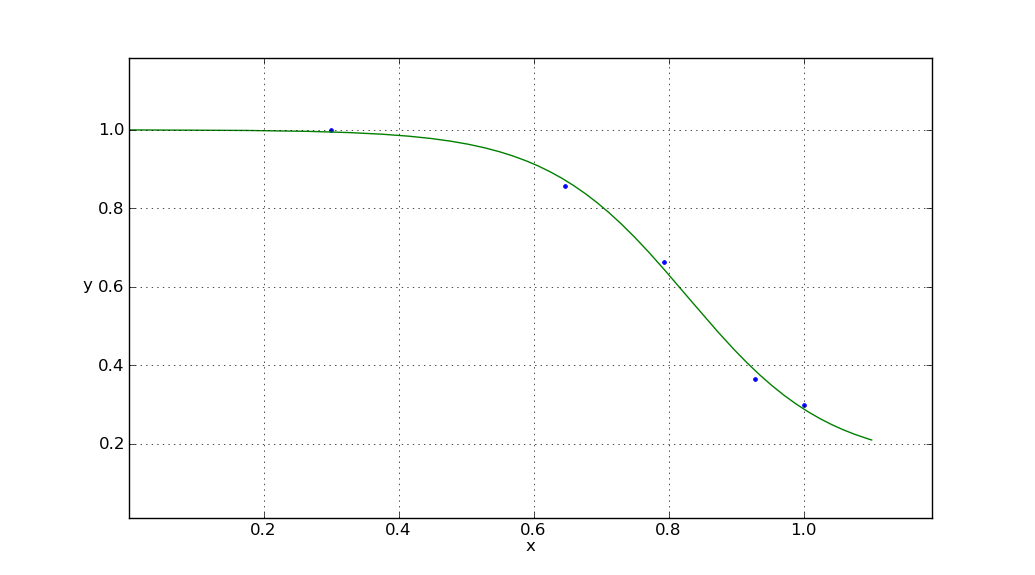
avec des paramètres sigmoïdes
x0 = 0.826964424481
y0 = 0.151506745435
c = 0.848564826467
k = -9.54442292022
notez que pour les versions plus récentes de scipy (par exemple 0.9) il y a aussi le scipy.optimiser.curve_fit fonction plus facile à utiliser que leastsq. Une discussion pertinente sur l'ajustement des sigmoïdes à l'aide de curve_fit peut être trouvé ici.
Edit: A resize la fonction a été ajoutée pour que les données brutes puissent être rééchelonnées et déplacées pour s'adapter à n'importe quelle boîte de limite désirée.
"votre nom semble pop up comme un écrivain de la documentation de scipy"
avertissement: Je ne suis pas un rédacteur de documentation scipy. Je suis juste un utilisateur, et un novice. Beaucoup de ce que je connais leastsq vient de la lecture ce tutoriel, écrit par Travis Oliphant.
1.) Faire leastsq() appel de résidus(), qui renvoie alors la différence entre le vecteur d'entrée y et le vecteur vecteur y retourné par le sigmoïde() la fonction?
Oui! exactement.
si c'est le cas, comment tient-il compte de la différence dans la longueur de l'entrée le vecteur y et le vecteur y sont retournés par le sigmoïde() la fonction?
Les longueurs sont identiques:
In [138]: x
Out[138]: array([821, 576, 473, 377, 326])
In [139]: y
Out[139]: array([255, 235, 208, 166, 157])
In [140]: p=(600,200,100,0.01)
In [141]: sigmoid(p,x)
Out[141]:
array([ 290.11439268, 244.02863507, 221.92572521, 209.7088641 ,
206.06539033])
une des choses merveilleuses à propos de Numpy est qu'il permet d'écrire des équations "vectorielles" qui fonctionnent sur des tableaux entiers.
y = c / (1 + np.exp(-k*(x-x0))) + y0
pourrait ressembler à cela fonctionne sur les flotteurs (en effet il le ferait) mais si vous faites x un tableau numpy, et c,k,x0,y0 flotteurs, alors l'équation définit y pour être un tableau numpy de la même forme que x. Donc sigmoid(p,x) renvoie un numpy array. Il y a une explication plus complète de la façon dont cela fonctionne dans le nummpybook (lecture requise pour les utilisateurs sérieux de numpy).
2.) Il semble que je puisse appeler leastsq () pour n'importe quelle équation mathématique, aussi longtemps que je accéder à cette équation mathématique au moyen d'un les fonctions résiduelles, qui à leur tour appelle la fonction mathématique. Est-ce vrai?
Vrai. leastsq tente de minimiser la somme des carrés des résidus (les différences). Il recherche le paramètre-espace (toutes les valeurs possibles de p) à la recherche de l' p qui minimise cette somme de carrés. x et y envoi residuals, sont vos données brutes. Ils sont fixes. Ils ne changent pas. C'est le ps (les paramètres de la fonction sigmoïde)leastsq tente de minimiser.
3.) De plus, je remarque que p_guess a le même nombre d'éléments que P. Does cela signifie que les quatre éléments de p_guess correspond dans l'ordre, respectivement, avec les valeurs retournées par x0, y0, c, et k?
Exactement! Comme la méthode de Newton,leastsq besoins une première supposition pour p. Vous fournir en tant que p_guess. Quand vous voyez
scipy.optimize.leastsq(residuals,p_guess,args=(x,y))
vous pouvez penser que dans le cadre de l'algorithme de leastsq (en fait l'algorithme de Levenburg-Marquardt) comme premier passage, leastsq appelle residuals(p_guess,x,y).
Remarquez la similitude visuelle entre
(residuals,p_guess,args=(x,y))
et
residuals(p_guess,x,y)
Il peut vous aider à vous souvenir de l'ordre et le sens de l'argumentation leastsq.
residuals, comme sigmoid renvoie un numpy array. Valeur dans le tableau sont au carré, puis additionnées. C'est le nombre de battre. p_guess est alors varié comme leastsq regarde pour un ensemble de valeurs qui minimise residuals(p_guess,x,y).
4.) Est le p qui est envoyé comme argument aux résidus () et sigmoid() fonctionne avec le même p que sera produit par leastsq (), et le la fonction leastsq() utilise ce p en interne avant de le retourner?
eh Bien, pas exactement. Comme vous le savez maintenant, p_guess est varié comme leastsq recherche p valeur qui minimise residuals(p,x,y). p (re, p_guess) envoyé à leastsq a la même forme que l' p qui est retournée par leastsq. Évidemment, les valeurs devraient être différentes sauf si vous êtes un enfer d'une devinette :)
5.) P et p_guess peuvent avoir n'importe quel nombre d'éléments, selon le la complexité de l'équation utilisée comme un modèle, tant que le nombre de les éléments de p est égal au nombre des éléments en p_guess?
Oui. Je n'ai pas un test de contrainte leastsq pour un très grand nombre de paramètres, mais c'est un outil extrêmement puissant.
je ne pense pas que vous allez obtenir de bons résultats avec une approximation polynomiale de degré -- depuis tous les polynômes vont à l'infini pour un X suffisamment grand et petit, mais une courbe sigmoïde approchera asymptotiquement une valeur finie dans chaque direction.
Je ne suis pas un programmeur Python, donc je ne sais pas si numpy a un ajustement de courbe plus général routine. Si vous devez rouler votre propre, peut-être cet article sur régression Logistique vous donnera quelques idées.
Pour la régression logistique en Python, le scikits-apprendre expose le code de raccord haute performance:
http://scikit-learn.sourceforge.net/modules/linear_model.html#logistic-regression
def sigmoid(x, k, x0):
return 1.0 / (1 + np.exp(-k * (x - x0)))
# Parameters of the true function
n_samples = 1000
true_x0 = 15
true_k = 1.5
sigma = 0.2
# Build the true function and add some noise
x = np.linspace(0, 30, num=n_samples)
y = sigmoid(x, k=true_k, x0=true_x0)
y_with_noise = y + sigma * np.random.randn(n_samples)
# Sample the data from the real function (this will be your data)
some_points = np.random.choice(1000, size=30) # take 30 data points
xdata = x[some_points]
ydata = y_with_noise[some_points]
# Fit the curve
popt, pcov = curve_fit(return_sigmoid, xdata, ydata)
estimated_k, estimated_x0 = popt
# Plot the fitted curve
y_fitted = sigmoid(x, k=estimated_k, x0=estimated_x0)
# Plot everything for illustration
fig = plt.figure()
ax = fig.add_subplot(111)
ax.plot(x, y_fitted, '--', label='fitted')
ax.plot(x, y, '-', label='true')
ax.plot(xdata, ydata, 'o', label='samples')
ax.legend()
Le résultat de ceci est montré dans la figure suivante:
