Méthode pythonique de détection des valeurs aberrantes dans les données d'observation unidimensionnelles
pour les données Données Données, je veux définir les valeurs aberrantes (définies par un niveau de confiance de 95% ou une fonction de quantile de 95% ou tout ce qui est requis) comme des valeurs nan. Voici mes données et le code que j'utilise actuellement. Je serais heureux si quelqu'un pouvait m'expliquer davantage.
import numpy as np, matplotlib.pyplot as plt
data = np.random.rand(1000)+5.0
plt.plot(data)
plt.xlabel('observation number')
plt.ylabel('recorded value')
plt.show()
5 réponses
le problème avec l'utilisation de percentile est que les points identifiés comme valeurs aberrantes sont fonction de la taille de votre échantillon.
il y a un grand nombre de façons de tester les valeurs aberrantes, et vous devriez réfléchir à la façon de les classer. Idéalement, vous devriez utiliser l'information a priori (par exemple, " tout ce qui est au-dessus ou au-dessous de cette valeur est irréaliste parce que...")
toutefois, un critère commun, qui n'est pas trop déraisonnable, pour les valeurs aberrantes est de supprimer les points en fonction de leur valeur. "écart médian absolu".
Voici une implémentation pour le cas n-dimensionnel (à partir d'un code pour un papier ici: https://github.com/joferkington/oost_paper_code/blob/master/utilities.py ):
def is_outlier(points, thresh=3.5):
"""
Returns a boolean array with True if points are outliers and False
otherwise.
Parameters:
-----------
points : An numobservations by numdimensions array of observations
thresh : The modified z-score to use as a threshold. Observations with
a modified z-score (based on the median absolute deviation) greater
than this value will be classified as outliers.
Returns:
--------
mask : A numobservations-length boolean array.
References:
----------
Boris Iglewicz and David Hoaglin (1993), "Volume 16: How to Detect and
Handle Outliers", The ASQC Basic References in Quality Control:
Statistical Techniques, Edward F. Mykytka, Ph.D., Editor.
"""
if len(points.shape) == 1:
points = points[:,None]
median = np.median(points, axis=0)
diff = np.sum((points - median)**2, axis=-1)
diff = np.sqrt(diff)
med_abs_deviation = np.median(diff)
modified_z_score = 0.6745 * diff / med_abs_deviation
return modified_z_score > thresh
c'est très similaire à une de mes réponses précédentes , mais je voulais illustrer l'effet de la taille de l'échantillon en détail.
comparons une valeur aberrante basée sur un percentile test (semblable à la réponse de @CTZhu) avec un test d'écart médian absolu (MAD) pour une variété de tailles d'échantillon différentes:
import numpy as np
import matplotlib.pyplot as plt
import seaborn as sns
def main():
for num in [10, 50, 100, 1000]:
# Generate some data
x = np.random.normal(0, 0.5, num-3)
# Add three outliers...
x = np.r_[x, -3, -10, 12]
plot(x)
plt.show()
def mad_based_outlier(points, thresh=3.5):
if len(points.shape) == 1:
points = points[:,None]
median = np.median(points, axis=0)
diff = np.sum((points - median)**2, axis=-1)
diff = np.sqrt(diff)
med_abs_deviation = np.median(diff)
modified_z_score = 0.6745 * diff / med_abs_deviation
return modified_z_score > thresh
def percentile_based_outlier(data, threshold=95):
diff = (100 - threshold) / 2.0
minval, maxval = np.percentile(data, [diff, 100 - diff])
return (data < minval) | (data > maxval)
def plot(x):
fig, axes = plt.subplots(nrows=2)
for ax, func in zip(axes, [percentile_based_outlier, mad_based_outlier]):
sns.distplot(x, ax=ax, rug=True, hist=False)
outliers = x[func(x)]
ax.plot(outliers, np.zeros_like(outliers), 'ro', clip_on=False)
kwargs = dict(y=0.95, x=0.05, ha='left', va='top')
axes[0].set_title('Percentile-based Outliers', **kwargs)
axes[1].set_title('MAD-based Outliers', **kwargs)
fig.suptitle('Comparing Outlier Tests with n={}'.format(len(x)), size=14)
main()
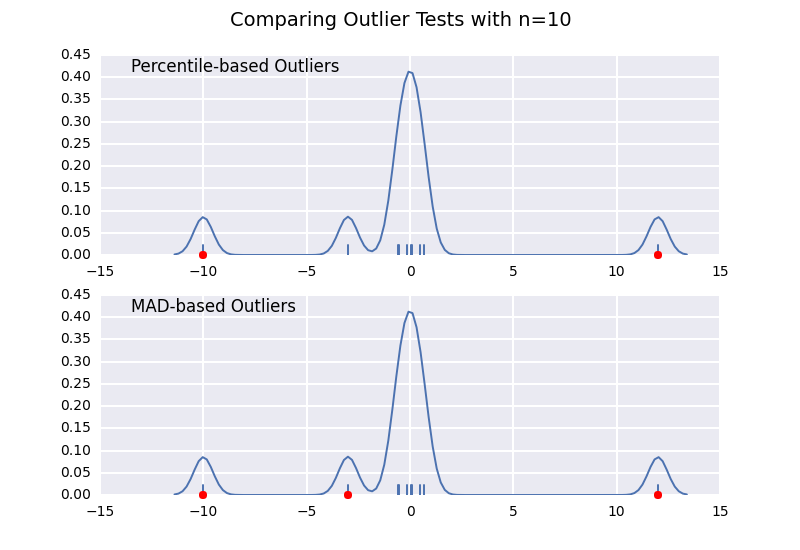
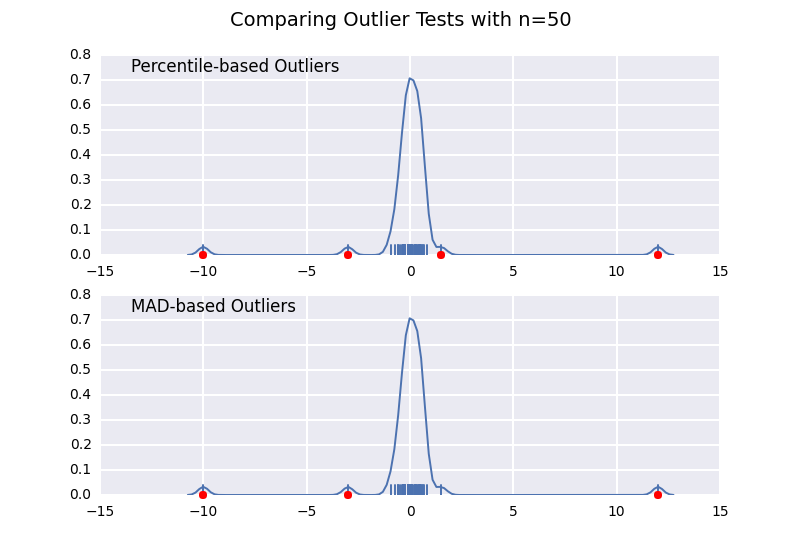
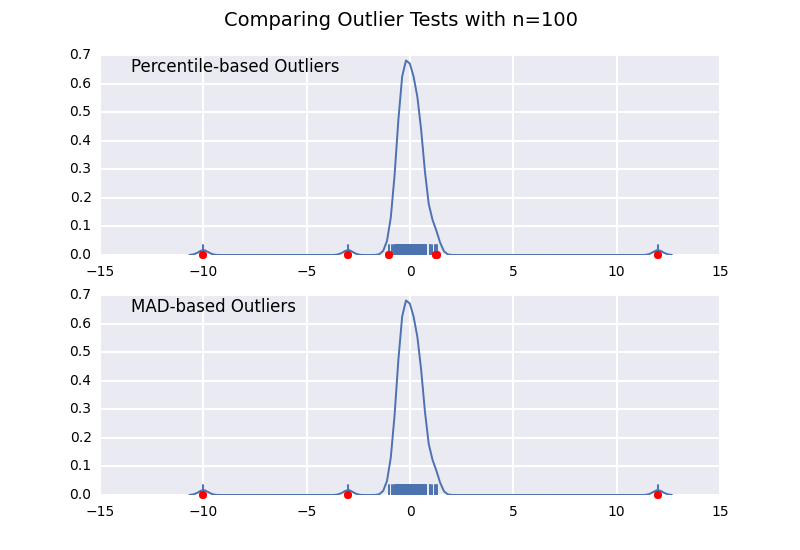
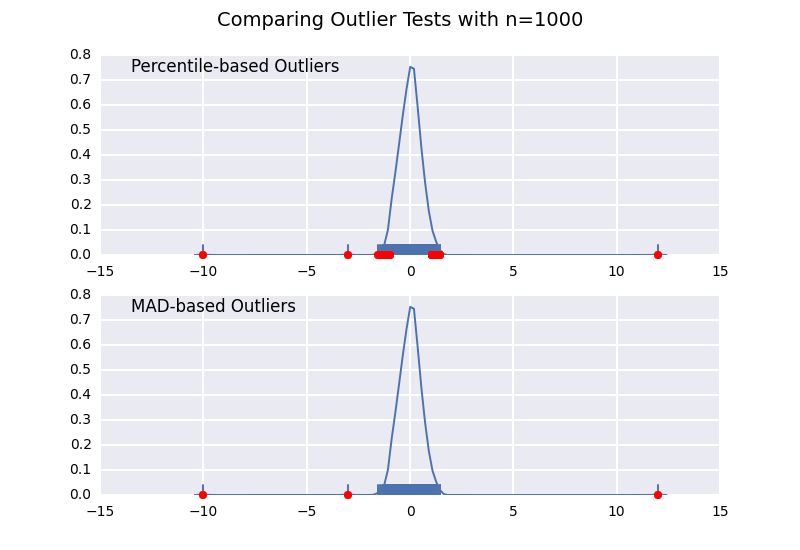
Notez que le classificateur basé sur le MAD fonctionne correctement quelle que soit la taille de l'échantillon, tandis que le classificateur basé sur le percentile classifie plus de points que la taille de l'échantillon est plus grande, indépendamment du fait qu'ils soient ou non des valeurs aberrantes.
j'ai adapté le code de http://eurekastatistics.com/using-the-median-absolute-deviation-to-find-outliers et il donne les mêmes résultats que Joe Kington, mais utilise la distance L1 au lieu de la distance L2, et a le soutien pour les distributions asymétriques. Le code R original n'avait pas le multiplicateur 0.6745 de Joe, donc j'ai aussi ajouté que pour la cohérence dans ce thread. Pas sûr à 100% Si c'est nécessaire, mais fait la comparaison pommes-à-pommes.
def doubleMADsfromMedian(y,thresh=3.5):
# warning: this function does not check for NAs
# nor does it address issues when
# more than 50% of your data have identical values
m = np.median(y)
abs_dev = np.abs(y - m)
left_mad = np.median(abs_dev[y <= m])
right_mad = np.median(abs_dev[y >= m])
y_mad = left_mad * np.ones(len(y))
y_mad[y > m] = right_mad
modified_z_score = 0.6745 * abs_dev / y_mad
modified_z_score[y == m] = 0
return modified_z_score > thresh
la Détection de valeurs aberrantes dans un dimensionnelle des données dépend de sa distribution
1- Distribution Normale :
- les valeurs des données sont distribuées de façon presque égale dans l'intervalle prévu: Dans ce cas, vous utilisez facilement toutes les méthodes qui comprennent moyenne ,comme l'intervalle de confiance de 3 ou 2 écarts-types(95% ou 99,7%) en conséquence pour un données réparties (théorème de la limite centrale et distribution d'échantillonnage de la moyenne de l'échantillon).Je est une méthode très efficace. Expliqué dans la Khan Academy, des statistiques et des Probabilités d'échantillonnage de la distribution de la bibliothèque.
une autre façon est l'intervalle de prédiction si vous voulez l'intervalle de confiance des points de données plutôt que la moyenne.
-
les valeurs des données sont distribuées au hasard dans une fourchette de 151950920" : moyenne peut ne pas être un représentation équitable des données, parce que la moyenne est facilement influencée par des valeurs aberrantes (très petites ou grandes valeurs dans l'ensemble des données qui ne sont pas typiques) La médiane est une autre façon de mesurer le centre numérique de l'ensemble de données.
médiane déviation absolue - une méthode qui mesure la distance de tous les points de la médiane en termes de distance médiane http://www.itl.nist.gov/div898/handbook/eda/section3/eda35h.htm - has une bonne explication comme expliqué dans la réponse de Joe Kington ci-dessus
2 - Distribution symétrique : encore une fois L'écart médian absolu est une bonne méthode si le calcul de z-score et le seuil est modifié en conséquence
explication : http://eurekastatistics.com/using-the-median-absolute-deviation-to-find-outliers /
3-asymétrique Distribution: double MAD-Double médiane écart absolu Explication dans le lien ci-dessus
Attacher mon code python pour la référence :
def is_outlier_doubleMAD(self,points):
"""
FOR ASSYMMETRIC DISTRIBUTION
Returns : filtered array excluding the outliers
Parameters : the actual data Points array
Calculates median to divide data into 2 halves.(skew conditions handled)
Then those two halves are treated as separate data with calculation same as for symmetric distribution.(first answer)
Only difference being , the thresholds are now the median distance of the right and left median with the actual data median
"""
if len(points.shape) == 1:
points = points[:,None]
median = np.median(points, axis=0)
medianIndex = (points.size/2)
leftData = np.copy(points[0:medianIndex])
rightData = np.copy(points[medianIndex:points.size])
median1 = np.median(leftData, axis=0)
diff1 = np.sum((leftData - median1)**2, axis=-1)
diff1 = np.sqrt(diff1)
median2 = np.median(rightData, axis=0)
diff2 = np.sum((rightData - median2)**2, axis=-1)
diff2 = np.sqrt(diff2)
med_abs_deviation1 = max(np.median(diff1),0.000001)
med_abs_deviation2 = max(np.median(diff2),0.000001)
threshold1 = ((median-median1)/med_abs_deviation1)*3
threshold2 = ((median2-median)/med_abs_deviation2)*3
#if any threshold is 0 -> no outliers
if threshold1==0:
threshold1 = sys.maxint
if threshold2==0:
threshold2 = sys.maxint
#multiplied by a factor so that only the outermost points are removed
modified_z_score1 = 0.6745 * diff1 / med_abs_deviation1
modified_z_score2 = 0.6745 * diff2 / med_abs_deviation2
filtered1 = []
i = 0
for data in modified_z_score1:
if data < threshold1:
filtered1.append(leftData[i])
i += 1
i = 0
filtered2 = []
for data in modified_z_score2:
if data < threshold2:
filtered2.append(rightData[i])
i += 1
filtered = filtered1 + filtered2
return filtered
utilisez np.percentile comme @Martin l'a suggéré:
In [33]:
P=np.percentile(A, [2.5, 97.5])
In [34]:
A[(P[0]<A)&(P[1]>A)] #or =>, <= for within 95%
A[(P[0]>A)|(P[1]<A)]=np.nan #to set the outliners to np.nan
une solution simple peut aussi être, en enlevant quelque chose qui en dehors de 2 écarts-types (ou 1.96):
import random
def outliers(tmp):
"""tmp is a list of numbers"""
outs = []
mean = sum(tmp)/(1.0*len(tmp))
var = sum((tmp[i] - mean)**2 for i in range(0, len(tmp)))/(1.0*len(tmp))
std = var**0.5
outs = [tmp[i] for i in range(0, len(tmp)) if abs(tmp[i]-mean) > 1.96*std]
return outs
lst = [random.randrange(-10, 55) for _ in range(40)]
print lst
print outliers(lst)