Python insert numpy array dans la base de données sqlite3
j'essaie de stocker un tableau numpy d'environ 1000 flotteurs dans une base de données sqlite3 mais je continue à obtenir l'erreur "InterfaceError: Error binding parameter 1 - probably unsupported type".
j'étais sous l'impression qu'un type de données BLOB pourrait être n'importe quoi mais il ne fonctionne certainement pas avec un tableau de numpy. Voici ce que j'ai essayé:
import sqlite3 as sql
import numpy as np
con = sql.connect('test.bd',isolation_level=None)
cur = con.cursor()
cur.execute("CREATE TABLE foobar (id INTEGER PRIMARY KEY, array BLOB)")
cur.execute("INSERT INTO foobar VALUES (?,?)", (None,np.arange(0,500,0.5)))
con.commit()
y a-t-il un autre module que je peux utiliser pour obtenir le tableau de numpy dans la table? Ou Puis-je convertir le tableau de numpy dans une autre forme Python (comme une liste ou une chaîne que je peux partager) que sqlite acceptera? La Performance n'est pas une priorité. J'ai juste envie de travailler!
Merci!
4 réponses
vous pouvez enregistrer un nouveau array type de données avec sqlite3:
import sqlite3
import numpy as np
import io
def adapt_array(arr):
"""
http://stackoverflow.com/a/31312102/190597 (SoulNibbler)
"""
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read())
def convert_array(text):
out = io.BytesIO(text)
out.seek(0)
return np.load(out)
# Converts np.array to TEXT when inserting
sqlite3.register_adapter(np.ndarray, adapt_array)
# Converts TEXT to np.array when selecting
sqlite3.register_converter("array", convert_array)
x = np.arange(12).reshape(2,6)
con = sqlite3.connect(":memory:", detect_types=sqlite3.PARSE_DECLTYPES)
cur = con.cursor()
cur.execute("create table test (arr array)")
avec cette configuration, vous pouvez simplement insérer le tableau NumPy sans changement de syntaxe:
cur.execute("insert into test (arr) values (?)", (x, ))
et récupérez le tableau directement à partir de sqlite comme un tableau NumPy:
cur.execute("select arr from test")
data = cur.fetchone()[0]
print(data)
# [[ 0 1 2 3 4 5]
# [ 6 7 8 9 10 11]]
print(type(data))
# <type 'numpy.ndarray'>
Cela fonctionne pour moi:
import sqlite3 as sql
import numpy as np
import json
con = sql.connect('test.db',isolation_level=None)
cur = con.cursor()
cur.execute("DROP TABLE FOOBAR")
cur.execute("CREATE TABLE foobar (id INTEGER PRIMARY KEY, array BLOB)")
cur.execute("INSERT INTO foobar VALUES (?,?)", (None, json.dumps(np.arange(0,500,0.5).tolist())))
con.commit()
cur.execute("SELECT * FROM FOOBAR")
data = cur.fetchall()
print data
data = cur.fetchall()
my_list = json.loads(data[0][1])
Happy Leap Second est proche mais je continuais à obtenir un casting automatique à la corde. Aussi, si vous découvrez cet autre post: un débat amusant sur l'utilisation d'un tampon ou D'un binaire pour pousser des données non textuelles dans sqlite vous voyez que l'approche documentée est d'éviter le buffer tous ensemble et d'utiliser ce morceau de code.
def adapt_array(arr):
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read()
Je n'ai pas beaucoup testé cela en python 3, mais il semble fonctionner en python 2.7
je pense que matlab le format est un moyen très pratique pour stocker et récupérer des tableaux vides. Est vraiment rapide et empreinte disque et mémoire est tout à fait le même.
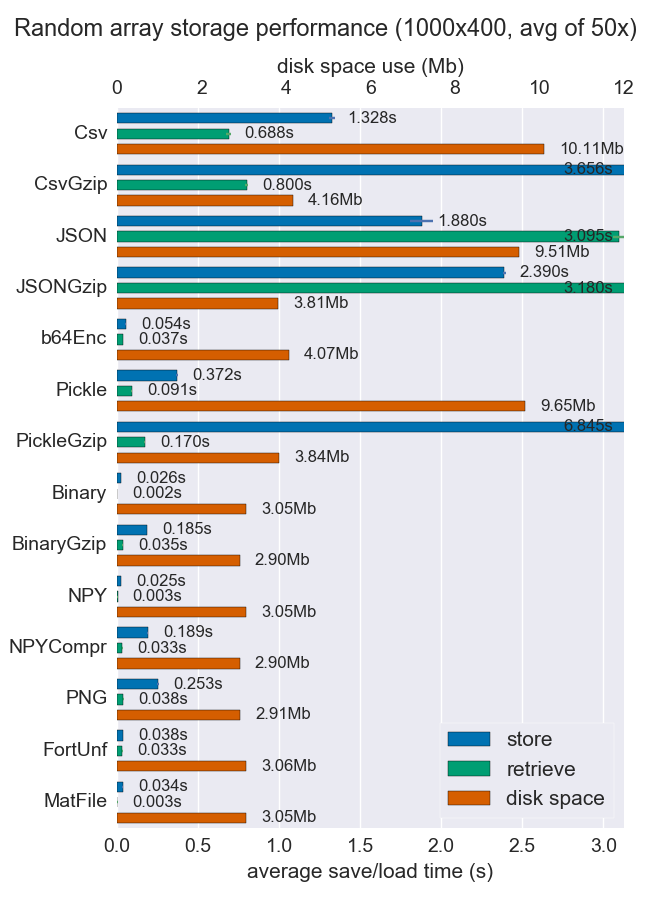
(image à partir d' mverleg repères)
mais si pour une raison quelconque vous avez besoin de stocker les tableaux numpy dans SQLite je suggère d'ajouter quelques capacités de compression.
Les lignes supplémentaires de unutbu le code est assez simple
compressor = 'zlib' # zlib, bz2
def adapt_array(arr):
"""
http://stackoverflow.com/a/31312102/190597 (SoulNibbler)
"""
# zlib uses similar disk size that Matlab v5 .mat files
# bz2 compress 4 times zlib, but storing process is 20 times slower.
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read().encode(compressor)) # zlib, bz2
def convert_array(text):
out = io.BytesIO(text)
out.seek(0)
out = io.BytesIO(out.read().decode(compressor))
return np.load(out)
Les résultats des tests avec base de données MNIST donne étaient les suivants:
$ ./test_MNIST.py
[69900]: 99% remain: 0 secs
Storing 70000 images in 379.9 secs
Retrieve 6990 images in 9.5 secs
$ ls -lh example.db
-rw-r--r-- 1 agp agp 69M sep 22 07:27 example.db
$ ls -lh mnist-original.mat
-rw-r--r-- 1 agp agp 53M sep 20 17:59 mnist-original.mat
```
en utilisant zlib et
$ ./test_MNIST.py
[69900]: 99% remain: 12 secs
Storing 70000 images in 8536.2 secs
Retrieve 6990 images in 37.4 secs
$ ls -lh example.db
-rw-r--r-- 1 agp agp 19M sep 22 03:33 example.db
$ ls -lh mnist-original.mat
-rw-r--r-- 1 agp agp 53M sep 20 17:59 mnist-original.mat
en utilisant bz2
comparer Matlab V5 format avec bz2 sur SQLite, la compression bz2 est d'environ 2,8, mais le temps d'accès est assez long par rapport au format Matlab (presque instantanément vs plus de 30 secondes). Peut-être n'est digne que pour les bases de données vraiment énormes où le le processus d'apprentissage est beaucoup plus long que le temps d'accès ou que l'empreinte de la base de données doit être aussi petite que possible.
Enfin, notez que bipz/zlib rapport est d'environ 3,7 et zlib/matlab nécessite 30% plus d'espace.
Le code complet si vous voulez jouer vous-même est:
import sqlite3
import numpy as np
import io
compressor = 'zlib' # zlib, bz2
def adapt_array(arr):
"""
http://stackoverflow.com/a/31312102/190597 (SoulNibbler)
"""
# zlib uses similar disk size that Matlab v5 .mat files
# bz2 compress 4 times zlib, but storing process is 20 times slower.
out = io.BytesIO()
np.save(out, arr)
out.seek(0)
return sqlite3.Binary(out.read().encode(compressor)) # zlib, bz2
def convert_array(text):
out = io.BytesIO(text)
out.seek(0)
out = io.BytesIO(out.read().decode(compressor))
return np.load(out)
sqlite3.register_adapter(np.ndarray, adapt_array)
sqlite3.register_converter("array", convert_array)
dbname = 'example.db'
def test_save_sqlite_arrays():
"Load MNIST database (70000 samples) and store in a compressed SQLite db"
os.path.exists(dbname) and os.unlink(dbname)
con = sqlite3.connect(dbname, detect_types=sqlite3.PARSE_DECLTYPES)
cur = con.cursor()
cur.execute("create table test (idx integer primary key, X array, y integer );")
mnist = fetch_mldata('MNIST original')
X, y = mnist.data, mnist.target
m = X.shape[0]
t0 = time.time()
for i, x in enumerate(X):
cur.execute("insert into test (idx, X, y) values (?,?,?)",
(i, y, int(y[i])))
if not i % 100 and i > 0:
elapsed = time.time() - t0
remain = float(m - i) / i * elapsed
print "\r[%5d]: %3d%% remain: %d secs" % (i, 100 * i / m, remain),
sys.stdout.flush()
con.commit()
con.close()
elapsed = time.time() - t0
print
print "Storing %d images in %0.1f secs" % (m, elapsed)
def test_load_sqlite_arrays():
"Query MNIST SQLite database and load some samples"
con = sqlite3.connect(dbname, detect_types=sqlite3.PARSE_DECLTYPES)
cur = con.cursor()
# select all images labeled as '2'
t0 = time.time()
cur.execute('select idx, X, y from test where y = 2')
data = cur.fetchall()
elapsed = time.time() - t0
print "Retrieve %d images in %0.1f secs" % (len(data), elapsed)
if __name__ == '__main__':
test_save_sqlite_arrays()
test_load_sqlite_arrays()