Mettez des étoiles sur les barplots ggplot et les boxplots-pour indiquer le niveau de signification (valeur p)
il est courant de mettre des étoiles sur des barplots ou des boxplots pour montrer le niveau de signification (valeur p) d'un ou entre deux groupes, voici quelques exemples:
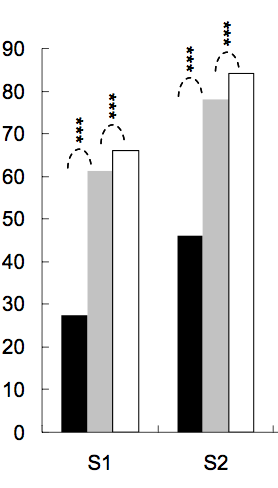
le nombre d'étoiles est défini par la valeur p, par exemple on peut mettre 3 étoiles pour la valeur p < 0,001, deux étoiles pour la valeur p < 0,01, et ainsi de suite (bien que cela change d'un article à l'autre).
et mes questions: comment générer similaire les graphiques? Les méthodes qui mettent automatiquement les étoiles en fonction du niveau de signification sont plus que bienvenues.
4 réponses
Veuillez trouver ma tentative ci-dessous.
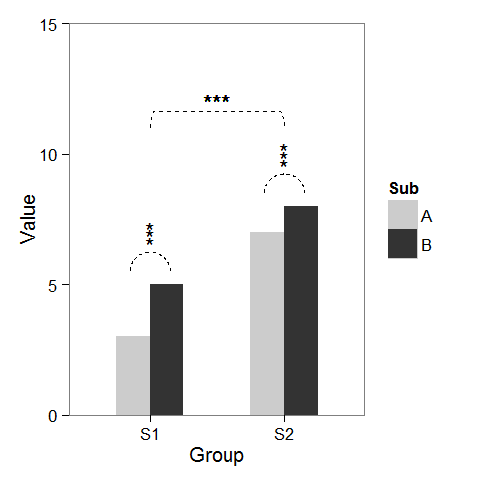
tout d'abord, j'ai créé quelques données factices et un plan à barres qui peut être modifié comme nous le souhaitons.
windows(4,4)
dat <- data.frame(Group = c("S1", "S1", "S2", "S2"),
Sub = c("A", "B", "A", "B"),
Value = c(3,5,7,8))
## Define base plot
p <-
ggplot(dat, aes(Group, Value)) +
theme_bw() + theme(panel.grid = element_blank()) +
coord_cartesian(ylim = c(0, 15)) +
scale_fill_manual(values = c("grey80", "grey20")) +
geom_bar(aes(fill = Sub), stat="identity", position="dodge", width=.5)
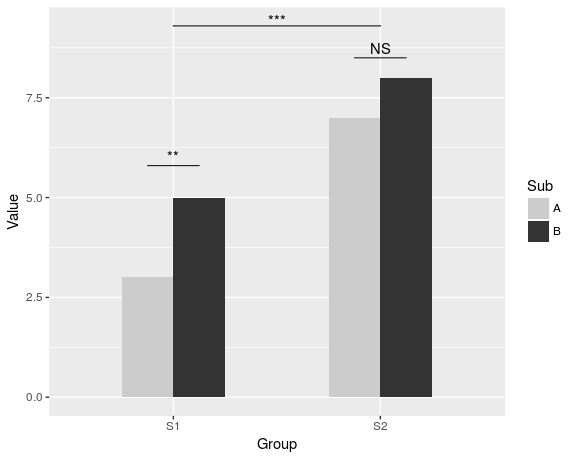
ajouter des astérisques au-dessus d'une colonne est facile, comme baptiste l'a déjà mentionné. Il suffit de créer un data.frame avec les coordonnées.
label.df <- data.frame(Group = c("S1", "S2"),
Value = c(6, 9))
p + geom_text(data = label.df, label = "***")
pour ajouter les arcs qui indiquent une comparaison de sous-groupe, j'ai calculé les coordonnées paramétriques d'un demi-cercle et je les ai ajoutés reliés avec geom_line. Astérisque besoin de nouvelles coordonnées, trop.
label.df <- data.frame(Group = c(1,1,1, 2,2,2),
Value = c(6.5,6.8,7.1, 9.5,9.8,10.1))
# Define arc coordinates
r <- 0.15
t <- seq(0, 180, by = 1) * pi / 180
x <- r * cos(t)
y <- r*5 * sin(t)
arc.df <- data.frame(Group = x, Value = y)
p2 <-
p + geom_text(data = label.df, label = "*") +
geom_line(data = arc.df, aes(Group+1, Value+5.5), lty = 2) +
geom_line(data = arc.df, aes(Group+2, Value+8.5), lty = 2)
Enfin, pour indiquer la comparaison entre les groupes, j'ai construit un cercle plus large et aplatie au sommet.
r <- .5
x <- r * cos(t)
y <- r*4 * sin(t)
y[20:162] <- y[20] # Flattens the arc
arc.df <- data.frame(Group = x, Value = y)
p2 + geom_line(data = arc.df, aes(Group+1.5, Value+11), lty = 2) +
geom_text(x = 1.5, y = 12, label = "***")
je sais que c'est une vieille question et la réponse de Jens Tierling fournit déjà une solution au problème. Mais j'ai récemment créé une extension ggplot qui simplifie tout le processus d'ajout de barres de signification: ggsignif
au lieu d'ajouter legeom_line et geom_text à votre parcelle vous ajoutez juste un calque simple geom_signif:
library(ggplot2)
library(ggsignif)
ggplot(iris, aes(x=Species, y=Sepal.Length)) +
geom_boxplot() +
geom_signif(comparisons = list(c("versicolor", "virginica")),
map_signif_level=TRUE)
pour créer un tracé plus avancé similaire à celui montré par Jens Tierling, vous pouvez faire:
dat <- data.frame(Group = c("S1", "S1", "S2", "S2"),
Sub = c("A", "B", "A", "B"),
Value = c(3,5,7,8))
ggplot(dat, aes(Group, Value)) +
geom_bar(aes(fill = Sub), stat="identity", position="dodge", width=.5) +
geom_signif(stat="identity",
data=data.frame(x=c(0.875, 1.875), xend=c(1.125, 2.125),
y=c(5.8, 8.5), annotation=c("**", "NS")),
aes(x=x,xend=xend, y=y, yend=y, annotation=annotation)) +
geom_signif(comparisons=list(c("S1", "S2")), annotations="***",
y_position = 9.3, tip_length = 0, vjust=0.4) +
scale_fill_manual(values = c("grey80", "grey20"))
la documentation complète du paquet est disponible à CRAN.
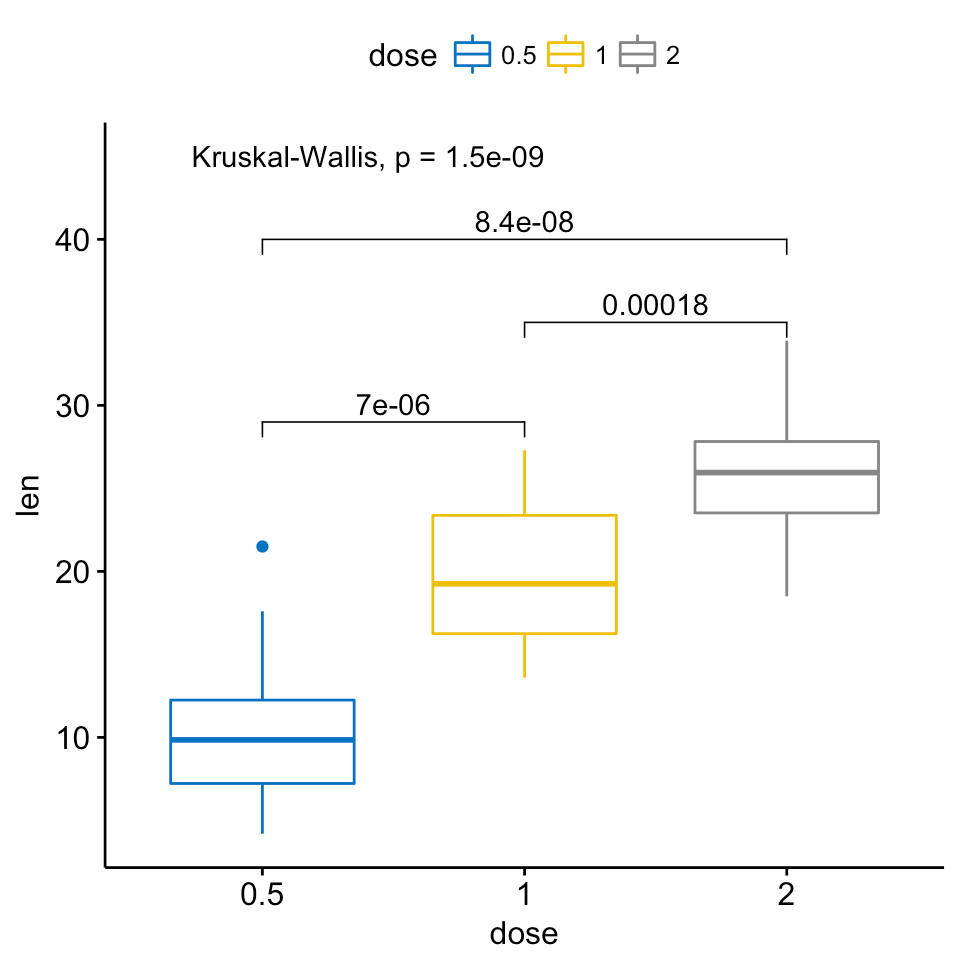
Il y a aussi une extension du ggsignif paquet ggpubr c'est plus puissant quand il s'agit de comparaisons multi-groupes. Il s'appuie sur ggsignif, mais aussi sur anova et kruskal-wallis ainsi que sur des comparaisons par paire avec la moyenne de gobal.
Exemple:
ggboxplot(ToothGrowth, x = "dose", y = "len",
color = "dose", palette = "jco")+
stat_compare_means(comparisons = my_comparisons, label.y = c(29, 35, 40))+
stat_compare_means(label.y = 45)
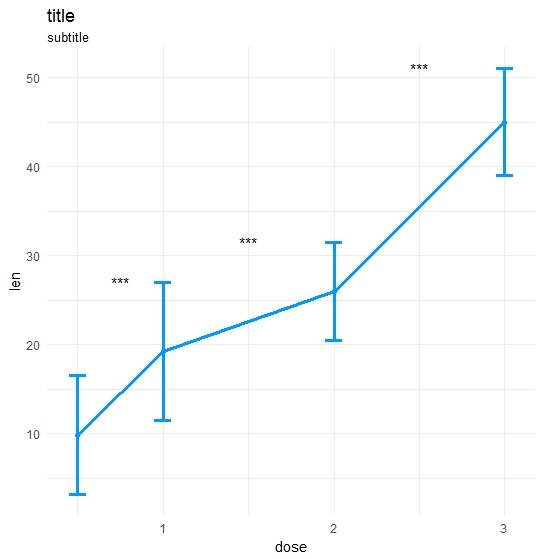
Fait ma propre fonction:
ts_test <- function(dataL,x,y,method="t.test",idCol=NULL,paired=F,label = "p.signif",p.adjust.method="none",alternative = c("two.sided", "less", "greater"),...) {
options(scipen = 999)
annoList <- list()
setDT(dataL)
if(paired) {
allSubs <- dataL[,.SD,.SDcols=idCol] %>% na.omit %>% unique
dataL <- dataL[,merge(.SD,allSubs,by=idCol,all=T),by=x] #idCol!!!
}
if(method =="t.test") {
dataA <- eval(parse(text=paste0(
"dataL[,.(",as.name(y),"=mean(get(y),na.rm=T),sd=sd(get(y),na.rm=T)),by=x] %>% setDF"
)))
res<-pairwise.t.test(x=dataL[[y]], g=dataL[[x]], p.adjust.method = p.adjust.method,
pool.sd = !paired, paired = paired,
alternative = alternative, ...)
}
if(method =="wilcox.test") {
dataA <- eval(parse(text=paste0(
"dataL[,.(",as.name(y),"=median(get(y),na.rm=T),sd=IQR(get(y),na.rm=T,type=6)),by=x] %>% setDF"
)))
res<-pairwise.wilcox.test(x=dataL[[y]], g=dataL[[x]], p.adjust.method = p.adjust.method,
paired = paired, ...)
}
#Output the groups
res$p.value %>% dimnames %>% {paste(.[[2]],.[[1]],sep="_")} %>% cat("Groups ",.)
#Make annotations ready
annoList[["label"]] <- res$p.value %>% diag %>% round(5)
if(!is.null(label)) {
if(label == "p.signif"){
annoList[["label"]] %<>% cut(.,breaks = c(-0.1, 0.0001, 0.001, 0.01, 0.05, 1),
labels = c("****", "***", "**", "*", "ns")) %>% as.character
}
}
annoList[["x"]] <- dataA[[x]] %>% {diff(.)/2 + .[-length(.)]}
annoList[["y"]] <- {dataA[[y]] + dataA[["sd"]]} %>% {pmax(lag(.), .)} %>% na.omit
#Make plot
coli="#0099ff";sizei=1.3
p <-ggplot(dataA, aes(x=get(x), y=get(y))) +
geom_errorbar(aes(ymin=len-sd, ymax=len+sd),width=.1,color=coli,size=sizei) +
geom_line(color=coli,size=sizei) + geom_point(color=coli,size=sizei) +
scale_color_brewer(palette="Paired") + theme_minimal() +
xlab(x) + ylab(y) + ggtitle("title","subtitle")
#Annotate significances
p <-p + annotate("text", x = annoList[["x"]], y = annoList[["y"]], label = annoList[["label"]])
return(p)
}
Données et de les appeler:
library(ggplot2);library(data.table);library(magrittr);
df_long <- rbind(ToothGrowth[,-2],data.frame(len=40:50,dose=3.0))
df_long$ID <- data.table::rowid(df_long$dose)
ts_test(dataL=df_long,x="dose",y="len",idCol="ID",method="wilcox.test",paired=T)