Création programmatique de tables Markdown dans R avec KnitR
Je commence tout juste à en apprendre davantage sur KnitR et l'utilisation de Markdown dans la génération de documents et de rapports R. Cela semble être parfait pour beaucoup de rapports quotidiens que j'ai à faire avec mon travail. Cependant, une chose que je ne vois pas est un moyen facile d'imprimer des cadres de données et des tables en utilisant le formatage Markdown (un peu comme xtable, mais avec Markdown au lieu de LaTeX ou HTML). Je sais que je peux simplement intégrer la sortie HTML de xtable, mais je me demandais s'il y avait un Markdown des solutions?
7 réponses
Maintenant knitr (depuis la version 1.3) le paquet comprend la fonction kable pour une création de tables:
> library(knitr)
> kable(head(iris[,1:3]), format = "markdown")
| Sepal.Length| Sepal.Width| Petal.Length|
|-------------:|------------:|-------------:|
| 5,1| 3,5| 1,4|
| 4,9| 3,0| 1,4|
| 4,7| 3,2| 1,3|
| 4,6| 3,1| 1,5|
| 5,0| 3,6| 1,4|
| 5,4| 3,9| 1,7|
Mise à jour : Si vous obtenez une démarque brute dans un document, essayez l'option de bloc setup results = "asis".
Deux paquets qui ne ce sont flatter
library(devtools)
install_github('pander', 'Rapporter')
Ou ascii
pander est une approche légèrement différente de la construction de rapports (mais peut être utile pour cette fonctionnalité).
ascii vous permettra de print avec type = 'pandoc (ou de divers autres démarque des saveurs)
library(ascii)
print(ascii(head(iris[,1:3])), type = 'pandoc')
**Sepal.Length** **Sepal.Width** **Petal.Length**
--- ------------------ ----------------- ------------------
1 5.10 3.50 1.40
2 4.90 3.00 1.40
3 4.70 3.20 1.30
4 4.60 3.10 1.50
5 5.00 3.60 1.40
6 5.40 3.90 1.70
--- ------------------ ----------------- ------------------
Notez que dans ces deux cas, il est dirigé vers l'utilisation de pandoc pour convertir de markdown au type de document souhaité, mais l'utilisation de style='rmarkdown' créera des tables qui sont compatibles avec ce paquet markdown et la conversion intégrée dans rstudio.
Je voulais juste mettre à jour ceci avec ce que j'ai décidé de faire. J'utilise le paquet hwriter en ce moment pour imprimer des tables, et j'utilise les fonctionnalités row.* et col.* pour mettre des classes CSS sur différents éléments. Ensuite, j'ai écrit CSS personnalisé pour faire mon affichage comme je le voulais. Donc, voici un exemple au cas où quelqu'un d'autre a affaire à quelque chose de similaire.
Tout d'abord, créez un fichier qui fera le knitting et modifiez le Markdown en HTML:
FILE: file_knit.r
#!/usr/bin/env Rscript
library(knitr)
library(markdown)
knit("file.Rmd")
markdownToHTML("file.md","file.html",stylesheet="~/custom.css")
Ensuite, créez la démarque réelle fichier:
FILE: file.Rmd
Report of Fruit vs. Animal Choices
==================================
This is a report of fruit vs. animal choices.
```{r echo=FALSE,results='asis'}
library(hwriter)
set.seed(9850104)
my.df <- data.frame(Var1=sample(x=c("Apple","Orange","Banana"),size=40,replace=TRUE),
Var2=sample(x=c("Dog","Cat","Bunny"),size=40,replace=TRUE))
tbl1 <- table(my.df$Var1,my.df$Var2)
tbl1 <- cbind(tbl1,rowSums(tbl1))
tbl1 <- rbind(tbl1,colSums(tbl1))
colnames(tbl1)[4] <- "TOTAL"
rownames(tbl1)[4] <- "TOTAL"
# Because I used results='asis' for this chunk, I can just use cat() and hwrite() to
# write out the table in HTML. Using hwrite()'s row.* function, I can assign classes
# to the various table elements.
cat(hwrite(tbl1,
border=NA,
table.class="t1",
row.class=list(c("header col_first","header col","header col","header col", "header col_last"),
c("col_first","col","col","col","col_last"),
c("col_first","col","col","col","col_last"),
c("col_first","col","col","col","col_last"),
c("footer col_first","footer col","footer col","footer col","footer col_last"))))
```
Enfin, il suffit de créer un fichier CSS personnalisé.
FILE: custom.css
body {
font-family: sans-serif;
background-color: white;
font-size: 12px;
margin: 20px;
}
h1 {font-size:1.5em;}
table {
border: solid;
border-color: black;
border-width: 2px;
border-collapse: collapse;
margin-bottom: 20px;
text-align: center;
padding: 0px;
}
.t1 .header {
color: white;
background-color: black;
border-bottom: solid;
border-color: black;
border-width: 2px;
font-weight: bold;
}
.t1 .footer {
border-top: solid;
border-color: black;
border-width: 2px;
}
.t1 .col_first {
border-right: solid;
border-color: black;
border-width: 2px;
text-align: left;
font-weight: bold;
width: 75px;
}
.t1 .col {
width: 50px;
}
.t1 .col_last {
width: 50px;
border-left: solid;
border-color: black;
border-width: 2px;
}
L'exécution de ./file_knit.r me donne un fichier.html, qui ressemble à ceci:
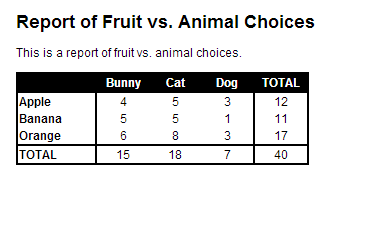
Donc, j'espère que cela pourrait être utile à d'autres qui veulent un peu plus de formatage dans la sortie Markdown!
Il y a des fonctions dans le paquet pander:
> library(pander)
> pandoc.table(head(iris)[, 1:3])
-------------------------------------------
Sepal.Length Sepal.Width Petal.Length
-------------- ------------- --------------
5.1 3.5 1.4
4.9 3 1.4
4.7 3.2 1.3
4.6 3.1 1.5
5 3.6 1.4
5.4 3.9 1.7
-------------------------------------------
Il n'est pas très difficile de faire votre propre fonction personnalisée. Voici une preuve de concept très simple pour générer une table rmarkdown d'un data.frame:
rmarkdownTable <- function(df){
cat(paste(names(df), collapse = "|"))
cat("\n")
cat(paste(rep("-", ncol(df)), collapse = "|"))
cat("\n")
for(i in 1:nrow(df)){
cat(paste(df[i,], collapse = "|"))
cat("\n")
}
invisible(NULL)
}
Dans .Document Rmd vous utiliseriez alors la fonction avec results = 'asis':
```{r, results = 'asis'}
rmarkdownTable <- function(df){
cat(paste(names(df), collapse = "|"))
cat("\n")
cat(paste(rep("-", ncol(df)), collapse = "|"))
cat("\n")
for(i in 1:nrow(df)){
cat(paste(df[i,], collapse = "|"))
cat("\n")
}
invisible(NULL)
}
rmarkdownTable(head(iris))
```
Le code ci-dessus vous donnerait la figure suivante (dans l'exemple, c'est la sortie pdf, mais puisque la table est dans markdwon, vous pouvez aussi tricoter en html ou word).
 De là - et en lisant le code d'autres peuples-vous pouvez comprendre comment manipulez le texte pour générer la table souhaitée et créer des fonctions plus personnalisées.
De là - et en lisant le code d'autres peuples-vous pouvez comprendre comment manipulez le texte pour générer la table souhaitée et créer des fonctions plus personnalisées.
Utilisez une combinaison de knitr:: kable et xtable dans votre document markdown.
library("knitr","xtable")
Pour une donnée simple.cadre -
kable(head(mtcars[,1:4]),format="markdown")
kable(head(mtcars[,1:4]),format="pandoc",caption="Title of the table")
format="pandoc" permet plus d'options comme légende.
Maintenant la combinaison pour résumé du modèle .
data(tli)
fm1 <- aov(tlimth ~ sex + ethnicty + grade + disadvg, data=tli)
kable(xtable(fm1), caption = "Annova table")
Pour encore plus d'options, regardez le paquet stargazer au lieu de xtable.
Pour écrire / créer Démarque des tables dans R, vous pouvez également utiliser MarkdownReports' MarkDown_Table_writer_DF_RowColNames() ou MarkDown_Table_writer_NamedVector() fonctions. Vous passez simplement une trame / matrice de données avec des noms de dimension, ou un vecteur avec des noms, et il analyse et écrit la table au format Markdown.