Mémoire alternative efficace à rbind en place rbind?
j'ai besoin de trouver deux grandes bases de données. En ce moment j'utilise
df <- rbind(df, df.extension)
mais j'ai (presque) instantanément perdu la mémoire. Je suppose que c'est parce que df est tenu dans la mémoire deux fois. Je pourrais voir des images encore plus grandes dans le futur, donc j'ai besoin d'une sorte de rbind en place.
alors ma question Est: y a-t-il un moyen d'éviter la duplication de données en mémoire lors de l'utilisation de rbind?
j'ai trouvé cette question , qui utilise SqlLite, mais je veux vraiment éviter d'utiliser le disque dur comme mémoire cache.
4 réponses
data.table est votre ami!
C. F. http://www.mail-archive.com/r-help@r-project.org/msg175877.html
suite au commentaire de nikola, voici la description de ?rbindlist (nouveau dans v1.8.2):
identique à
do.call("rbind",l), mais beaucoup plus rapide.
tout d'abord: utilisez la solution de l'autre question que vous liez si vous voulez être sûr. Comme R est call-by-value, oubliez une méthode "in-place" qui ne copie pas vos images de données dans la mémoire.
One not advisable méthode pour économiser un peu de mémoire, est de prétendre que vos images de données sont des listes, en exerçant une pression sur une liste en utilisant une boucle for (apply va manger la mémoire comme l'enfer) et faire croire Qu'il s'agit en fait d'une image de données.
je vous préviens encore une fois : utiliser ceci sur des images de données plus complexes demande des problèmes et des bugs difficiles à trouver. Assurez-vous donc de tester assez bien, et si possible, évitez autant que possible.
vous pouvez essayer l'approche suivante:
n1 <- 1000000
n2 <- 1000000
ncols <- 20
dtf1 <- as.data.frame(matrix(sample(n1*ncols), n1, ncols))
dtf2 <- as.data.frame(matrix(sample(n2*ncols), n1, ncols))
dtf <- list()
for(i in names(dtf1)){
dtf[[i]] <- c(dtf1[[i]],dtf2[[i]])
}
attr(dtf,"row.names") <- 1:(n1+n2)
attr(dtf,"class") <- "data.frame"
il efface les noms de lignes que vous avez réellement eu (vous pouvez les reconstruire, mais vérifiez les noms de lignes dupliqués!). Il n'effectue pas non plus tous les autres tests inclus dans rbind.
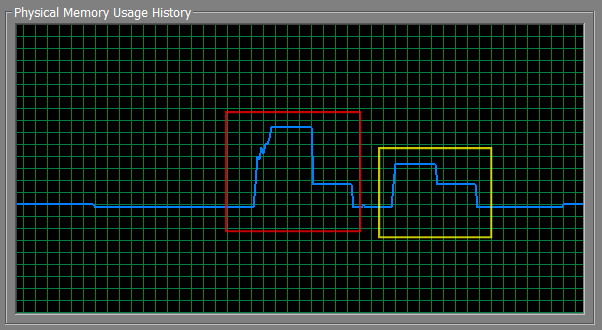
Vous sauve environ la moitié de la mémoire dans mes tests, et dans mon test à la fois le dtfcomb et le dtf sont égaux. La zone rouge est rbind, le jaune est ma liste.
script de Test :
n1 <- 3000000
n2 <- 3000000
ncols <- 20
dtf1 <- as.data.frame(matrix(sample(n1*ncols), n1, ncols))
dtf2 <- as.data.frame(matrix(sample(n2*ncols), n1, ncols))
gc()
Sys.sleep(10)
dtfcomb <- rbind(dtf1,dtf2)
Sys.sleep(10)
gc()
Sys.sleep(10)
rm(dtfcomb)
gc()
Sys.sleep(10)
dtf <- list()
for(i in names(dtf1)){
dtf[[i]] <- c(dtf1[[i]],dtf2[[i]])
}
attr(dtf,"row.names") <- 1:(n1+n2)
attr(dtf,"class") <- "data.frame"
Sys.sleep(10)
gc()
Sys.sleep(10)
rm(dtf)
gc()
en ce moment j'ai trouvé la solution suivante:
nextrow = nrow(df)+1
df[nextrow:(nextrow+nrow(df.extension)-1),] = df.extension
# we need to assure unique row names
row.names(df) = 1:nrow(df)
maintenant je ne manque pas de mémoire. Je pense que c'est parce que je stocke
object.size(df) + 2 * object.size(df.extension)
alors qu'avec rbind R aurait besoin
object.size(rbind(df,df.extension)) + object.size(df) + object.size(df.extension).
après ça j'utilise
rm(df.extension)
gc(reset=TRUE)
pour libérer la mémoire dont je n'ai plus besoin.
cela a résolu mon problème pour l'instant, mais je pense qu'il y a une façon plus avancée de faire une mémoire efficace rbind. J'apprécie tous les commentaires sur cette solution.
c'est un candidat parfait pour bigmemory . Voir sur le site pour plus d'informations. Voici trois aspects d'utilisation à considérer:
- il est correct d'utiliser la HD: la conversion de la mémoire à la HD est beaucoup plus rapide que pratiquement n'importe quel autre accès, donc vous ne pouvez pas voir des ralentissements. Parfois, je me fie à plus de 1 To de matrices mappées en mémoire, bien que la plupart soient entre 6 et 50 Go. En outre, comme l'objet est une matrice, ce ne nécessite pas de frais généraux réels de réécriture code afin d'utiliser l'objet.
- que vous utilisiez ou non une matrice à base de fichiers, vous pouvez utiliser
separated = TRUEpour séparer les colonnes. Je n'ai pas utilisé autant, à cause de mon 3ème conseil: - vous pouvez sur-affecter L'espace HD pour permettre une plus grande taille de matrice potentielle, mais seulement charger la sous-matrice d'intérêt. De cette façon, il n'y a pas besoin de faire
rbind.
Note: Bien que la question originale portait sur les bases de données et bigmemory est appropriée pour les matrices, on peut facilement créer des matrices différentes pour différents types de données et ensuite combiner les objets en RAM pour créer une base de données, si c'est vraiment nécessaire.