Matplotlib: éviter le chevauchement des points de données dans un graphique "scatter / dot/beeswarm"
quand je dessine un point en utilisant matplotlib, je voudrais décaler les points de données qui se chevauchent pour les garder tous visibles. Pour des exemples, si j'ai
CategoryA: 0,0,3,0,5
CategoryB: 5,10,5,5,10
je veux que chacun de l' CategoryA les points de données " 0 " doivent être placés côte à côte, plutôt qu'à droite les uns sur les autres, tout en restant distincts de CategoryB.
Dans R (ggplot2) il y a un "jitter" option qui fait cela. Est-il une option similaire dans matplotlib, ou est-il une autre approche qui permettrait d'aboutir à une résultat similaire?
Edit: pour préciser, "beeswarm" plot dans la R est essentiellement ce que j'ai à l'esprit, et pybeeswarm est un début précoce mais utile à une version matplotlib/Python.
Edit: ajouter que Seaborn Swarmplot, introduit dans la version 0.7, est une excellente implémentation de ce que je voulais.
6 réponses
prolongeant la réponse par @user2467675, voici comment je l'ai fait:
def rand_jitter(arr):
stdev = .01*(max(arr)-min(arr))
return arr + np.random.randn(len(arr)) * stdev
def jitter(x, y, s=20, c='b', marker='o', cmap=None, norm=None, vmin=None, vmax=None, alpha=None, linewidths=None, verts=None, hold=None, **kwargs):
return scatter(rand_jitter(x), rand_jitter(y), s=s, c=c, marker=marker, cmap=cmap, norm=norm, vmin=vmin, vmax=vmax, alpha=alpha, linewidths=linewidths, verts=verts, hold=hold, **kwargs)
stdev variable s'assure que le jitter est suffisant pour être vu sur différentes échelles, mais il suppose que les limites des axes sont 0 et la valeur max.
vous pouvez alors appeler jitter au lieu de scatter.
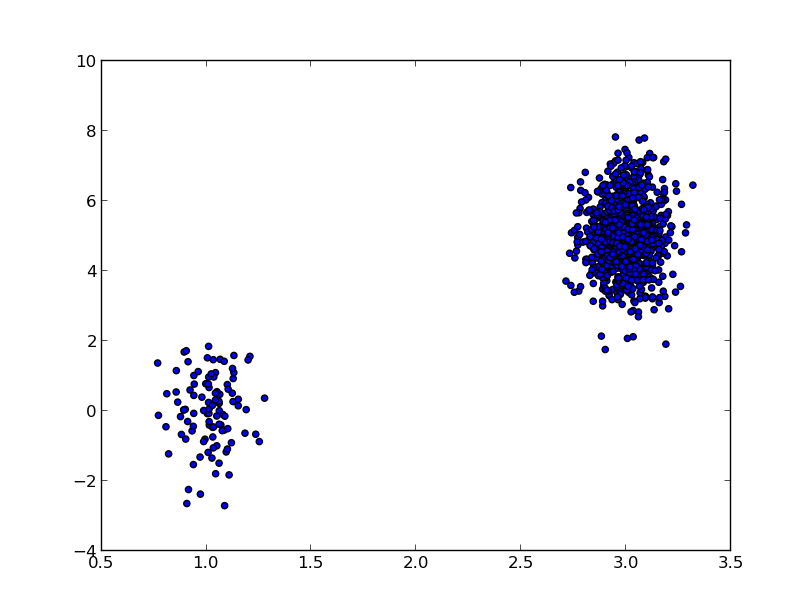
j'ai utilisé num PY.random à "disperser / beeswarm" les données le long de l'axe des X, mais autour d'un point fixe pour chaque catégorie, et puis essentiellement faire le tour.scatter() pour chaque catégorie:
import matplotlib.pyplot as plt
import numpy as np
#random data for category A, B, with B "taller"
yA, yB = np.random.randn(100), 5.0+np.random.randn(1000)
xA, xB = np.random.normal(1, 0.1, len(yA)),
np.random.normal(3, 0.1, len(yB))
plt.scatter(xA, yA)
plt.scatter(xB, yB)
plt.show()
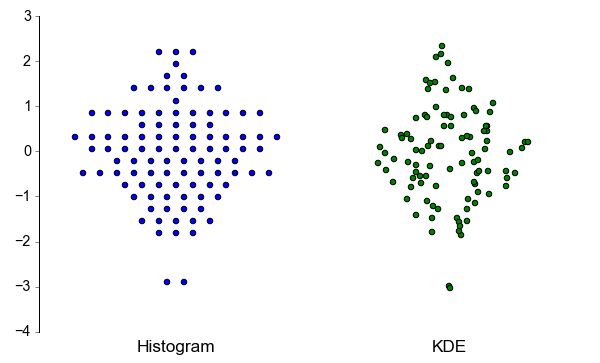
Une façon d'aborder le problème est de penser à chaque "ligne" dans votre scatter/dot/beeswarm intrigue comme une poubelle dans un histogramme:
data = np.random.randn(100)
width = 0.8 # the maximum width of each 'row' in the scatter plot
xpos = 0 # the centre position of the scatter plot in x
counts, edges = np.histogram(data, bins=20)
centres = (edges[:-1] + edges[1:]) / 2.
yvals = centres.repeat(counts)
max_offset = width / counts.max()
offsets = np.hstack((np.arange(cc) - 0.5 * (cc - 1)) for cc in counts)
xvals = xpos + (offsets * max_offset)
fig, ax = plt.subplots(1, 1)
ax.scatter(xvals, yvals, s=30, c='b')
cela implique évidemment de classer les données, donc vous pouvez perdre un peu de précision. Si vous avez des données discrètes, vous pouvez le remplacer:
counts, edges = np.histogram(data, bins=20)
centres = (edges[:-1] + edges[1:]) / 2.
avec:
centres, counts = np.unique(data, return_counts=True)
Une approche alternative qui préserve l'exacte des coordonnées y, même pour les données continues, est d'utiliser un estimation de la densité du grain pour dimensionner l'amplitude du jitter aléatoire dans l'axe des x:
from scipy.stats import gaussian_kde
kde = gaussian_kde(data)
density = kde(data) # estimate the local density at each datapoint
# generate some random jitter between 0 and 1
jitter = np.random.rand(*data.shape) - 0.5
# scale the jitter by the KDE estimate and add it to the centre x-coordinate
xvals = 1 + (density * jitter * width * 2)
ax.scatter(xvals, data, s=30, c='g')
for sp in ['top', 'bottom', 'right']:
ax.spines[sp].set_visible(False)
ax.tick_params(top=False, bottom=False, right=False)
ax.set_xticks([0, 1])
ax.set_xticklabels(['Histogram', 'KDE'], fontsize='x-large')
fig.tight_layout()
cette seconde méthode est vaguement basée sur la façon dont violon parcelles travail. Elle ne peut toujours pas garantir qu'aucun des points ne se chevauchent, mais je trouve qu'en pratique elle tend à donner des résultats assez beaux aussi longtemps qu'il y a un nombre décent de points (>20), et la distribution peut être raisonnablement bien approximée par une somme de gaussiens.
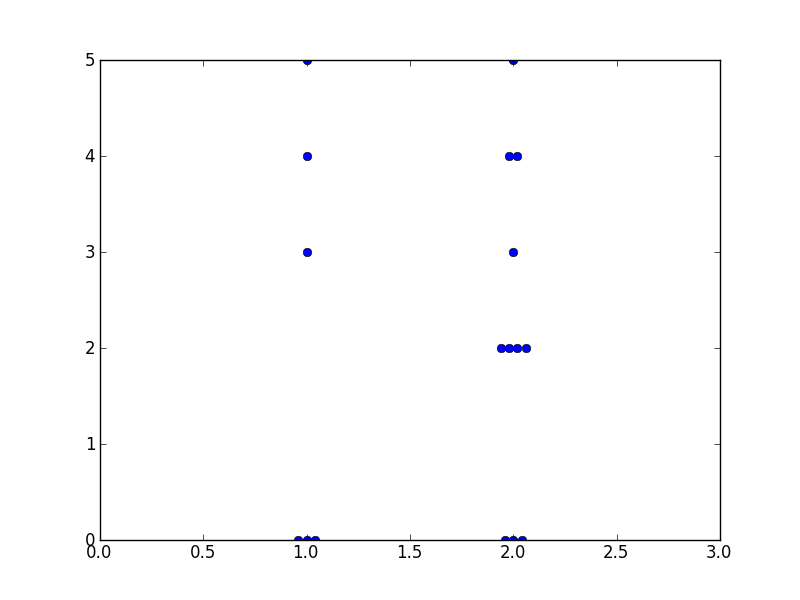
ne connaissant pas d'alternative directe au mpl, vous avez ici une proposition très rudimentaire:
from matplotlib import pyplot as plt
from itertools import groupby
CA = [0,4,0,3,0,5]
CB = [0,0,4,4,2,2,2,2,3,0,5]
x = []
y = []
for indx, klass in enumerate([CA, CB]):
klass = groupby(sorted(klass))
for item, objt in klass:
objt = list(objt)
points = len(objt)
pos = 1 + indx + (1 - points) / 50.
for item in objt:
x.append(pos)
y.append(item)
pos += 0.04
plt.plot(x, y, 'o')
plt.xlim((0,3))
plt.show()
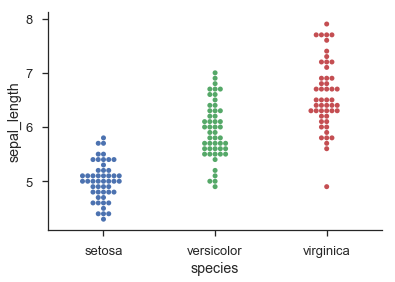
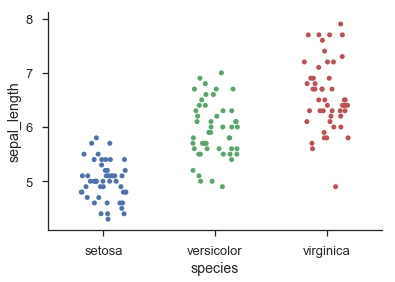
Seaborn fournit des tracés de points catégoriques ressemblant à des histogrammes par sns.swarmplot() et vacillant catégorique point de parcelles à l'aide de sns.stripplot():
import seaborn as sns
sns.set(style='ticks', context='talk')
iris = sns.load_dataset('iris')
sns.swarmplot('species', 'sepal_length', data=iris)
sns.despine()
sns.stripplot('species', 'sepal_length', data=iris, jitter=0.2)
sns.despine()
Le swarmplot de Seaborn semble être le plus approprié pour ce que vous avez en tête, mais vous pouvez aussi jitter avec le regplot de Seaborn:
import seaborn as sns
iris = sns.load_dataset('iris')
sns.regplot(x='sepal_length',
y='sepal_width',
data=iris,
fit_reg=False, # do not fit a regression line
x_jitter=0.1, # could also dynamically set this with range of data
y_jitter=0.1,
scatter_kws={'alpha': 0.5}) # set transparency to 50%