Existe-t-il une fonction d'autocorrélation numpy avec une sortie standardisée?
j'ai suivi le Conseil de définir la fonction d'autocorrélation dans un autre post:
def autocorr(x):
result = np.correlate(x, x, mode = 'full')
maxcorr = np.argmax(result)
#print 'maximum = ', result[maxcorr]
result = result / result[maxcorr] # <=== normalization
return result[result.size/2:]
cependant, la valeur maximale n'était pas "1,0". par conséquent, j'ai introduit la ligne marquée avec" < = = = normalisation "
j'ai essayé la fonction avec l'ensemble de données de" Time series analysis " (Box - Jenkins) chapitre 2. Je m'attendais à un résultat comme fig. 2.7 dans ce livre. Mais j'ai eu ce qui suit:
quelqu'un a une explication pour cet étrange comportement inattendu de l'autocorrélation?
Plus (2012-09-07):
je suis entré dans la programmation Python et j'ai fait ce qui suit:
from ClimateUtilities import *
import phys
#
# the above imports are from R.T.Pierrehumbert's book "principles of planetary
# climate"
# and the homepage of that book at "cambridge University press" ... they mostly
# define the
# class "Curve()" used in the below section which is not necessary in order to solve
# my
# numpy-problem ... :)
#
import numpy as np;
import scipy.spatial.distance;
# functions to be defined ... :
#
#
def autocorr(x):
result = np.correlate(x, x, mode = 'full')
maxcorr = np.argmax(result)
# print 'maximum = ', result[maxcorr]
result = result / result[maxcorr]
#
return result[result.size/2:]
##
# second try ... "Box and Jenkins" chapter 2.1 Autocorrelation Properties
# of stationary models
##
# from table 2.1 I get:
s1 = np.array([47,64,23,71,38,64,55,41,59,48,71,35,57,40,58,44,
80,55,37,74,51,57,50,60,45,57,50,45,25,59,50,71,56,74,50,58,45,
54,36,54,48,55,45,57,50,62,44,64,43,52,38,59,
55,41,53,49,34,35,54,45,68,38,50,
60,39,59,40,57,54,23],dtype=float);
# alternatively in order to test:
s2 = np.array([47,64,23,71,38,64,55,41,59,48,71])
##################################################################################3
# according to BJ, ch.2
###################################################################################3
print '*************************************************'
global s1short, meanshort, stdShort, s1dev, s1shX, s1shXk
s1short = s1
#s1short = s2 # for testing take s2
meanshort = s1short.mean()
stdShort = s1short.std()
s1dev = s1short - meanshort
#print 's1short = n', s1short, 'nmeanshort = ', meanshort, 'ns1deviation = n',
# s1dev,
# 'nstdShort = ', stdShort
s1sh_len = s1short.size
s1shX = np.arange(1,s1sh_len + 1)
#print 'Len = ', s1sh_len, 'nx-value = ', s1shX
##########################################################
# c0 to be computed ...
##########################################################
sumY = 0
kk = 1
for ii in s1shX:
#print 'ii-1 = ',ii-1,
if ii > s1sh_len:
break
sumY += s1dev[ii-1]*s1dev[ii-1]
#print 'sumY = ',sumY, 's1dev**2 = ', s1dev[ii-1]*s1dev[ii-1]
c0 = sumY / s1sh_len
print 'c0 = ', c0
##########################################################
# now compute autocorrelation
##########################################################
auCorr = []
s1shXk = s1shX
lenS1 = s1sh_len
nn = 1 # factor by which lenS1 should be divided in order
# to reduce computation length ... 1, 2, 3, 4
# should not exceed 4
#print 's1shX = ',s1shX
for kk in s1shXk:
sumY = 0
for ii in s1shX:
#print 'ii-1 = ',ii-1, ' kk = ', kk, 'kk+ii-1 = ', kk+ii-1
if ii >= s1sh_len or ii + kk - 1>=s1sh_len/nn:
break
sumY += s1dev[ii-1]*s1dev[ii+kk-1]
#print sumY, s1dev[ii-1], '*', s1dev[ii+kk-1]
auCorrElement = sumY / s1sh_len
auCorrElement = auCorrElement / c0
#print 'sum = ', sumY, ' element = ', auCorrElement
auCorr.append(auCorrElement)
#print '', auCorr
#
#manipulate s1shX
#
s1shX = s1shXk[:lenS1-kk]
#print 's1shX = ',s1shX
#print 'AutoCorr = n', auCorr
#########################################################
#
# first 15 of above Values are consistent with
# Box-Jenkins "Time Series Analysis", p.34 Table 2.2
#
#########################################################
s1sh_sdt = s1dev.std() # Standardabweichung short
#print 'ns1sh_std = ', s1sh_sdt
print '#########################################'
# "Curve()" is a class from RTP ClimateUtilities.py
c2 = Curve()
s1shXfloat = np.ndarray(shape=(1,lenS1),dtype=float)
s1shXfloat = s1shXk # to make floating point from integer
# might be not necessary
#print 'test plotting ... ', s1shXk, s1shXfloat
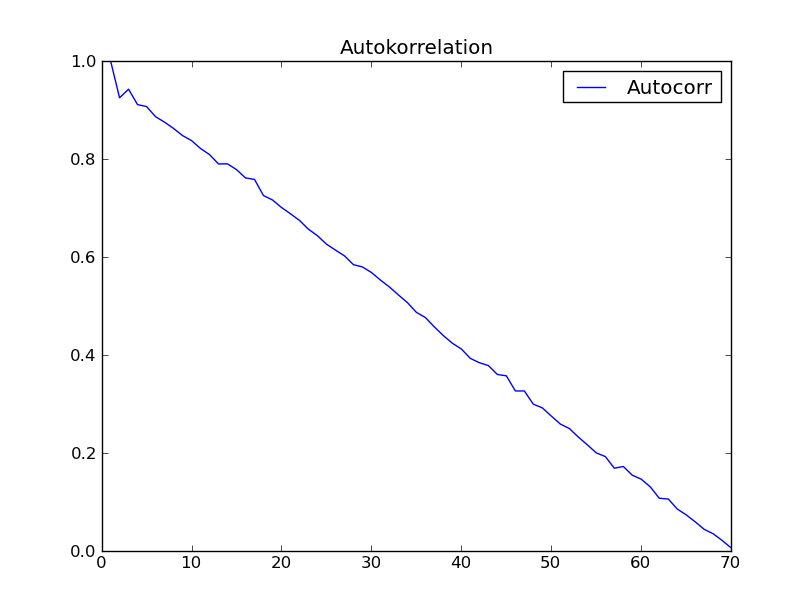
c2.addCurve(s1shXfloat)
c2.addCurve(auCorr, '', 'Autocorr')
c2.PlotTitle = 'Autokorrelation'
w2 = plot(c2)
##########################################################
#
# now try function "autocorr(arr)" and plot it
#
##########################################################
auCorr = autocorr(s1short)
c3 = Curve()
c3.addCurve( s1shXfloat )
c3.addCurve( auCorr, '', 'Autocorr' )
c3.PlotTitle = 'Autocorr with "autocorr"'
w3 = plot(c3)
#
# well that should it be!
#
2 réponses
donc votre problème avec votre tentative initiale est que vous n'avez pas soustrait la moyenne de votre signal. Le code suivant devrait fonctionner:
timeseries = (your data here)
mean = np.mean(timeseries)
timeseries -= np.mean(timeseries)
autocorr_f = np.correlate(timeseries, timeseries, mode='full')
temp = autocorr_f[autocorr_f.size/2:]/autocorr_f[autocorr_f.size/2]
iact.append(sum(autocorr_f[autocorr_f.size/2:]/autocorr_f[autocorr_f.size/2]))
Dans mon exemple temp est la variable qui vous intéressent; c'est l'avant intégrés fonction d'autocorrélation. Si vous voulez le temps d'autocorrélation intégré, vous êtes intéressé par iact .
Je ne sais pas quel est le problème.
l'autocorrélation d'un vecteur x doit être 1 à lag 0 puisque c'est juste la norme carrée L2 divisée par elle-même, i.e., dot(x, x) / dot(x, x) == 1 .
en général, pour tout lag i, j in Z, where i != j l'autocorrélation à l'échelle de l'unité est dot(shift(x, i), shift(x, j)) / dot(x, x) où shift(y, n) est une fonction qui déplace le vecteur y par n points de temps et Z est l'ensemble des entiers puisque nous sommes parler de la mise en œuvre (en théorie, les gal peuvent être en nombre réel).
je obtenir 1.0 comme le max avec le code suivant (départ sur la ligne de commande comme $ ipython --pylab ), comme prévu:
In[1]: n = 1000
In[2]: x = randn(n)
In[3]: xc = correlate(x, x, mode='full')
In[4]: xc /= xc[xc.argmax()]
In[5]: xchalf = xc[xc.size / 2:]
In[6]: xchalf_max = xchalf.max()
In[7]: print xchalf_max
Out[1]: 1.0
le seul moment où l'autocorrélation lag 0 n'est pas égale à 1 est lorsque x est le signal zéro (tous les zéros).
la réponse à votre question Est : Non , il n'y a pas de fonction NumPy qui effectue automatiquement la standardisation pour vous.
de plus, même si c'était le cas, vous auriez tout de même à le vérifier par rapport à votre résultat attendu, et si vous êtes en mesure de dire" oui, cela a effectué la standardisation correctement", alors je suppose que vous savez comment l'implémenter vous-même.
je vais suggérer que cela pourrait être le cas que vous avez mis en œuvre leur algorithme incorrectement, bien que je ne peux pas soyez sûr puisque je ne suis pas familier avec elle.