L'importation de plusieurs.fichiers csv dans R
supposons que nous ayons un dossier contenant plusieurs données.fichiers csv, contenant chacun le même nombre de variables mais provenant de périodes différentes. Est-il possible de R pour les importer simultanément, plutôt que d'avoir à les importer individuellement?
mon problème est que j'ai environ 2000 fichiers de données à importer et à devoir les importer individuellement juste en utilisant le code:
read.delim(file="filename", header=TRUE, sep="t")
n'est pas très efficace.
13 réponses
quelque chose comme ce qui suit devrait résulter dans chaque base de données comme un élément distinct dans une liste unique:
temp = list.files(pattern="*.csv")
myfiles = lapply(temp, read.delim)
cela suppose que vous avez ces CSVs dans un seul répertoire--votre répertoire courant de travail--et que tous ont l'extension minuscule .csv .
si vous voulez ensuite combiner ces bases de données en une seule base de données, voir les solutions dans d'autres réponses en utilisant des choses comme do.call(rbind,...) , dplyr::bind_rows() ou data.table::rbindlist() .
si vous voulez vraiment chaque base de données dans un objet séparé, même si c'est souvent déconseillé, vous pouvez faire ce qui suit avec assign :
temp = list.files(pattern="*.csv")
for (i in 1:length(temp)) assign(temp[i], read.csv(temp[i]))
Ou sans assign , et à démontrer (1) comment le nom de fichier peut être nettoyé et (2) montrent comment utiliser list2env , vous pouvez essayer ce qui suit:
temp = list.files(pattern="*.csv")
list2env(
lapply(setNames(temp, make.names(gsub("*.csv$", "", temp))),
read.csv), envir = .GlobalEnv)
Mais encore une fois, il est souvent préférable de les laisser dans un seul liste.
Voici une autre option pour convertir le .les fichiers csv en une seule donnée.cadre. En utilisant les fonctions de base R. C'est un ordre de grandeur plus lent que les options ci-dessous.
# Get the files names
files = list.files(pattern="*.csv")
# First apply read.csv, then rbind
myfiles = do.call(rbind, lapply(files, function(x) read.csv(x, stringsAsFactors = FALSE)))
Edit: - quelques choix supplémentaires en utilisant data.table et readr
Un fread() version, qui est une fonction de la data.table . Ce devrait être l'option la plus rapide.
library(data.table)
DT = do.call(rbind, lapply(files, fread)
# the same using `rbindlist()`
DT = rbindlist(lapply(files, fread))
à l'Aide de readr , qui est un nouveau paquet hadley pour lire des fichiers csv. Un peu plus lent que fread, mais avec des fonctionnalités différentes.
library(readr)
library(dplyr)
tbl = lapply(files, read_csv) %>% bind_rows()
une solution rapide et succincte tidyverse :
(plus de deux fois plus rapide que Base R read.csv )
tbl <-
list.files(pattern = "*.csv") %>%
map_df(~read_csv(.))
"1519200920 des données".tableau 's fread() peut même couper ces temps de charge en deux.
library(data.table)
tbl_fread <-
list.files(pattern = "*.csv") %>%
map_df(~fread(., stringsAsFactors = FALSE))
l'argument stringsAsFactors = FALSE maintient le facteur dataframe libre.
si la typographie est cheeky, vous pouvez forcer toutes les colonnes à être des caractères avec l'argument col_types .
tbl <-
list.files(pattern = "*.csv") %>%
map_df(~read_csv(., col_types = cols(.default = "c")))
si vous voulez aller dans des sous-répertoires pour construire votre liste de fichiers pour éventuellement lier, alors assurez-vous d'inclure le nom du chemin, ainsi que d'enregistrer les fichiers avec leurs noms complets dans votre liste. Cela permettra au travail de liaison de continuer en dehors du répertoire courant. (En pensant que les noms de chemins complets fonctionnent comme des passeports pour permettre le mouvement retour à travers le répertoire "frontières".)
tbl <-
list.files(path = "./subdirectory/",
pattern = "*.csv",
full.names = T) %>%
map_df(~read_csv(., col_types = cols(.default = "c")))
comme Hadley décrit ici (environ à mi-chemin):
map_df(x, f)est effectivement identique àdo.call("rbind", lapply(x, f))....
Bonus Feature - ajout de noms de fichiers à la demande d'enregistrement par Niks feature dans les commentaires ci-dessous:
* Ajout d'origine filename à chaque enregistrement.
code expliqué: créer une fonction pour ajouter le nom de fichier à chaque enregistrement lors de la lecture initiale des tables. Puis utilisez cette fonction à la place de la simple fonction read_csv() .
read_plus <- function(flnm) {
read_csv(flnm) %>%
mutate(filename = flnm)
}
tbl_with_sources <-
list.files(pattern = "*.csv",
full.names = T) %>%
map_df(~read_plus(.))
(les approches de la typographie et de la manipulation des sous-répertoires peuvent également être traitées à l'intérieur de la fonction read_plus() de la même manière qu'illustrées dans les deuxième et troisième variantes suggérées ci-dessus.)
### Benchmark Code & Results
library(tidyverse)
library(data.table)
library(microbenchmark)
### Base R Approaches
#### Instead of a dataframe, this approach creates a list of lists
#### removed from analysis as this alone doubled analysis time reqd
# lapply_read.delim <- function(path, pattern = "*.csv") {
# temp = list.files(path, pattern, full.names = TRUE)
# myfiles = lapply(temp, read.delim)
# }
#### `read.csv()`
do.call_rbind_read.csv <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
do.call(rbind, lapply(files, function(x) read.csv(x, stringsAsFactors = FALSE)))
}
map_df_read.csv <- function(path, pattern = "*.csv") {
list.files(path, pattern, full.names = TRUE) %>%
map_df(~read.csv(., stringsAsFactors = FALSE))
}
### *dplyr()*
#### `read_csv()`
lapply_read_csv_bind_rows <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
lapply(files, read_csv) %>% bind_rows()
}
map_df_read_csv <- function(path, pattern = "*.csv") {
list.files(path, pattern, full.names = TRUE) %>%
map_df(~read_csv(., col_types = cols(.default = "c")))
}
### *data.table* / *purrr* hybrid
map_df_fread <- function(path, pattern = "*.csv") {
list.files(path, pattern, full.names = TRUE) %>%
map_df(~fread(., stringsAsFactors = FALSE))
}
### *data.table*
rbindlist_fread <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
rbindlist(lapply(files, function(x) fread(x, stringsAsFactors = FALSE)))
}
do.call_rbind_fread <- function(path, pattern = "*.csv") {
files = list.files(path, pattern, full.names = TRUE)
do.call(rbind, lapply(files, function(x) fread(x, stringsAsFactors = FALSE)))
}
read_results <- function(dir_size){
microbenchmark(
# lapply_read.delim = lapply_read.delim(dir_size), # too slow to include in benchmarks
do.call_rbind_read.csv = do.call_rbind_read.csv(dir_size),
map_df_read.csv = map_df_read.csv(dir_size),
lapply_read_csv_bind_rows = lapply_read_csv_bind_rows(dir_size),
map_df_read_csv = map_df_read_csv(dir_size),
rbindlist_fread = rbindlist_fread(dir_size),
do.call_rbind_fread = do.call_rbind_fread(dir_size),
map_df_fread = map_df_fread(dir_size),
times = 10L)
}
read_results_lrg_mid_mid <- read_results('./testFolder/500MB_12.5MB_40files')
print(read_results_lrg_mid_mid, digits = 3)
read_results_sml_mic_mny <- read_results('./testFolder/5MB_5KB_1000files/')
read_results_sml_tny_mod <- read_results('./testFolder/5MB_50KB_100files/')
read_results_sml_sml_few <- read_results('./testFolder/5MB_500KB_10files/')
read_results_med_sml_mny <- read_results('./testFolder/50MB_5OKB_1000files')
read_results_med_sml_mod <- read_results('./testFolder/50MB_5OOKB_100files')
read_results_med_med_few <- read_results('./testFolder/50MB_5MB_10files')
read_results_lrg_sml_mny <- read_results('./testFolder/500MB_500KB_1000files')
read_results_lrg_med_mod <- read_results('./testFolder/500MB_5MB_100files')
read_results_lrg_lrg_few <- read_results('./testFolder/500MB_50MB_10files')
read_results_xlg_lrg_mod <- read_results('./testFolder/5000MB_50MB_100files')
print(read_results_sml_mic_mny, digits = 3)
print(read_results_sml_tny_mod, digits = 3)
print(read_results_sml_sml_few, digits = 3)
print(read_results_med_sml_mny, digits = 3)
print(read_results_med_sml_mod, digits = 3)
print(read_results_med_med_few, digits = 3)
print(read_results_lrg_sml_mny, digits = 3)
print(read_results_lrg_med_mod, digits = 3)
print(read_results_lrg_lrg_few, digits = 3)
print(read_results_xlg_lrg_mod, digits = 3)
# display boxplot of my typical use case results & basic machine max load
par(oma = c(0,0,0,0)) # remove overall margins if present
par(mfcol = c(1,1)) # remove grid if present
par(mar = c(12,5,1,1) + 0.1) # to display just a single boxplot with its complete labels
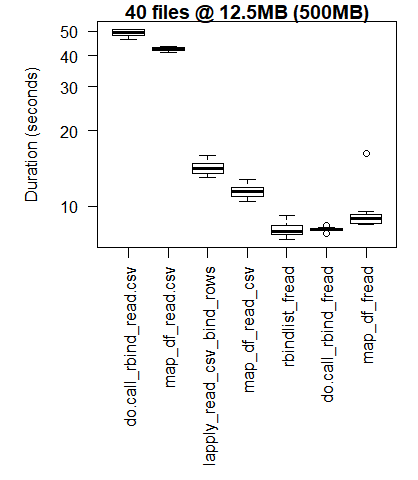
boxplot(read_results_lrg_mid_mid, las = 2, xlab = "", ylab = "Duration (seconds)", main = "40 files @ 12.5MB (500MB)")
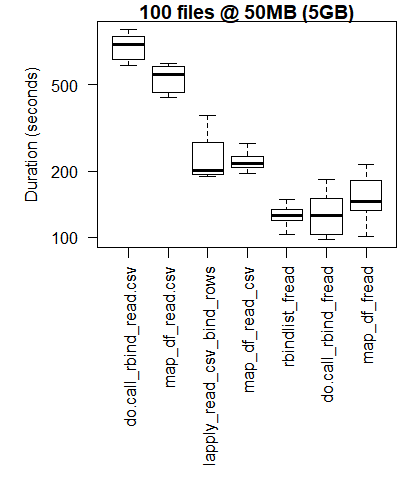
boxplot(read_results_xlg_lrg_mod, las = 2, xlab = "", ylab = "Duration (seconds)", main = "100 files @ 50MB (5GB)")
# generate 3x3 grid boxplots
par(oma = c(12,1,1,1)) # margins for the whole 3 x 3 grid plot
par(mfcol = c(3,3)) # create grid (filling down each column)
par(mar = c(1,4,2,1)) # margins for the individual plots in 3 x 3 grid
boxplot(read_results_sml_mic_mny, las = 2, xlab = "", ylab = "Duration (seconds)", main = "1000 files @ 5KB (5MB)", xaxt = 'n')
boxplot(read_results_sml_tny_mod, las = 2, xlab = "", ylab = "Duration (milliseconds)", main = "100 files @ 50KB (5MB)", xaxt = 'n')
boxplot(read_results_sml_sml_few, las = 2, xlab = "", ylab = "Duration (milliseconds)", main = "10 files @ 500KB (5MB)",)
boxplot(read_results_med_sml_mny, las = 2, xlab = "", ylab = "Duration (microseconds) ", main = "1000 files @ 50KB (50MB)", xaxt = 'n')
boxplot(read_results_med_sml_mod, las = 2, xlab = "", ylab = "Duration (microseconds)", main = "100 files @ 500KB (50MB)", xaxt = 'n')
boxplot(read_results_med_med_few, las = 2, xlab = "", ylab = "Duration (seconds)", main = "10 files @ 5MB (50MB)")
boxplot(read_results_lrg_sml_mny, las = 2, xlab = "", ylab = "Duration (seconds)", main = "1000 files @ 500KB (500MB)", xaxt = 'n')
boxplot(read_results_lrg_med_mod, las = 2, xlab = "", ylab = "Duration (seconds)", main = "100 files @ 5MB (500MB)", xaxt = 'n')
boxplot(read_results_lrg_lrg_few, las = 2, xlab = "", ylab = "Duration (seconds)", main = "10 files @ 50MB (500MB)")
Milieu Cas D'Utilisation
Large Use Case
Variété de Cas d'Utilisation
Lignes : nombre de fichiers(1000, 100, 10)
Colonnes: taille finale de la base de données (5 Mo, 50 Mo, 500 Mo)
(cliquez sur l'image pour voir la taille originale)
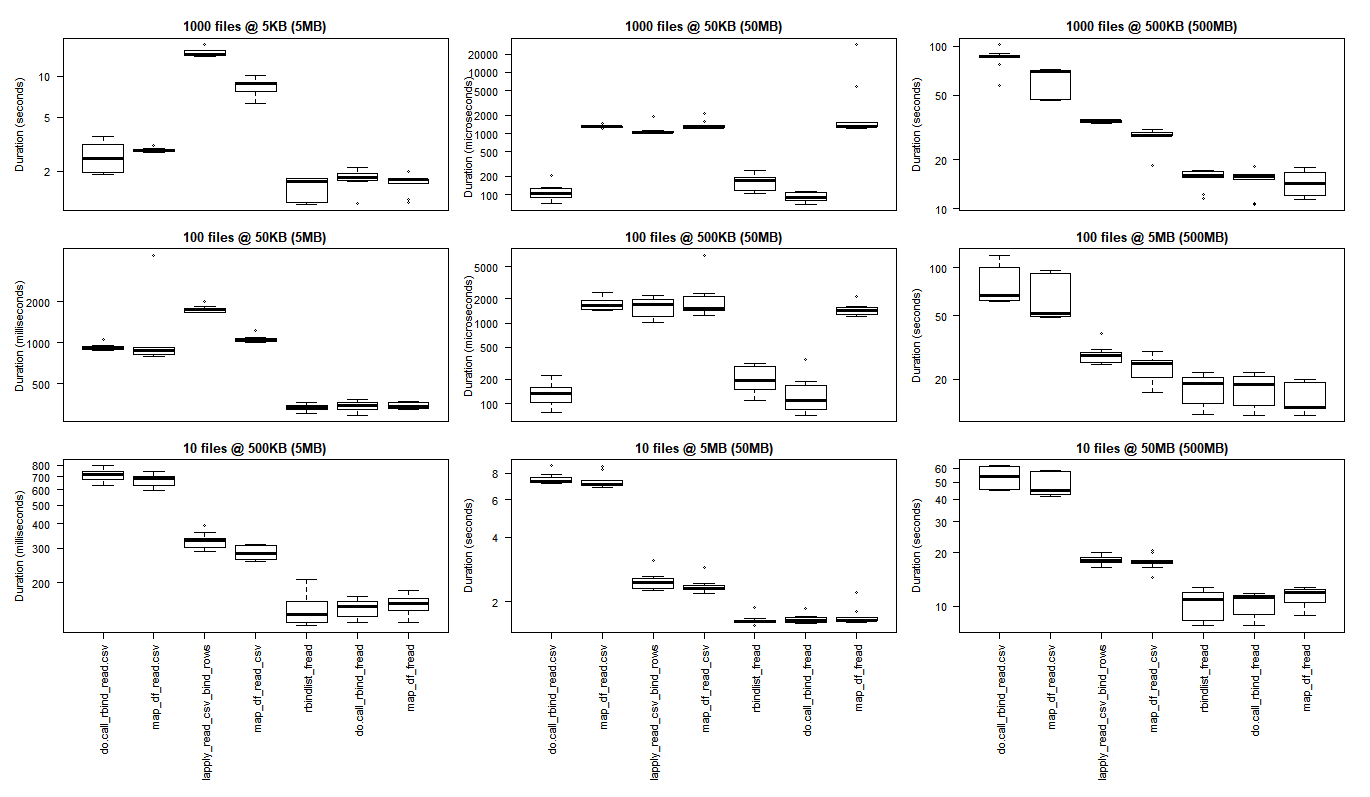
les résultats de base R sont meilleurs pour les cas d'utilisation les plus petits où les frais généraux de mise à contribution des bibliothèques C de purrr et dplyr dépassent les gains de performance observés lors de l'exécution de tâches de traitement à plus grande échelle.
si vous voulez exécuter vos propres tests, vous pouvez trouver ce script bash utile.
for ((i=1; i<=; i++)); do
cp "" "${1:0:8}_${i}.csv";
done
bash what_you_name_this_script.sh "fileName_you_want_copied" 100 créera 100 copies de votre fichier numérotées séquentiellement (après les 8 caractères initiaux du nom du fichier et un caractère de soulignement).
Attributions et appréciations
avec un remerciement spécial à:
- Tyler Rinker et Akrun for demonstrating microbenchmark.
- Jake Kaupp pour m'avoir présenté à
map_df()ici . - David McLaughlin pour des commentaires utiles sur l'amélioration des visualisations et discuter/confirmer les inversions de performance observées dans le petit fichier, résultats d'analyse de base de données.
ainsi qu'en utilisant lapply ou une autre construction en boucle dans R vous pourriez fusionner vos fichiers CSV dans un fichier.
dans Unix, si les fichiers n'ont pas de headers, alors c'est aussi simple que:
cat *.csv > all.csv
ou s'il y a des en-têtes, et que vous pouvez trouver une chaîne qui correspond aux en-têtes et seulement aux en-têtes (supposons que les lignes d'en-tête commencent toutes par "âge"), vous feriez:
cat *.csv | grep -v ^Age > all.csv
je pense que dans Windows vous pouvez faire cela avec COPY et SEARCH (ou FIND ou quelque chose) à partir de la commande DOS de la boîte, mais pourquoi ne pas installer cygwin et bénéficiez de la puissance de la commande Unix shell?
c'est le code que j'ai développé pour lire tous les fichiers csv dans R. Il va créer un datagramme pour chaque fichier csv individuellement et le titre qui datagramme le nom original du fichier (en supprimant les espaces et le .csv) j'espère qu'elle vous sera utile!
path <- "C:/Users/cfees/My Box Files/Fitness/"
files <- list.files(path=path, pattern="*.csv")
for(file in files)
{
perpos <- which(strsplit(file, "")[[1]]==".")
assign(
gsub(" ","",substr(file, 1, perpos-1)),
read.csv(paste(path,file,sep="")))
}
en utilisant plyr::ldply il y a une augmentation de vitesse d'environ 50% en activant l'option .parallel tout en lisant 400 fichiers csv d'environ 30-40 MB chacun. L'exemple inclut une barre de progression de texte.
library(plyr)
library(data.table)
library(doSNOW)
csv.list <- list.files(path="t:/data", pattern=".csv$", full.names=TRUE)
cl <- makeCluster(4)
registerDoSNOW(cl)
pb <- txtProgressBar(max=length(csv.list), style=3)
pbu <- function(i) setTxtProgressBar(pb, i)
dt <- setDT(ldply(csv.list, fread, .parallel=TRUE, .paropts=list(.options.snow=list(progress=pbu))))
stopCluster(cl)
à mon avis, la plupart des autres réponses sont obsolètes par rio::import_list , qui est un résumé:
library(rio)
my_data <- import_list(dir("path_to_directory", pattern = ".csv", rbind = TRUE))
tous les arguments supplémentaires sont passés à rio::import . rio peut traiter avec presque n'importe quel format de fichier R peut lire, et il utilise data.table 's fread si possible, donc il devrait être rapide aussi.
en S'appuyant sur le commentaire de dnlbrk, assign peut être considérablement plus rapide que list2env pour les gros fichiers.
library(readr)
library(stringr)
List_of_file_paths <- list.files(path ="C:/Users/Anon/Documents/Folder_with_csv_files/", pattern = ".csv", all.files = TRUE, full.names = TRUE)
en réglant le plein.argument de noms à true, vous obtiendrez le chemin complet de chaque dossier comme une chaîne de caractères séparée dans votre liste de dossiers, par exemple, List_of_file_paths[1] sera quelque chose comme "C:/Users/Anon/Documents/Folder_with_csv_files/file1.csv "
for(f in 1:length(List_of_filepaths)) {
file_name <- str_sub(string = List_of_filepaths[f], start = 46, end = -5)
file_df <- read_csv(List_of_filepaths[f])
assign( x = file_name, value = file_df, envir = .GlobalEnv)
}
, Vous pouvez utiliser les données.table package's fread ou base R lire.csv au lieu de read_csv. L'étape file_name vous permet de ranger le nom de sorte que chaque base de données ne reste pas avec le chemin complet vers le fichier comme son nom. Vous pouvez étendre votre boucle pour faire d'autres choses à la table de données avant de la transférer à l'environnement global, par exemple:
for(f in 1:length(List_of_filepaths)) {
file_name <- str_sub(string = List_of_filepaths[f], start = 46, end = -5)
file_df <- read_csv(List_of_filepaths[f])
file_df <- file_df[,1:3] #if you only need the first three columns
assign( x = file_name, value = file_df, envir = .GlobalEnv)
}
ma fourchette de la réponse acceptée est un peu plus rapide et supprime .csv du nom de l'objet dans R.
temp = list.files(pattern="*.csv")
for (i in 1:length(temp)) assign(gsub(".csv", "", temp[i]), read_csv(temp[i]))
elle s'appuie sur le paquet readr (i.e., library(readr) ). Vous pouvez faire quelque chose de similaire avec data.table si vous le souhaitez.
j'utilise ceci avec succès:
xlist<-list.files(pattern = "*.csv")
for(i in xlist) {
x <- read.csv((i))
assign(i, x)
}
si vous voulez collecter différents fichiers csv dans une seule donnée.châssis, vous pouvez utiliser ce qui suit. notez que les données "x".cadre devrait être créé à l'avance.
temp <- list.files(pattern="*.csv")
for (i in 1:length(temp)) {
temp2 = read.csv(temp[i], header = TRUE)
x <- rbind(x,temp2)
}
vous pouvez utiliser le superbe paquet sparklyr pour ceci:
# RStudio will help you get set-up with the Spark dependencies
library(sparklyr)
library(dplyr)
sc <- spark_connect(master = "local", version = "2.0.2")
df <- spark_read_csv(sc,
"dummy",
"file:////Users/bob/dev/data/results/*/*/*-metrics.csv") %>%
collect()
C'est une partie de mon script.
#This cycle read the files in a directory and assign the filenames to datasets
files <- list.files(pattern=".csv$")
for(i in files) {
X <- read.table(i, header=TRUE)
SN<-X$A/X$B
X<-cbind(X,SN)
ds<-paste("data_",i, sep="")#this add "data_" to the name of file
ds<-substr(ds, 1, nchar(ds)-4)#remove the last 4 char (.csv)
assign(ds, X)
}