Comment profiler plusieurs sous-processus en utilisant Python multiprocessing et profileur de mémoire?
j'ai un utilitaire qui génère plusieurs travailleurs à l'aide de Python multiprocessing et j'aimerais pouvoir suivre leur utilisation de la mémoire via l'excellentmemory_profiler l'utilité, qui fait tout ce que je veux - en particulier l'échantillonnage de l'utilisation de la mémoire au fil du temps et le tracé du résultat final (je ne suis pas concerné par le profilage de la mémoire ligne par ligne pour cette question).
afin de configurer cette question, j'ai créé une version plus simple du script, qui a une fonction worker qui attribue une mémoire similaire à la exemple donné memory_profiler bibliothèque. Le travailleur est comme suit:
import time
X6 = 10 ** 6
X7 = 10 ** 7
def worker(num, wait, amt=X6):
"""
A function that allocates memory over time.
"""
frame = []
for idx in range(num):
frame.extend([1] * amt)
time.sleep(wait)
del frame
compte tenu d'une charge de travail séquentielle de 4 travailleurs comme suit:
if __name__ == '__main__':
worker(5, 5, X6)
worker(5, 2, X7)
worker(5, 5, X6)
worker(5, 2, X7)
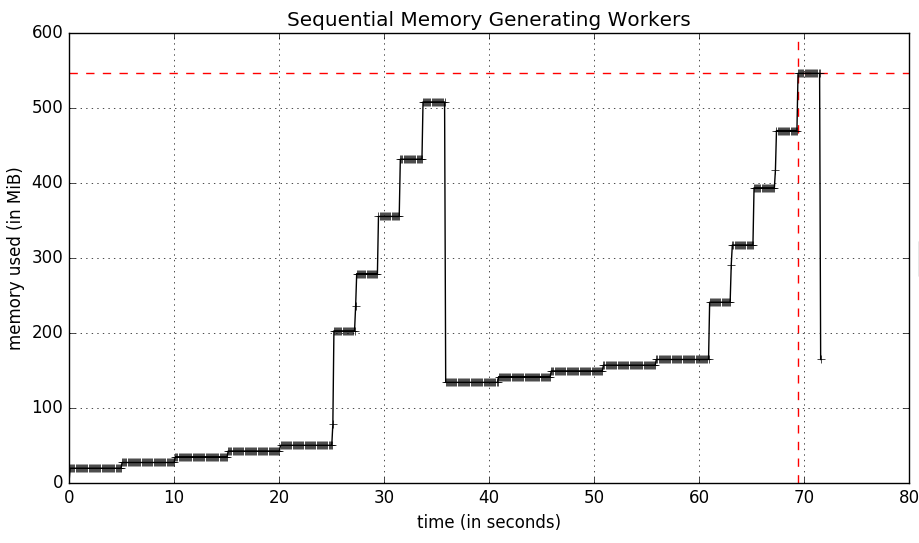
Exécution mprof exécutable pour profiler mon script prend 70 secondes pour que chaque travailleur exécute l'un après l'autre. Le script, exécutez comme suit:
$ mprof run python myscript.py
produit la mémoire suivante graphique:
que ces travailleurs aillent en parallèle avec multiprocessing signifie que le script finira aussi lentement que l'opérateur le plus lent (25 secondes). Ce script est comme suit:
import multiprocessing as mp
if __name__ == '__main__':
pool = mp.Pool(processes=4)
tasks = [
pool.apply_async(worker, args) for args in
[(5, 5, X6), (5, 2, X7), (5, 5, X6), (5, 2, X7)]
]
results = [p.get() for p in tasks]
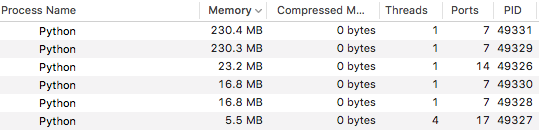
le profileur de mémoire fonctionne en effet, ou du moins il n'y a pas d'erreurs quand on utilise mprof mais les résultats sont un peu étrange:
un coup d'oeil rapide au moniteur D'activité montre qu'en fait il y a 6 Processus Python, un pour mprof pour python myscript.py et ensuite un pour chaque sous-processus de travail. Il semble que mprof ne mesure que l'utilisation de la mémoire pour le python myscript.py processus.
memory_profiler la Bibliothèque est hautement personnalisable, et je suis assez confiant que je devrais être en mesure de capturer la mémoire de chaque processus et peut-être les écrire pour séparer les fichiers journaux en utilisant la bibliothèque elle-même. Je ne sais pas par où commencer ou comment approcher ce niveau de personnalisation.
EDIT
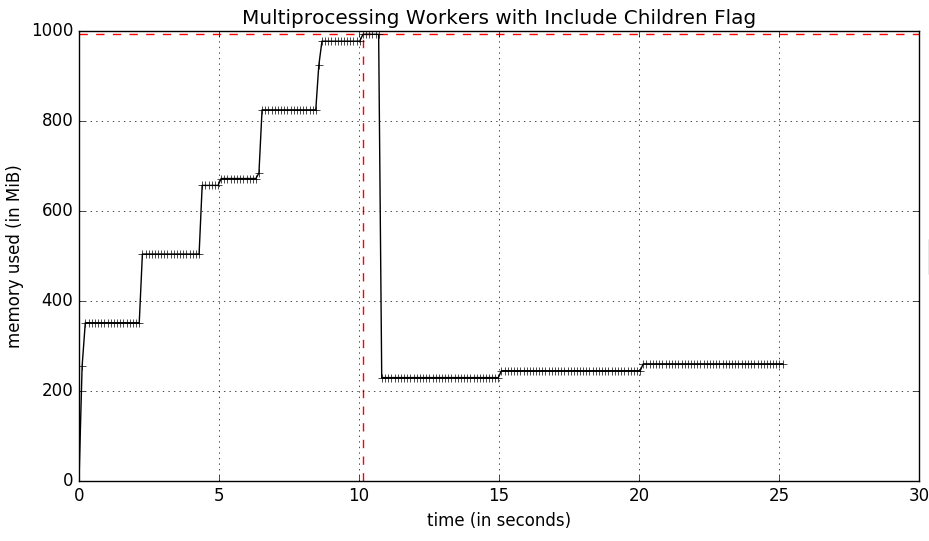
Après la lecture par le biais de l' mprof script je l'ai fait découvrir l' -C indicateur qui résume l'utilisation de la mémoire de tous les enfants (fourche). Cela conduit à un graphique (beaucoup amélioré) comme suit:
mais ce que je cherche c'est l'utilisation de la mémoire de chaque sous-processus individuel au fil du temps donc que je peux tracer tous les travailleurs (et le maître) sur le même graphique. Mon idée est d'avoir chaque sous-processus memory_usage écrit dans un fichier log différent, que je peux ensuite visualiser.
1 réponses
A partir d'aujourd'hui, une nouvelle fonctionnalité a été ajoutée à la bibliothèque de profileurs de mémoire qui fait exactement cela. Si vous avez besoin de cette fonctionnalité, mettez tout d'abord à jour memory_profiler comme suit:
$ pip install -U memory_profiler
ceci devrait installer le v0.44 libération du profileur de mémoire. Pour vérifier que c'est le cas, utilisez la commande help sur la piste d'action:
mprof run --help
Usage: mprof run [options]
Options:
--version show program's version number and exit
-h, --help show this help message and exit
--python Activates extra features when the profiling executable
is a Python program (currently: function
timestamping.)
--nopython Disables extra features when the profiled executable
is a Python program (currently: function
timestamping.)
-T INTERVAL, --interval=INTERVAL
Sampling period (in seconds), defaults to 0.1
-C, --include-children
Monitors forked processes as well (sum up all process
memory)
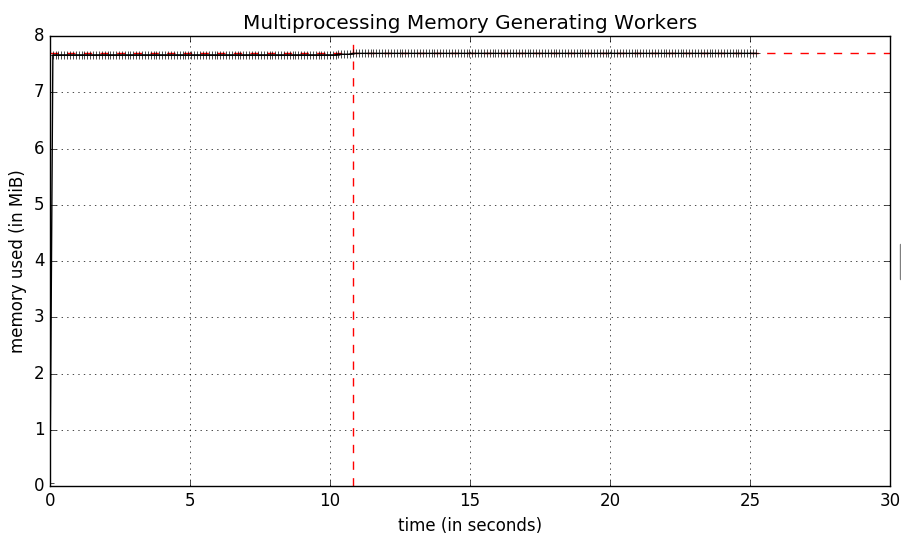
-M, --multiprocess Monitors forked processes creating individual plots
for each child
Si vous voyez les -M drapeau, alors vous êtes bon pour aller!
Vous pouvez alors lancer le votre script suit:
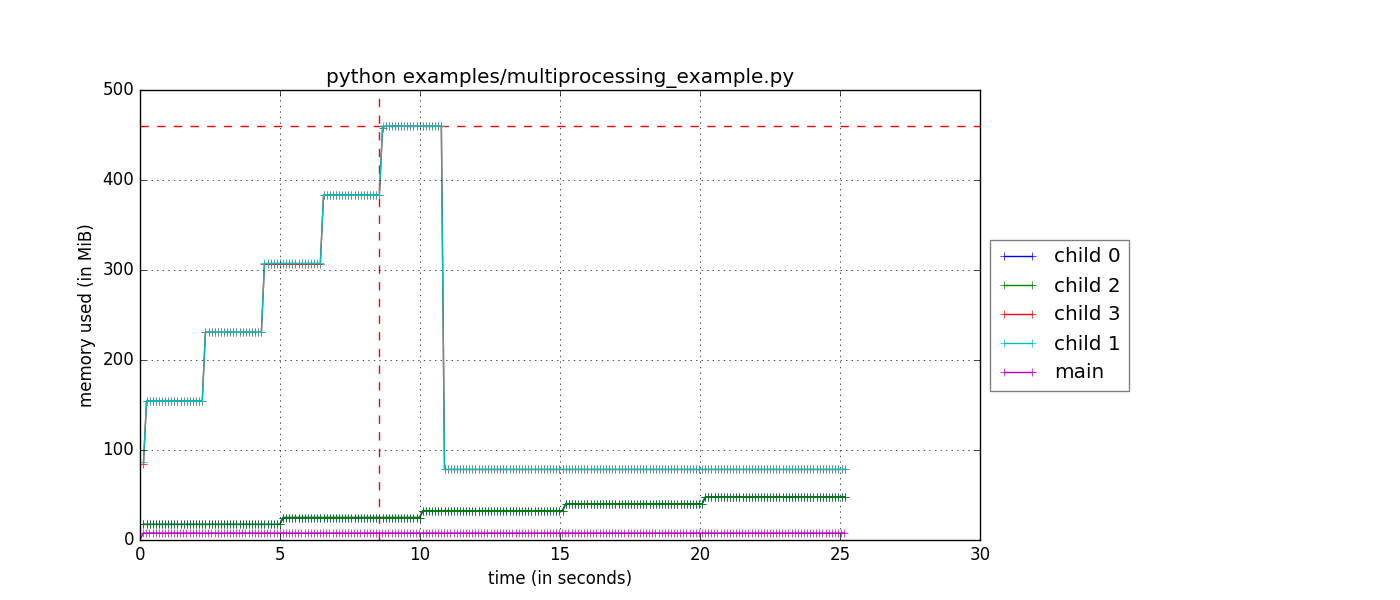
$ mprof run -M python myscript.py
$ mprof plot
Et vous devriez obtenir une figure qui ressemble à ceci:
Notez que si vous utilisez le --include-children drapeau aussi, la mémoire principale du processus sera l'utilisation totale de la mémoire de tous les enfants et de main, qui est également un tracé utile.