Comment joindre (fusionner) des cadres de données (interne, externe, gauche, droite)?
avec deux bases de données:
df1 = data.frame(CustomerId = c(1:6), Product = c(rep("Toaster", 3), rep("Radio", 3)))
df2 = data.frame(CustomerId = c(2, 4, 6), State = c(rep("Alabama", 2), rep("Ohio", 1)))
df1
# CustomerId Product
# 1 Toaster
# 2 Toaster
# 3 Toaster
# 4 Radio
# 5 Radio
# 6 Radio
df2
# CustomerId State
# 2 Alabama
# 4 Alabama
# 6 Ohio
Comment puis-je faire le style de base de données, i.e., le style sql, rejoint ? Qui est, comment puis-je obtenir:
- Un inner join de
df1etdf2:
Retourner uniquement les lignes dont la table de gauche ont touches correspondantes dans la table de droite. - Un outer join de
df1etdf2:
Renvoie toutes les lignes des deux tables, rejoignez les enregistrements de gauche qui ont des clés correspondantes dans la table de droite. - A jointure extérieure gauche (ou simplement jointure gauche) de
df1etdf2
Renvoyer toutes les lignes de la table de gauche, et toutes les lignes avec les touches correspondantes de la table de droite. - Un jointure externe droite de
df1etdf2
Renvoyer toutes les lignes de la table de droite, et toutes les lignes avec les touches correspondantes de la table de gauche.
le crédit Supplémentaire:
Comment puis-je faire une déclaration de sélection de style SQL?
13 réponses
en utilisant la fonction merge et ses paramètres optionnels:
inner jointure: merge(df1, df2) fonctionnera pour ces exemples parce que r rejoint automatiquement les cadres par des noms de variables communs, mais vous voudriez probablement spécifier merge(df1, df2, by = "CustomerId") pour s'assurer que vous vous appariez seulement sur les champs que vous vouliez. Vous pouvez également utiliser les paramètres by.x et by.y les variables ont des noms différents dans les différentes trames de données.
Outer jointure: merge(x = df1, y = df2, by = "CustomerId", all = TRUE)
externe Gauche: merge(x = df1, y = df2, by = "CustomerId", all.x = TRUE)
externe Droit: merge(x = df1, y = df2, by = "CustomerId", all.y = TRUE)
Cross join: merge(x = df1, y = df2, by = NULL)
tout comme avec la jointure interne, vous voudriez probablement passer explicitement "CustomerId" à R comme la variable correspondante. je pense qu'il est presque toujours préférable d'indiquer explicitement les identificateurs sur lequel vous souhaitez fusionner; il est plus sûr si les données d'entrée.les cadres changent de façon inattendue et sont plus faciles à lire plus tard.
vous pouvez fusionner sur plusieurs colonnes en donnant by un vecteur, par exemple, by = c("CustomerId", "OrderId") .
si les noms de colonne à fusionner ne sont pas les mêmes, vous pouvez spécifier, par exemple, by.x = "CustomerId_in_df1", by.y = "CustomerId_in_df2" where CustomerId_in_df1 is the name of the column in the first data frame and CustomerId_in_df2` est le nom de la colonne dans la deuxième base de données. (Ceux-ci peuvent également être des vecteurs si vous avez besoin de fusionner plusieurs colonnes.)
je vous recommande de vérifier paquet sqldf de Gabor Grothendieck , qui vous permet d'exprimer ces opérations en SQL.
library(sqldf)
## inner join
df3 <- sqldf("SELECT CustomerId, Product, State
FROM df1
JOIN df2 USING(CustomerID)")
## left join (substitute 'right' for right join)
df4 <- sqldf("SELECT CustomerId, Product, State
FROM df1
LEFT JOIN df2 USING(CustomerID)")
je trouve la syntaxe SQL plus simple et plus naturelle que son équivalent R (mais cela peut juste refléter mon biais RDBMS).
Voir Gabor est sqldf GitHub pour plus d'informations sur les jointures.
il y a les données .table approche pour une jointure interne, qui est très efficace de temps et de mémoire (et nécessaire pour certaines données plus grandes.cadres):
library(data.table)
dt1 <- data.table(df1, key = "CustomerId")
dt2 <- data.table(df2, key = "CustomerId")
joined.dt1.dt.2 <- dt1[dt2]
merge fonctionne également sur les données.tables (comme c'est le générique et les appels merge.data.table )
merge(dt1, dt2)
Comment faire une donnée.opération de fusion de tableaux
Traduire des jointures SQL sur les clés étrangères pour R des données.syntaxe de la table
solutions de rechange efficaces pour fusionner des données plus volumineuses.cadres
Comment faire une jointure basique externe gauche avec des données.table en R?
encore une autre option est la fonction join trouvée dans le plyr paquet
library(plyr)
join(df1, df2,
type = "inner")
# CustomerId Product State
# 1 2 Toaster Alabama
# 2 4 Radio Alabama
# 3 6 Radio Ohio
Options pour type : inner , left , right , full .
à Partir de ?join : à la différence de merge , [ join ] conserve l'ordre de x n'importe quel type de jointure est utilisé.
vous pouvez faire des jointures aussi bien en utilisant Hadley Wickham impressionnant dplyr paquet.
library(dplyr)
#make sure that CustomerId cols are both type numeric
#they ARE not using the provided code in question and dplyr will complain
df1$CustomerId <- as.numeric(df1$CustomerId)
df2$CustomerId <- as.numeric(df2$CustomerId)
jointures mutantes: ajouter des colonnes à df1 en utilisant des allumettes dans df2
#inner
inner_join(df1, df2)
#left outer
left_join(df1, df2)
#right outer
right_join(df1, df2)
#alternate right outer
left_join(df2, df1)
#full join
full_join(df1, df2)
Filtrage des jointures: éliminer les lignes en df1, ne pas modifier les colonnes
semi_join(df1, df2) #keep only observations in df1 that match in df2.
anti_join(df1, df2) #drops all observations in df1 that match in df2.
il y a quelques bons exemples de faire cela au R Wiki . Je vais en voler quelques-uns ici:
Méthode De La Fusion
puisque vos clés sont nommées de la même façon la manière courte de faire une jointure interne est la fusion ():
merge(df1,df2)
une jointure interne complète (tous les enregistrements des deux tables) peut être créée avec le mot-clé" tout":
merge(df1,df2, all=TRUE)
une jointure extérieure gauche de df1 et df2:
merge(df1,df2, all.x=TRUE)
une jointure externe droite de df1 et df2:
merge(df1,df2, all.y=TRUE)
vous pouvez flip 'em, slap 'em et rub 'em vers le bas pour obtenir les deux autres jointures externes vous m'avez demandé :)
Méthode En Indice
une jonction extérieure gauche avec df1 à gauche en utilisant une méthode en indice serait:
df1[,"State"]<-df2[df1[ ,"Product"], "State"]
l'autre combinaison de jointures externes peut être créée par mungling la jointure externe gauche indice exemple. (ouais, je sais que c'est l'équivalent de dire "je vais le laisser comme un exercice pour le lecteur...")
nouveau en 2014:
surtout si vous êtes également intéressé par la manipulation de données en général (y compris le tri, le filtrage, le sous-titrage, le résumé, etc.), vous devriez certainement jeter un oeil à dplyr , qui est livré avec une variété de fonctions toutes conçues pour faciliter votre travail spécifiquement avec les cadres de données et certains autres types de bases de données. Il offre même une interface SQL très élaborée, et même une fonction pour convertir (la plupart) le code SQL directement en R.
les quatre fonctions de jointure dans le paquet dplyr sont (pour citer):
-
inner_join(x, y, by = NULL, copy = FALSE, ...): retourner toutes les lignes de x où il y a des valeurs correspondantes dans y, et toutes les colonnes de x et y -
left_join(x, y, by = NULL, copy = FALSE, ...): retourner toutes les lignes de x, et toutes les colonnes de x et y -
semi_join(x, y, by = NULL, copy = FALSE, ...): retournez toutes les lignes de x où il y a des valeurs correspondantes dans y, en gardant juste des colonnes de X. -
anti_join(x, y, by = NULL, copy = FALSE, ...): retourner toutes les lignes de x lorsqu'il n'y a pas de valeurs correspondantes dans y, en gardant seulement les colonnes de x
c'est tout ici dans les moindres détails.
sélectionner les colonnes peut être fait par select(df,"column") . Si ce N'est pas assez SQL-ish pour vous, alors il y a la fonction sql() , dans laquelle vous pouvez entrer le code SQL tel quel, et elle fera l'opération que vous avez spécifiée comme vous écriviez dans R all (pour plus d'informations, veuillez vous référer à la dplyr/bases de données vignette ). Par exemple, si appliqué correctement, sql("SELECT * FROM hflights") sélectionnera toutes les colonnes de la table dplyr "hflights" (un "tbl").
mise à Jour des données.méthodes de table pour joindre des ensembles de données. Voir ci-dessous des exemples pour chaque type de jointure. Il existe deux méthodes, l'une à partir de [.data.table lors de la transmission des secondes données.table comme premier argument à sous-ensemble, une autre façon est d'utiliser la fonction merge qui a expédié aux données rapides.le tableau de la méthode.
mise à Jour sur 2016-04-01 - et ce n'est pas un poisson d'avril blague!
Dans la version 1.9.7 des données.les jointures de table sont maintenant capables de utilisez l'index existant qui réduit considérablement le temps de jointure. ci-dessous code et benchmark n'utilise pas de données.(') indices sur la période . Si vous êtes à la recherche de près en temps réel joindre vous devriez utiliser des données.tableau des indices.
df1 = data.frame(CustomerId = c(1:6), Product = c(rep("Toaster", 3), rep("Radio", 3)))
df2 = data.frame(CustomerId = c(2L, 4L, 7L), State = c(rep("Alabama", 2), rep("Ohio", 1))) # one value changed to show full outer join
library(data.table)
dt1 = as.data.table(df1)
dt2 = as.data.table(df2)
setkey(dt1, CustomerId)
setkey(dt2, CustomerId)
# right outer join keyed data.tables
dt1[dt2]
setkey(dt1, NULL)
setkey(dt2, NULL)
# right outer join unkeyed data.tables - use `on` argument
dt1[dt2, on = "CustomerId"]
# left outer join - swap dt1 with dt2
dt2[dt1, on = "CustomerId"]
# inner join - use `nomatch` argument
dt1[dt2, nomatch=0L, on = "CustomerId"]
# anti join - use `!` operator
dt1[!dt2, on = "CustomerId"]
# inner join
merge(dt1, dt2, by = "CustomerId")
# full outer join
merge(dt1, dt2, by = "CustomerId", all = TRUE)
# see ?merge.data.table arguments for other cases
ci-Dessous les tests de référence de la base de R, sqldf, dplyr et de données.table.
Tests de référence ensembles de données non indexés/non indexés. Vous pouvez obtenir des performances encore meilleures si vous utilisez des clés sur vos données.tables ou index avec sqldf. La Base R et dplyr n'ayant pas d'indices ou de clés, je n'ai pas inclus ce scénario dans le benchmark.
Le Benchmark est effectué sur des ensembles de données de 5m-1 rows, il y a des valeurs communes de 5M-2 sur la colonne de jointure donc chaque scénario (gauche, droite, plein, intérieur) peut être testé et la jointure n'est pas encore triviale à effectuer.
library(microbenchmark)
library(sqldf)
library(dplyr)
library(data.table)
n = 5e6
set.seed(123)
df1 = data.frame(x=sample(n,n-1L), y1=rnorm(n-1L))
df2 = data.frame(x=sample(n,n-1L), y2=rnorm(n-1L))
dt1 = as.data.table(df1)
dt2 = as.data.table(df2)
# inner join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x"),
sqldf = sqldf("SELECT * FROM df1 INNER JOIN df2 ON df1.x = df2.x"),
dplyr = inner_join(df1, df2, by = "x"),
data.table = dt1[dt2, nomatch = 0L, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 15546.0097 16083.4915 16687.117 16539.0148 17388.290 18513.216 10
# sqldf 44392.6685 44709.7128 45096.401 45067.7461 45504.376 45563.472 10
# dplyr 4124.0068 4248.7758 4281.122 4272.3619 4342.829 4411.388 10
# data.table 937.2461 946.0227 1053.411 973.0805 1214.300 1281.958 10
# left outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all.x = TRUE),
sqldf = sqldf("SELECT * FROM df1 LEFT OUTER JOIN df2 ON df1.x = df2.x"),
dplyr = left_join(df1, df2, by = c("x"="x")),
data.table = dt2[dt1, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 16140.791 17107.7366 17441.9538 17414.6263 17821.9035 19453.034 10
# sqldf 43656.633 44141.9186 44777.1872 44498.7191 45288.7406 47108.900 10
# dplyr 4062.153 4352.8021 4780.3221 4409.1186 4450.9301 8385.050 10
# data.table 823.218 823.5557 901.0383 837.9206 883.3292 1277.239 10
# right outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all.y = TRUE),
sqldf = sqldf("SELECT * FROM df2 LEFT OUTER JOIN df1 ON df2.x = df1.x"),
dplyr = right_join(df1, df2, by = "x"),
data.table = dt1[dt2, on = "x"])
#Unit: milliseconds
# expr min lq mean median uq max neval
# base 15821.3351 15954.9927 16347.3093 16044.3500 16621.887 17604.794 10
# sqldf 43635.5308 43761.3532 43984.3682 43969.0081 44044.461 44499.891 10
# dplyr 3936.0329 4028.1239 4102.4167 4045.0854 4219.958 4307.350 10
# data.table 820.8535 835.9101 918.5243 887.0207 1005.721 1068.919 10
# full outer join
microbenchmark(times = 10L,
base = merge(df1, df2, by = "x", all = TRUE),
#sqldf = sqldf("SELECT * FROM df1 FULL OUTER JOIN df2 ON df1.x = df2.x"), # not supported
dplyr = full_join(df1, df2, by = "x"),
data.table = merge(dt1, dt2, by = "x", all = TRUE))
#Unit: seconds
# expr min lq mean median uq max neval
# base 16.176423 16.908908 17.485457 17.364857 18.271790 18.626762 10
# dplyr 7.610498 7.666426 7.745850 7.710638 7.832125 7.951426 10
# data.table 2.052590 2.130317 2.352626 2.208913 2.470721 2.951948 10
dplyr puisque 0,4 mis en œuvre toutes ces jointures, y compris outer_join, mais il convient de noter que pour les premières versions il l'habitude de ne pas offrir outer_join, et comme un résultat il y avait beaucoup de vraiment mauvais hacky solution de contournement code d'utilisateur flottant autour pour un certain temps (vous pouvez toujours trouver ce et Kaggle réponses à partir de cette période).
relatives à la Jointure de libération souligne :
- manipulation pour le type POSIXct, les fuseaux horaires, les doublons, les différents niveaux de facteurs. Meilleures erreurs et avertissements.
- nouvel argument de suffixe pour contrôler ce que le suffixe double les noms de variables reçoivent (#1296)
- mettre en œuvre le droit d'adhésion et l'adhésion externe (#96)
- les jointures mutantes, qui ajoutent de nouvelles variables à une table à partir des lignes correspondantes dans une autre. Le filtrage des jointures, qui filtre les observations d'une table en fonction de si elles correspondent ou non à une observation de l'autre table.
- peut maintenant left_join par différentes variables dans chaque tableau: df1 % > % left_join (df2, c ("var1" = "var2")
- *_join() n'est plus réorganise les noms de colonnes (n ° 324)
v0.1.3 (4/2014)
- has inner_join, left_join, semi_join, anti_join
- outer_join pas encore mis en œuvre, la modification du traitement est l'utilisation de la base de::merge() (ou plyr::join())
- n'a pas encore mettre en œuvre right_join et outer_join
- Hadley mentionnant d'autres avantages ici
- un mineur feature merge a actuellement que dplyr n'est pas la possibilité d'avoir séparé par.x,par.y colonnes comme par exemple Python pandas.
Solutions de contournement par hadley commentaires à cette question:
- right_join (x,y) est le même que left_join(y,x) en termes de lignes, juste les colonnes seront des ordres différents. Facilement travaillé avec select (new_column_order)
- outer_join est fondamentalement de l'union(left_join(x, y), right_join(x, y)) - c'est à dire préserver toutes les lignes dans les deux trames de données.
en joignant deux bases de données avec ~1 million de lignes chacune, une avec 2 colonnes et l'autre avec ~20, j'ai trouvé étonnamment merge(..., all.x = TRUE, all.y = TRUE) pour être plus rapide que dplyr::full_join() . C'est avec dplyr v0.4
la fusion prend ~ 17 secondes, full_join prend ~65 secondes.
un peu de nourriture pour bien, puisque je manque généralement à dplyr pour les tâches de manipulation.
dans le cas d'une jointure de gauche avec une cardinalité 0..*:0..1 ou d'une jointure de droite avec une cardinalité 0..1:0..* , il est possible d'assigner en place les colonnes unilatérales du jointeur (la table 0..1 ) directement sur le joinee (la table 0..* ), évitant ainsi la création d'une table de données entièrement nouvelle. Pour ce faire, il faut apparier les colonnes clés de la joinee dans le menuisier et indexer+ordonner les lignes du menuisier en conséquence pour la tâche.
Si la clé est une seule colonne, alors nous pouvons utiliser un seul appel à match() pour faire la correspondance. C'est le cas, je vais couvrir dans cette réponse.
voici un exemple basé sur L'OP, sauf que j'ai ajouté une ligne supplémentaire à df2 avec un id de 7 pour tester le cas d'une clé non-assortie dans le menuisier. C'est effectivement df1 joindre gauche df2 :
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L)));
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas'));
df1[names(df2)[-1L]] <- df2[match(df1[,1L],df2[,1L]),-1L];
df1;
## CustomerId Product State
## 1 1 Toaster <NA>
## 2 2 Toaster Alabama
## 3 3 Toaster <NA>
## 4 4 Radio Alabama
## 5 5 Radio <NA>
## 6 6 Radio Ohio
Dans le ci-dessus, je une hypothèse codée en dur selon laquelle la colonne clé est la première colonne des deux tableaux d'entrée. Je dirais que, en général, ce n'est pas une hypothèse déraisonnable, puisque, si vous avez des données.cadre avec une colonne de clé, il serait étrange si elle n'avait pas été mis en place comme la première colonne de données.cadre dès le départ. Et vous pouvez toujours modifier l'ordre des colonnes. Avantageux conséquence de cette hypothèse est que le nom de la colonne clé ne doit pas être codée en dur, même si je suppose c'est juste remplacer une hypothèse par une autre. La Concision est un autre avantage de l'indexation entière, ainsi que la vitesse. Dans les benchmarks ci-dessous, je vais changer l'implémentation pour utiliser l'indexation des noms de chaîne pour correspondre aux implémentations concurrentes.
je pense que c'est une solution particulièrement appropriée si vous avez plusieurs tables que vous voulez à gauche joindre contre une seule grande table. La reconstruction répétée de la table entière pour chaque Fusion serait inutile et inefficace.
d'un autre côté, si vous avez besoin que le joinee reste inchangé par cette opération pour quelque raison que ce soit, alors cette solution ne peut pas être utilisée, puisqu'elle modifie le joinee directement. Bien que dans ce cas vous puissiez simplement faire une copie et effectuer la(les) cession (s) en place sur la copie.
en guise de note, j'ai brièvement examiné les solutions de correspondance possibles pour les clés à plusieurs colonnes. Malheureusement, le seul correspondant les solutions que j'ai trouvées étaient:
- concaténations inefficaces. par exemple
match(interaction(df1$a,df1$b),interaction(df2$a,df2$b)), ou la même idée avecpaste(). - conjonctions cartésiennes inefficaces, p.ex.
outer(df1$a,df2$a,`==`) & outer(df1$b,df2$b,`==`). - base R
merge()et des fonctions équivalentes de fusion basées sur un paquet, qui attribuent toujours une nouvelle table pour retourner le résultat fusionné, et ne sont donc pas appropriés pour une solution basée sur une assignation en place.
par exemple, voir appariement de plusieurs colonnes sur différents cadres de données et obtention d'une autre colonne comme résultat , appariement de deux colonnes avec deux autres colonnes , appariement sur plusieurs colonnes , et le dupe de cette question Où je suis initialement venu avec la solution en place, combiner deux cadres de données avec un nombre différent de lignes dans R .
Benchmarking
j'ai décidé de faire ma propre analyse comparative pour voir comment l'approche d'affectation sur place se compare aux autres solutions qui ont été offertes dans cette question.
code D'essai:
library(microbenchmark);
library(data.table);
library(sqldf);
library(plyr);
library(dplyr);
solSpecs <- list(
merge=list(testFuncs=list(
inner=function(df1,df2,key) merge(df1,df2,key),
left =function(df1,df2,key) merge(df1,df2,key,all.x=T),
right=function(df1,df2,key) merge(df1,df2,key,all.y=T),
full =function(df1,df2,key) merge(df1,df2,key,all=T)
)),
data.table.unkeyed=list(argSpec='data.table.unkeyed',testFuncs=list(
inner=function(dt1,dt2,key) dt1[dt2,on=key,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2,key) dt2[dt1,on=key,allow.cartesian=T],
right=function(dt1,dt2,key) dt1[dt2,on=key,allow.cartesian=T],
full =function(dt1,dt2,key) merge(dt1,dt2,key,all=T,allow.cartesian=T) ## calls merge.data.table()
)),
data.table.keyed=list(argSpec='data.table.keyed',testFuncs=list(
inner=function(dt1,dt2) dt1[dt2,nomatch=0L,allow.cartesian=T],
left =function(dt1,dt2) dt2[dt1,allow.cartesian=T],
right=function(dt1,dt2) dt1[dt2,allow.cartesian=T],
full =function(dt1,dt2) merge(dt1,dt2,all=T,allow.cartesian=T) ## calls merge.data.table()
)),
sqldf.unindexed=list(testFuncs=list( ## note: must pass connection=NULL to avoid running against the live DB connection, which would result in collisions with the residual tables from the last query upload
inner=function(df1,df2,key) sqldf(paste0('select * from df1 inner join df2 using(',paste(collapse=',',key),')'),connection=NULL),
left =function(df1,df2,key) sqldf(paste0('select * from df1 left join df2 using(',paste(collapse=',',key),')'),connection=NULL),
right=function(df1,df2,key) sqldf(paste0('select * from df2 left join df1 using(',paste(collapse=',',key),')'),connection=NULL) ## can't do right join proper, not yet supported; inverted left join is equivalent
##full =function(df1,df2,key) sqldf(paste0('select * from df1 full join df2 using(',paste(collapse=',',key),')'),connection=NULL) ## can't do full join proper, not yet supported; possible to hack it with a union of left joins, but too unreasonable to include in testing
)),
sqldf.indexed=list(testFuncs=list( ## important: requires an active DB connection with preindexed main.df1 and main.df2 ready to go; arguments are actually ignored
inner=function(df1,df2,key) sqldf(paste0('select * from main.df1 inner join main.df2 using(',paste(collapse=',',key),')')),
left =function(df1,df2,key) sqldf(paste0('select * from main.df1 left join main.df2 using(',paste(collapse=',',key),')')),
right=function(df1,df2,key) sqldf(paste0('select * from main.df2 left join main.df1 using(',paste(collapse=',',key),')')) ## can't do right join proper, not yet supported; inverted left join is equivalent
##full =function(df1,df2,key) sqldf(paste0('select * from main.df1 full join main.df2 using(',paste(collapse=',',key),')')) ## can't do full join proper, not yet supported; possible to hack it with a union of left joins, but too unreasonable to include in testing
)),
plyr=list(testFuncs=list(
inner=function(df1,df2,key) join(df1,df2,key,'inner'),
left =function(df1,df2,key) join(df1,df2,key,'left'),
right=function(df1,df2,key) join(df1,df2,key,'right'),
full =function(df1,df2,key) join(df1,df2,key,'full')
)),
dplyr=list(testFuncs=list(
inner=function(df1,df2,key) inner_join(df1,df2,key),
left =function(df1,df2,key) left_join(df1,df2,key),
right=function(df1,df2,key) right_join(df1,df2,key),
full =function(df1,df2,key) full_join(df1,df2,key)
)),
in.place=list(testFuncs=list(
left =function(df1,df2,key) { cns <- setdiff(names(df2),key); df1[cns] <- df2[match(df1[,key],df2[,key]),cns]; df1; },
right=function(df1,df2,key) { cns <- setdiff(names(df1),key); df2[cns] <- df1[match(df2[,key],df1[,key]),cns]; df2; }
))
);
getSolTypes <- function() names(solSpecs);
getJoinTypes <- function() unique(unlist(lapply(solSpecs,function(x) names(x$testFuncs))));
getArgSpec <- function(argSpecs,key=NULL) if (is.null(key)) argSpecs$default else argSpecs[[key]];
initSqldf <- function() {
sqldf(); ## creates sqlite connection on first run, cleans up and closes existing connection otherwise
if (exists('sqldfInitFlag',envir=globalenv(),inherits=F) && sqldfInitFlag) { ## false only on first run
sqldf(); ## creates a new connection
} else {
assign('sqldfInitFlag',T,envir=globalenv()); ## set to true for the one and only time
}; ## end if
invisible();
}; ## end initSqldf()
setUpBenchmarkCall <- function(argSpecs,joinType,solTypes=getSolTypes(),env=parent.frame()) {
## builds and returns a list of expressions suitable for passing to the list argument of microbenchmark(), and assigns variables to resolve symbol references in those expressions
callExpressions <- list();
nms <- character();
for (solType in solTypes) {
testFunc <- solSpecs[[solType]]$testFuncs[[joinType]];
if (is.null(testFunc)) next; ## this join type is not defined for this solution type
testFuncName <- paste0('tf.',solType);
assign(testFuncName,testFunc,envir=env);
argSpecKey <- solSpecs[[solType]]$argSpec;
argSpec <- getArgSpec(argSpecs,argSpecKey);
argList <- setNames(nm=names(argSpec$args),vector('list',length(argSpec$args)));
for (i in seq_along(argSpec$args)) {
argName <- paste0('tfa.',argSpecKey,i);
assign(argName,argSpec$args[[i]],envir=env);
argList[[i]] <- if (i%in%argSpec$copySpec) call('copy',as.symbol(argName)) else as.symbol(argName);
}; ## end for
callExpressions[[length(callExpressions)+1L]] <- do.call(call,c(list(testFuncName),argList),quote=T);
nms[length(nms)+1L] <- solType;
}; ## end for
names(callExpressions) <- nms;
callExpressions;
}; ## end setUpBenchmarkCall()
harmonize <- function(res) {
res <- as.data.frame(res); ## coerce to data.frame
for (ci in which(sapply(res,is.factor))) res[[ci]] <- as.character(res[[ci]]); ## coerce factor columns to character
for (ci in which(sapply(res,is.logical))) res[[ci]] <- as.integer(res[[ci]]); ## coerce logical columns to integer (works around sqldf quirk of munging logicals to integers)
##for (ci in which(sapply(res,inherits,'POSIXct'))) res[[ci]] <- as.double(res[[ci]]); ## coerce POSIXct columns to double (works around sqldf quirk of losing POSIXct class) ----- POSIXct doesn't work at all in sqldf.indexed
res <- res[order(names(res))]; ## order columns
res <- res[do.call(order,res),]; ## order rows
res;
}; ## end harmonize()
checkIdentical <- function(argSpecs,solTypes=getSolTypes()) {
for (joinType in getJoinTypes()) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
if (length(callExpressions)<2L) next;
ex <- harmonize(eval(callExpressions[[1L]]));
for (i in seq(2L,len=length(callExpressions)-1L)) {
y <- harmonize(eval(callExpressions[[i]]));
if (!isTRUE(all.equal(ex,y,check.attributes=F))) {
ex <<- ex;
y <<- y;
solType <- names(callExpressions)[i];
stop(paste0('non-identical: ',solType,' ',joinType,'.'));
}; ## end if
}; ## end for
}; ## end for
invisible();
}; ## end checkIdentical()
testJoinType <- function(argSpecs,joinType,solTypes=getSolTypes(),metric=NULL,times=100L) {
callExpressions <- setUpBenchmarkCall(argSpecs,joinType,solTypes);
bm <- microbenchmark(list=callExpressions,times=times);
if (is.null(metric)) return(bm);
bm <- summary(bm);
res <- setNames(nm=names(callExpressions),bm[[metric]]);
attr(res,'unit') <- attr(bm,'unit');
res;
}; ## end testJoinType()
testAllJoinTypes <- function(argSpecs,solTypes=getSolTypes(),metric=NULL,times=100L) {
joinTypes <- getJoinTypes();
resList <- setNames(nm=joinTypes,lapply(joinTypes,function(joinType) testJoinType(argSpecs,joinType,solTypes,metric,times)));
if (is.null(metric)) return(resList);
units <- unname(unlist(lapply(resList,attr,'unit')));
res <- do.call(data.frame,c(list(join=joinTypes),setNames(nm=solTypes,rep(list(rep(NA_real_,length(joinTypes))),length(solTypes))),list(unit=units,stringsAsFactors=F)));
for (i in seq_along(resList)) res[i,match(names(resList[[i]]),names(res))] <- resList[[i]];
res;
}; ## end testAllJoinTypes()
testGrid <- function(makeArgSpecsFunc,sizes,overlaps,solTypes=getSolTypes(),joinTypes=getJoinTypes(),metric='median',times=100L) {
res <- expand.grid(size=sizes,overlap=overlaps,joinType=joinTypes,stringsAsFactors=F);
res[solTypes] <- NA_real_;
res$unit <- NA_character_;
for (ri in seq_len(nrow(res))) {
size <- res$size[ri];
overlap <- res$overlap[ri];
joinType <- res$joinType[ri];
argSpecs <- makeArgSpecsFunc(size,overlap);
checkIdentical(argSpecs,solTypes);
cur <- testJoinType(argSpecs,joinType,solTypes,metric,times);
res[ri,match(names(cur),names(res))] <- cur;
res$unit[ri] <- attr(cur,'unit');
}; ## end for
res;
}; ## end testGrid()
voici un benchmark de l'exemple basé sur L'OP que j'ai démontré plus tôt:
## OP's example, supplemented with a non-matching row in df2
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(CustomerId=1:6,Product=c(rep('Toaster',3L),rep('Radio',3L))),
df2 <- data.frame(CustomerId=c(2L,4L,6L,7L),State=c(rep('Alabama',2L),'Ohio','Texas')),
'CustomerId'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'CustomerId'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),CustomerId),
setkey(as.data.table(df2),CustomerId)
))
);
## prepare sqldf
initSqldf();
sqldf('create index df1_key on df1(CustomerId);'); ## upload and create an sqlite index on df1
sqldf('create index df2_key on df2(CustomerId);'); ## upload and create an sqlite index on df2
checkIdentical(argSpecs);
testAllJoinTypes(argSpecs,metric='median');
## join merge data.table.unkeyed data.table.keyed sqldf.unindexed sqldf.indexed plyr dplyr in.place unit
## 1 inner 644.259 861.9345 923.516 9157.752 1580.390 959.2250 270.9190 NA microseconds
## 2 left 713.539 888.0205 910.045 8820.334 1529.714 968.4195 270.9185 224.3045 microseconds
## 3 right 1221.804 909.1900 923.944 8930.668 1533.135 1063.7860 269.8495 218.1035 microseconds
## 4 full 1302.203 3107.5380 3184.729 NA NA 1593.6475 270.7055 NA microseconds
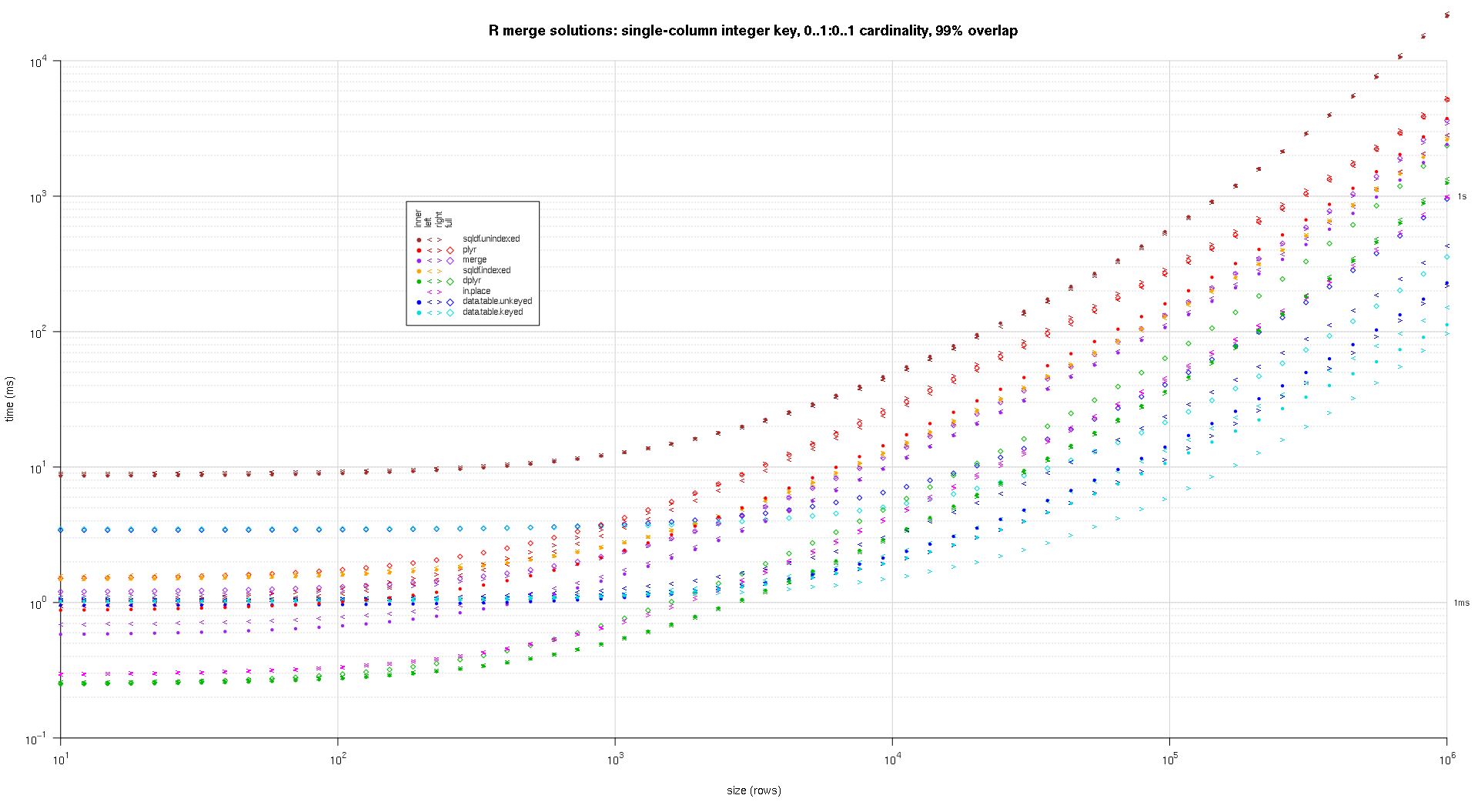
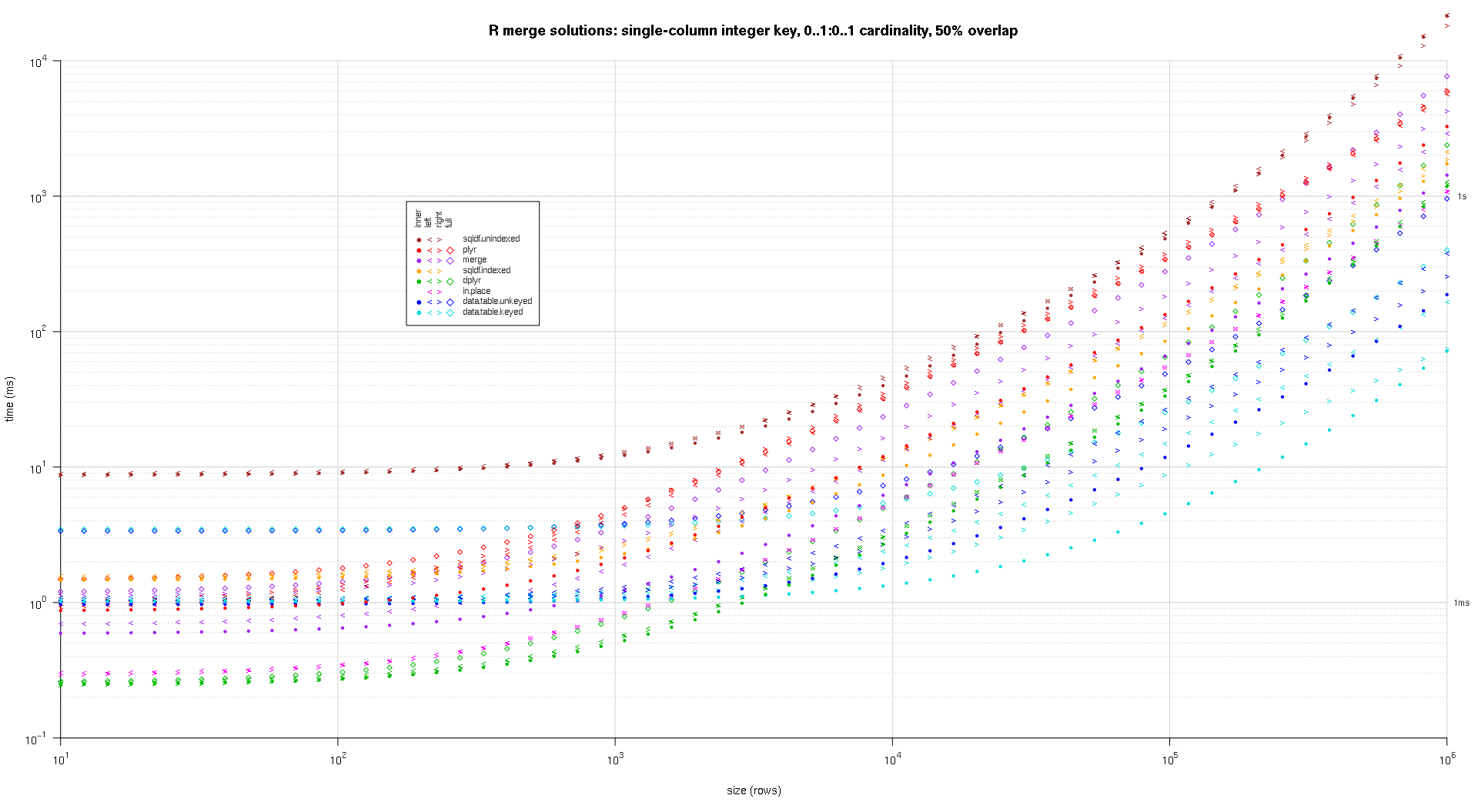
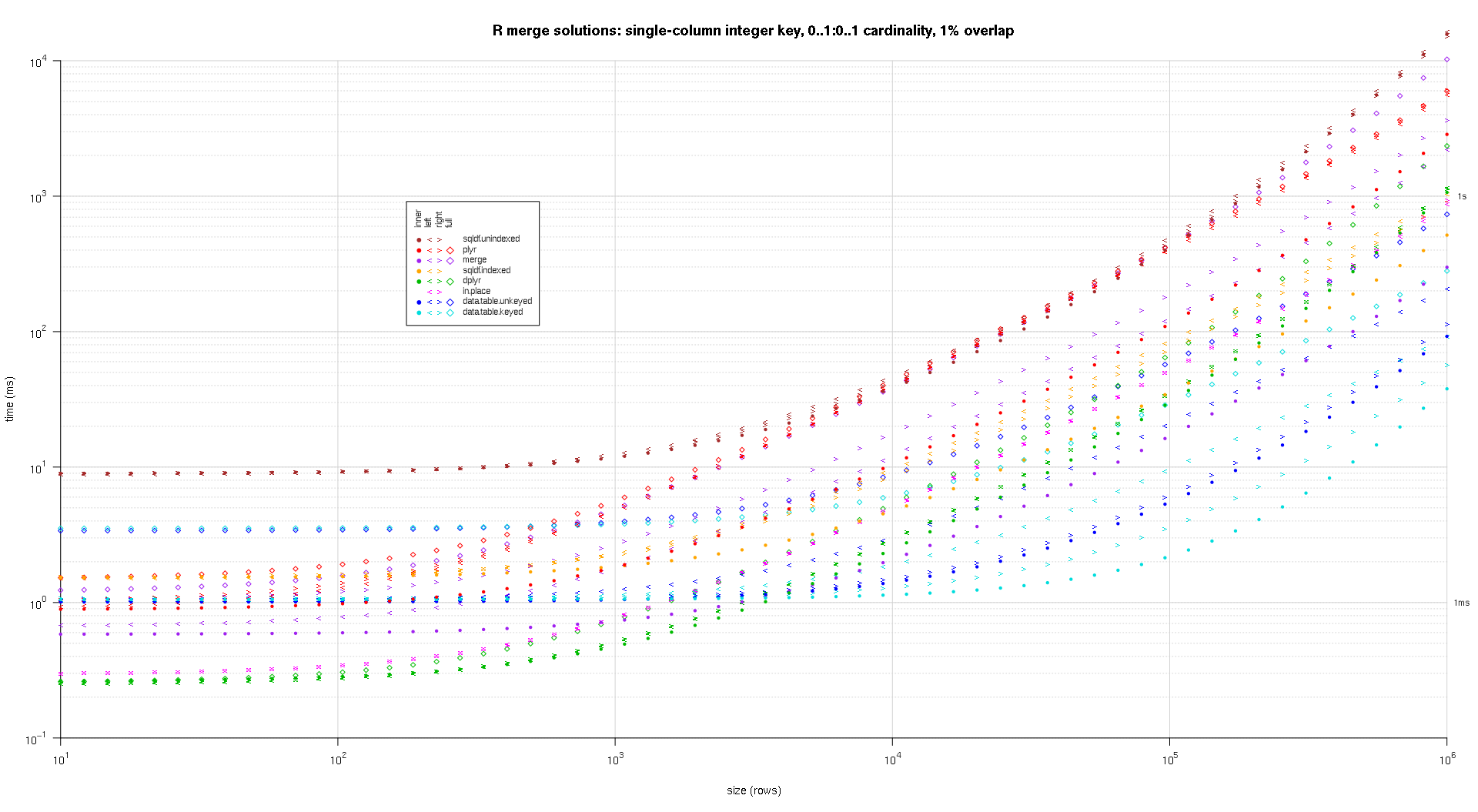
données d'entrée aléatoires, en essayant différentes échelles et différents modèles de chevauchement des clés entre les deux tables d'entrée. Cette référence est encore restreint au cas d'une seule colonne clé entière. De plus, pour s'assurer que la solution en place fonctionnerait pour les jointures gauche et droite des mêmes tables, toutes les données de tests aléatoires utilisent la cardinalité 0..1:0..1 . Pour ce faire, on procède à un échantillonnage sans remplacer la colonne clé des premières données.frame lors de la génération de la colonne clé de la deuxième donnée.cadre.
makeArgSpecs.singleIntegerKey.optionalOneToOne <- function(size,overlap) {
com <- as.integer(size*overlap);
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- data.frame(id=sample(size),y1=rnorm(size),y2=rnorm(size)),
df2 <- data.frame(id=sample(c(if (com>0L) sample(df1$id,com) else integer(),seq(size+1L,len=size-com))),y3=rnorm(size),y4=rnorm(size)),
'id'
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
'id'
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkey(as.data.table(df1),id),
setkey(as.data.table(df2),id)
))
);
## prepare sqldf
initSqldf();
sqldf('create index df1_key on df1(id);'); ## upload and create an sqlite index on df1
sqldf('create index df2_key on df2(id);'); ## upload and create an sqlite index on df2
argSpecs;
}; ## end makeArgSpecs.singleIntegerKey.optionalOneToOne()
## cross of various input sizes and key overlaps
sizes <- c(1e1L,1e3L,1e6L);
overlaps <- c(0.99,0.5,0.01);
system.time({ res <- testGrid(makeArgSpecs.singleIntegerKey.optionalOneToOne,sizes,overlaps); });
## user system elapsed
## 22024.65 12308.63 34493.19
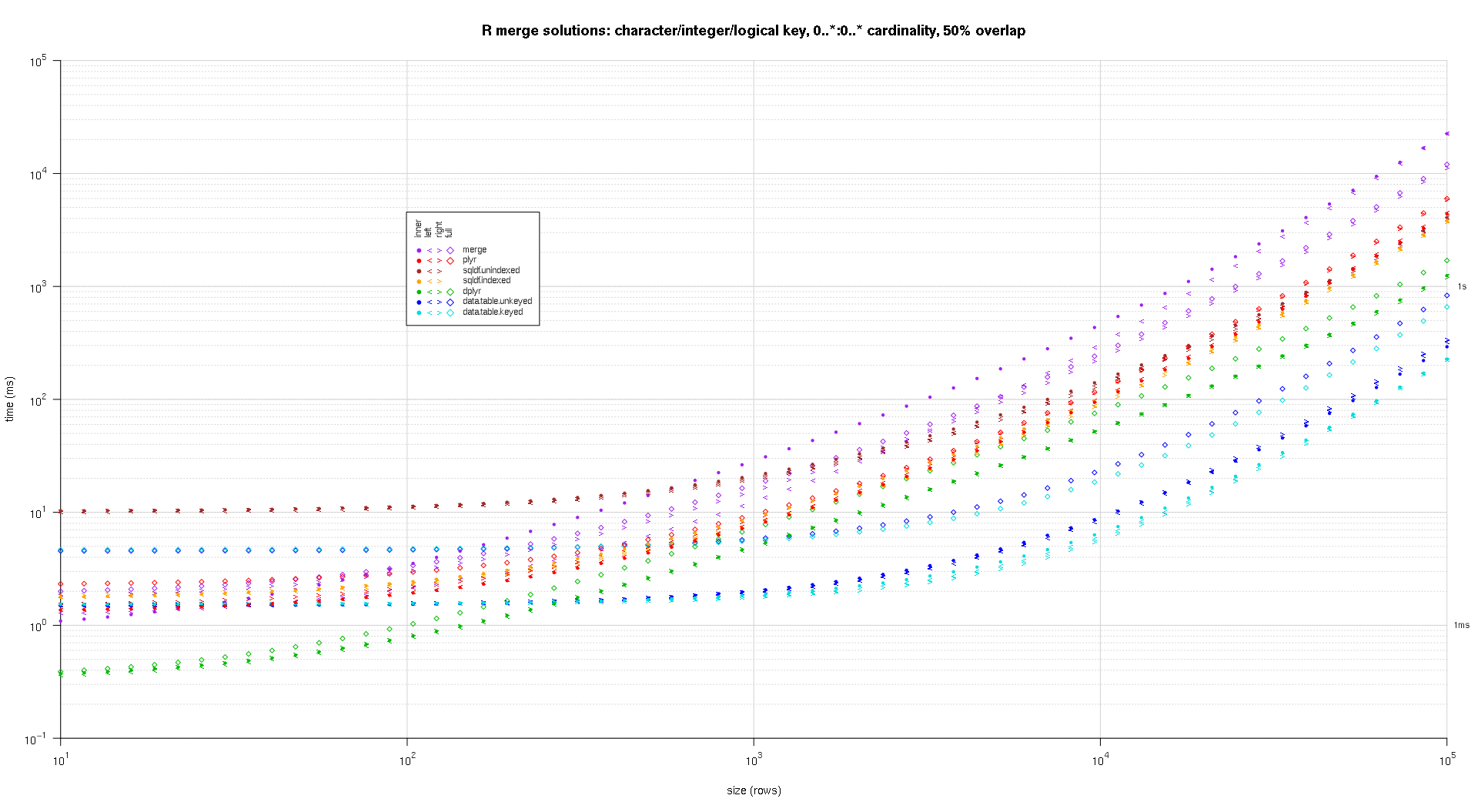
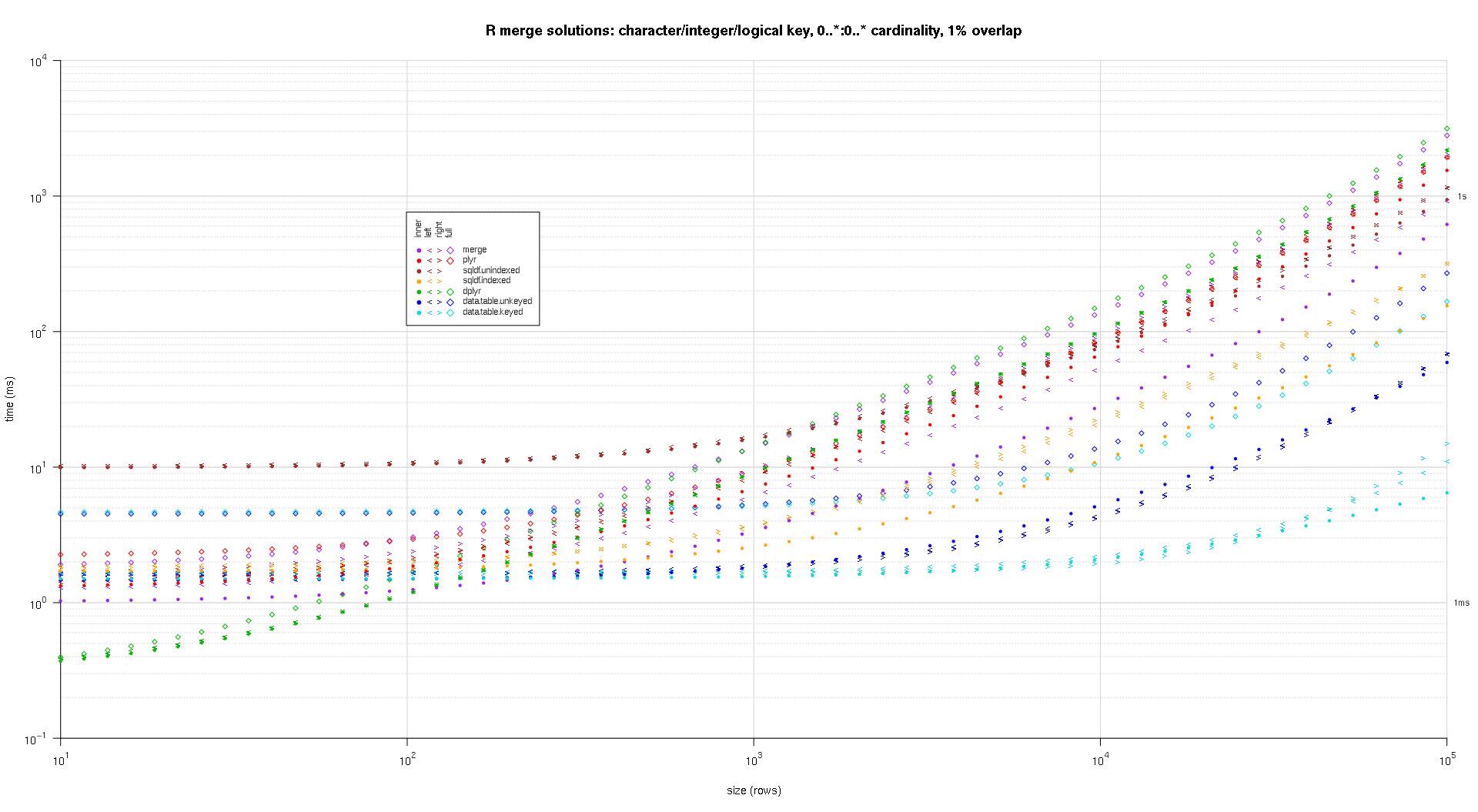
j'ai écrit du code pour créer des tracés log-log des résultats ci-dessus. J'ai généré un graphique séparé pour chaque pourcentage de chevauchement. C'est un peu encombré, mais j'aime avoir tous les types de solution et de jointure représentés dans la même parcelle.
j'ai utilisé l'interpolation spline pour montrer une courbe lisse pour chaque combinaison de type solution/jointure, dessinée avec les symboles individuels de pch. Le type de jointure est saisi par le symbole pch, en utilisant un point pour intérieure, à gauche et à droite crochets gauche et droit, et un diamant pour complet. Le type de solution est capturé par la couleur comme indiqué dans la légende.
plotRes <- function(res,titleFunc,useFloor=F) {
solTypes <- setdiff(names(res),c('size','overlap','joinType','unit')); ## derive from res
normMult <- c(microseconds=1e-3,milliseconds=1); ## normalize to milliseconds
joinTypes <- getJoinTypes();
cols <- c(merge='purple',data.table.unkeyed='blue',data.table.keyed='#00DDDD',sqldf.unindexed='brown',sqldf.indexed='orange',plyr='red',dplyr='#00BB00',in.place='magenta');
pchs <- list(inner=20L,left='<',right='>',full=23L);
cexs <- c(inner=0.7,left=1,right=1,full=0.7);
NP <- 60L;
ord <- order(decreasing=T,colMeans(res[res$size==max(res$size),solTypes],na.rm=T));
ymajors <- data.frame(y=c(1,1e3),label=c('1ms','1s'),stringsAsFactors=F);
for (overlap in unique(res$overlap)) {
x1 <- res[res$overlap==overlap,];
x1[solTypes] <- x1[solTypes]*normMult[x1$unit]; x1$unit <- NULL;
xlim <- c(1e1,max(x1$size));
xticks <- 10^seq(log10(xlim[1L]),log10(xlim[2L]));
ylim <- c(1e-1,10^((if (useFloor) floor else ceiling)(log10(max(x1[solTypes],na.rm=T))))); ## use floor() to zoom in a little more, only sqldf.unindexed will break above, but xpd=NA will keep it visible
yticks <- 10^seq(log10(ylim[1L]),log10(ylim[2L]));
yticks.minor <- rep(yticks[-length(yticks)],each=9L)*1:9;
plot(NA,xlim=xlim,ylim=ylim,xaxs='i',yaxs='i',axes=F,xlab='size (rows)',ylab='time (ms)',log='xy');
abline(v=xticks,col='lightgrey');
abline(h=yticks.minor,col='lightgrey',lty=3L);
abline(h=yticks,col='lightgrey');
axis(1L,xticks,parse(text=sprintf('10^%d',as.integer(log10(xticks)))));
axis(2L,yticks,parse(text=sprintf('10^%d',as.integer(log10(yticks)))),las=1L);
axis(4L,ymajors$y,ymajors$label,las=1L,tick=F,cex.axis=0.7,hadj=0.5);
for (joinType in rev(joinTypes)) { ## reverse to draw full first, since it's larger and would be more obtrusive if drawn last
x2 <- x1[x1$joinType==joinType,];
for (solType in solTypes) {
if (any(!is.na(x2[[solType]]))) {
xy <- spline(x2$size,x2[[solType]],xout=10^(seq(log10(x2$size[1L]),log10(x2$size[nrow(x2)]),len=NP)));
points(xy$x,xy$y,pch=pchs[[joinType]],col=cols[solType],cex=cexs[joinType],xpd=NA);
}; ## end if
}; ## end for
}; ## end for
## custom legend
## due to logarithmic skew, must do all distance calcs in inches, and convert to user coords afterward
## the bottom-left corner of the legend will be defined in normalized figure coords, although we can convert to inches immediately
leg.cex <- 0.7;
leg.x.in <- grconvertX(0.275,'nfc','in');
leg.y.in <- grconvertY(0.6,'nfc','in');
leg.x.user <- grconvertX(leg.x.in,'in');
leg.y.user <- grconvertY(leg.y.in,'in');
leg.outpad.w.in <- 0.1;
leg.outpad.h.in <- 0.1;
leg.midpad.w.in <- 0.1;
leg.midpad.h.in <- 0.1;
leg.sol.w.in <- max(strwidth(solTypes,'in',leg.cex));
leg.sol.h.in <- max(strheight(solTypes,'in',leg.cex))*1.5; ## multiplication factor for greater line height
leg.join.w.in <- max(strheight(joinTypes,'in',leg.cex))*1.5; ## ditto
leg.join.h.in <- max(strwidth(joinTypes,'in',leg.cex));
leg.main.w.in <- leg.join.w.in*length(joinTypes);
leg.main.h.in <- leg.sol.h.in*length(solTypes);
leg.x2.user <- grconvertX(leg.x.in+leg.outpad.w.in*2+leg.main.w.in+leg.midpad.w.in+leg.sol.w.in,'in');
leg.y2.user <- grconvertY(leg.y.in+leg.outpad.h.in*2+leg.main.h.in+leg.midpad.h.in+leg.join.h.in,'in');
leg.cols.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.join.w.in*(0.5+seq(0L,length(joinTypes)-1L)),'in');
leg.lines.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in-leg.sol.h.in*(0.5+seq(0L,length(solTypes)-1L)),'in');
leg.sol.x.user <- grconvertX(leg.x.in+leg.outpad.w.in+leg.main.w.in+leg.midpad.w.in,'in');
leg.join.y.user <- grconvertY(leg.y.in+leg.outpad.h.in+leg.main.h.in+leg.midpad.h.in,'in');
rect(leg.x.user,leg.y.user,leg.x2.user,leg.y2.user,col='white');
text(leg.sol.x.user,leg.lines.y.user,solTypes[ord],cex=leg.cex,pos=4L,offset=0);
text(leg.cols.x.user,leg.join.y.user,joinTypes,cex=leg.cex,pos=4L,offset=0,srt=90); ## srt rotation applies *after* pos/offset positioning
for (i in seq_along(joinTypes)) {
joinType <- joinTypes[i];
points(rep(leg.cols.x.user[i],length(solTypes)),ifelse(colSums(!is.na(x1[x1$joinType==joinType,solTypes[ord]]))==0L,NA,leg.lines.y.user),pch=pchs[[joinType]],col=cols[solTypes[ord]]);
}; ## end for
title(titleFunc(overlap));
readline(sprintf('overlap %.02f',overlap));
}; ## end for
}; ## end plotRes()
titleFunc <- function(overlap) sprintf('R merge solutions: single-column integer key, 0..1:0..1 cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,T);
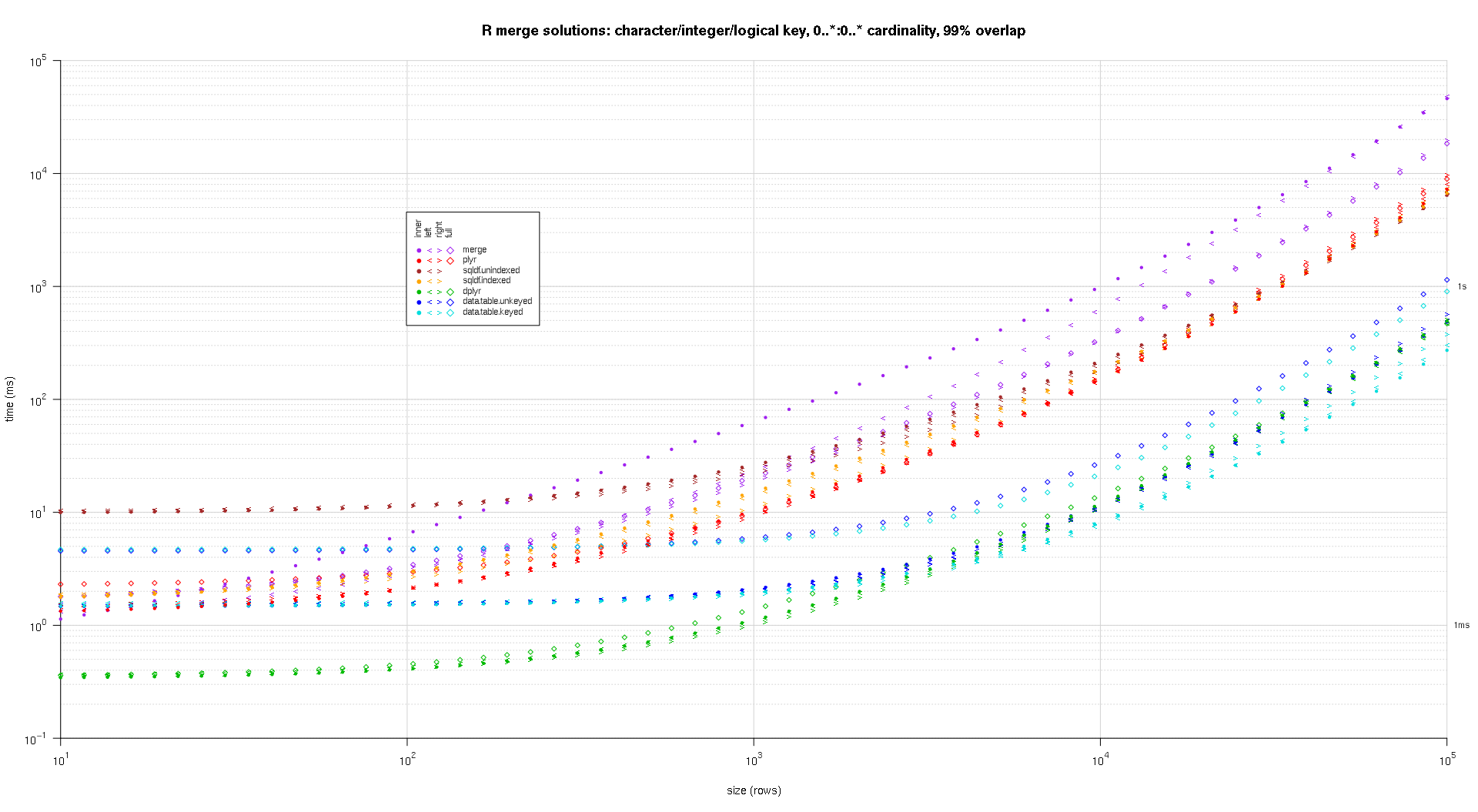
Voici une seconde référence à grande échelle qui est plus robuste, en ce qui concerne le nombre et les types de colonnes clés, ainsi que la cardinalité. Pour ce benchmark j'utilise trois colonnes clés: un caractère, un entier, et un logique, sans restrictions sur la cardinalité (c'est-à-dire, 0..*:0..* ). (En général, il n'est pas conseillé de définir des colonnes clés avec des valeurs doubles ou complexes en raison de complications de comparaison à virgule flottante, et fondamentalement personne n'utilise jamais le type brut, beaucoup moins pour les colonnes clés, donc je n'ai pas incluez ces types dans les colonnes clés. En outre, pour information, j'ai d'abord essayé d'utiliser quatre colonnes clés en incluant une colonne clé POSIXct, mais le type POSIXct ne fonctionnait pas bien avec la solution sqldf.indexed pour une raison quelconque, probablement en raison d'anomalies de comparaison à virgule flottante, donc je l'ai enlevé.)
makeArgSpecs.assortedKey.optionalManyToMany <- function(size,overlap,uniquePct=75) {
## number of unique keys in df1
u1Size <- as.integer(size*uniquePct/100);
## (roughly) divide u1Size into bases, so we can use expand.grid() to produce the required number of unique key values with repetitions within individual key columns
## use ceiling() to ensure we cover u1Size; will truncate afterward
u1SizePerKeyColumn <- as.integer(ceiling(u1Size^(1/3)));
## generate the unique key values for df1
keys1 <- expand.grid(stringsAsFactors=F,
idCharacter=replicate(u1SizePerKeyColumn,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=sample(u1SizePerKeyColumn),
idLogical=sample(c(F,T),u1SizePerKeyColumn,T)
##idPOSIXct=as.POSIXct('2016-01-01 00:00:00','UTC')+sample(u1SizePerKeyColumn)
)[seq_len(u1Size),];
## rbind some repetitions of the unique keys; this will prepare one side of the many-to-many relationship
## also scramble the order afterward
keys1 <- rbind(keys1,keys1[sample(nrow(keys1),size-u1Size,T),])[sample(size),];
## common and unilateral key counts
com <- as.integer(size*overlap);
uni <- size-com;
## generate some unilateral keys for df2 by synthesizing outside of the idInteger range of df1
keys2 <- data.frame(stringsAsFactors=F,
idCharacter=replicate(uni,paste(collapse='',sample(letters,sample(4:12,1L),T))),
idInteger=u1SizePerKeyColumn+sample(uni),
idLogical=sample(c(F,T),uni,T)
##idPOSIXct=as.POSIXct('2016-01-01 00:00:00','UTC')+u1SizePerKeyColumn+sample(uni)
);
## rbind random keys from df1; this will complete the many-to-many relationship
## also scramble the order afterward
keys2 <- rbind(keys2,keys1[sample(nrow(keys1),com,T),])[sample(size),];
##keyNames <- c('idCharacter','idInteger','idLogical','idPOSIXct');
keyNames <- c('idCharacter','idInteger','idLogical');
## note: was going to use raw and complex type for two of the non-key columns, but data.table doesn't seem to fully support them
argSpecs <- list(
default=list(copySpec=1:2,args=list(
df1 <- cbind(stringsAsFactors=F,keys1,y1=sample(c(F,T),size,T),y2=sample(size),y3=rnorm(size),y4=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
df2 <- cbind(stringsAsFactors=F,keys2,y5=sample(c(F,T),size,T),y6=sample(size),y7=rnorm(size),y8=replicate(size,paste(collapse='',sample(letters,sample(4:12,1L),T)))),
keyNames
)),
data.table.unkeyed=list(copySpec=1:2,args=list(
as.data.table(df1),
as.data.table(df2),
keyNames
)),
data.table.keyed=list(copySpec=1:2,args=list(
setkeyv(as.data.table(df1),keyNames),
setkeyv(as.data.table(df2),keyNames)
))
);
## prepare sqldf
initSqldf();
sqldf(paste0('create index df1_key on df1(',paste(collapse=',',keyNames),');')); ## upload and create an sqlite index on df1
sqldf(paste0('create index df2_key on df2(',paste(collapse=',',keyNames),');')); ## upload and create an sqlite index on df2
argSpecs;
}; ## end makeArgSpecs.assortedKey.optionalManyToMany()
sizes <- c(1e1L,1e3L,1e5L); ## 1e5L instead of 1e6L to respect more heavy-duty inputs
overlaps <- c(0.99,0.5,0.01);
solTypes <- setdiff(getSolTypes(),'in.place');
system.time({ res <- testGrid(makeArgSpecs.assortedKey.optionalManyToMany,sizes,overlaps,solTypes); });
## user system elapsed
## 38895.50 784.19 39745.53
les placettes résultantes, en utilisant le même code de pointage que ci-dessus:
titleFunc <- function(overlap) sprintf('R merge solutions: character/integer/logical key, 0..*:0..* cardinality, %d%% overlap',as.integer(overlap*100));
plotRes(res,titleFunc,F);
- en utilisant la fonction
mergenous pouvons sélectionner la variable de la table de gauche ou de droite, de la même manière que nous connaissons tous select statement en SQL (EX: Select a.* ...ou Sélectionnez b.* partir de .....) -
nous devons ajouter un code supplémentaire qui sera un sous-ensemble de la nouvelle table jointe .
-
SQL :-
select a.* from df1 a inner join df2 b on a.CustomerId=b.CustomerId -
R: -
merge(df1, df2, by.x = "CustomerId", by.y = "CustomerId")[,names(df1)]
-
Même façon
-
SQL :-
select b.* from df1 a inner join df2 b on a.CustomerId=b.CustomerId -
R :-
merge(df1, df2, by.x = "CustomerId", by.y = "CustomerId")[,names(df2)]
pour une jonction intérieure sur toutes les colonnes, vous pouvez également utiliser fintersect à partir des données .tableau colis ou intersect de la dplyr -paquet comme une alternative à la merge , sans préciser le by -colonnes. Cela donnera les lignes qui sont égales entre deux images de données:
merge(df1, df2)
# V1 V2
# 1 B 2
# 2 C 3
dplyr::intersect(df1, df2)
# V1 V2
# 1 B 2
# 2 C 3
data.table::fintersect(setDT(df1), setDT(df2))
# V1 V2
# 1: B 2
# 2: C 3
exemples de données:
df1 <- data.frame(V1 = LETTERS[1:4], V2 = 1:4)
df2 <- data.frame(V1 = LETTERS[2:3], V2 = 2:3)
mise à Jour de la rejoindre. une autre jointure importante de style SQL est une " update join " où les colonnes d'une table sont mises à jour (ou créées) en utilisant une autre table.
modifiant les tableaux d'exemples de L'OP...
sales = data.frame(
CustomerId = c(1, 1, 1, 3, 4, 6),
Year = 2000:2005,
Product = c(rep("Toaster", 3), rep("Radio", 3))
)
cust = data.frame(
CustomerId = c(1, 1, 4, 6),
Year = c(2001L, 2002L, 2002L, 2002L),
State = state.name[1:4]
)
sales
# CustomerId Year Product
# 1 2000 Toaster
# 1 2001 Toaster
# 1 2002 Toaster
# 3 2003 Radio
# 4 2004 Radio
# 6 2005 Radio
cust
# CustomerId Year State
# 1 2001 Alabama
# 1 2002 Alaska
# 4 2002 Arizona
# 6 2002 Arkansas
supposons que nous voulons ajouter l'état du client de cust à la table des achats, sales , en ignorant la colonne de l'année. Avec la base R, Nous pouvons identifier les lignes correspondantes et ensuite, copiez les valeurs sur:
sales$State <- cust$State[ match(sales$CustomerId, cust$CustomerId) ]
# CustomerId Year Product State
# 1 2000 Toaster Alabama
# 1 2001 Toaster Alabama
# 1 2002 Toaster Alabama
# 3 2003 Radio <NA>
# 4 2004 Radio Arizona
# 6 2005 Radio Arkansas
# cleanup for the next example
sales$State <- NULL
comme on peut le voir ici, match sélectionne la première ligne correspondante dans la table client.
mise à Jour de jointure avec plusieurs colonnes. l'approche ci-dessus fonctionne bien quand nous rejoignons sur une seule colonne et sommes satisfaits du premier match. Supposons que nous voulions que l'année de mesure dans la table des clients corresponde à l'année de vente.
comme le mentionne la réponse de @bgoldst, match avec interaction pourrait être une option dans ce cas. Plus simplement, on pourrait utiliser les données.tableau:
library(data.table)
setDT(sales); setDT(cust)
sales[, State := cust[sales, on=.(CustomerId, Year), x.State]]
# CustomerId Year Product State
# 1: 1 2000 Toaster <NA>
# 2: 1 2001 Toaster Alabama
# 3: 1 2002 Toaster Alaska
# 4: 3 2003 Radio <NA>
# 5: 4 2004 Radio <NA>
# 6: 6 2005 Radio <NA>
# cleanup for next example
sales[, State := NULL]
mise à jour en cours. alternativement, nous pouvons vouloir prendre le dernier état où le client a été trouvé:
sales[, State := cust[sales, on=.(CustomerId, Year), roll=TRUE, x.State]]
# CustomerId Year Product State
# 1: 1 2000 Toaster <NA>
# 2: 1 2001 Toaster Alabama
# 3: 1 2002 Toaster Alaska
# 4: 3 2003 Radio <NA>
# 5: 4 2004 Radio Arizona
# 6: 6 2005 Radio Arkansas
les trois exemples se concentrent avant tout sur la création/ajout d'une nouvelle colonne. Voir la recherche FAQ pour un exemple de mise à jour/modification d'une colonne existante.