Comment ajuster la taille des étiquettes de l'axe y uniquement en R?
Comment puis-je ajuster seulement la taille des étiquettes de l'axe des Y dans R?
<!-- Je connais ce cex.axis modifie la taille des étiquettes axis mais n'affecte que l'axe X. Pourquoi, et comment puis-je ajuster l'axe des y?3 réponses
ucfagls a raison, à condition que vous utilisiez le plot() la commande. Si non, merci de nous donner plus de détails.
dans tous les cas, vous pouvez contrôler chaque axe séparément en utilisant le axis() le commandement et le xaxt/yaxt options dans plot(). En utilisant les données de ucfagls, cela devient :
plot(Y ~ X, data=foo,yaxt="n")
axis(2,cex.axis=2)
l'option Yaht= " n " est nécessaire pour éviter que la commande plot trace l'axe des ordonnées sans changer. Pour l'axe des x, cela fonctionne exactement de la même chose :
plot(Y ~ X, data=foo,xaxt="n")
axis(1,cex.axis=2)
Voir aussi les fichiers d'aide ?par et ?axe
Edit: comme pour un barplot, regardez les options cex.axe et cex.noms :
tN <- table(sample(letters[1:5],100,replace=T,p=c(0.2,0.1,0.3,0.2,0.2)))
op <- par(mfrow=c(1,2))
barplot(tN, col=rainbow(5),cex.axis=0.5) # for the Y-axis
barplot(tN, col=rainbow(5),cex.names=0.5) # for the X-axis
par(op)
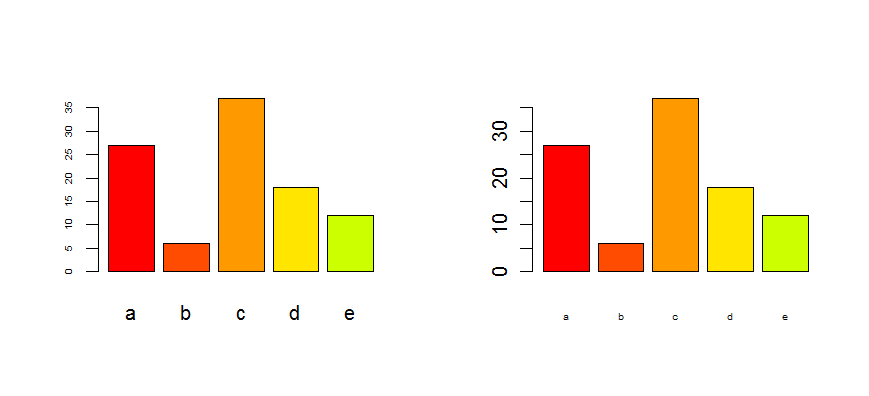
comme le titre suggère que nous voulons ajuster la taille des étiquettes et non les marques de tique, j'ai pensé que je pourrais en fait ajouter quelque chose à la question, vous devez utiliser le mtext() si vous voulez spécifier une des tailles d'étiquette, ou vous pouvez juste utiliser par(cex.lab=2) comme une alternative simple. Voici un exemple de mtext () plus avancé:
set.seed(123)
foo <- data.frame(X = rnorm(10), Y = rnorm(10))
plot(Y ~ X, data=foo,
yaxt="n", ylab="",
xlab="Regular boring x",
pch=16,
col="darkblue")
axis(2,cex.axis=1.2)
mtext("Awesome Y variable", side=2, line=2.2, cex=2)
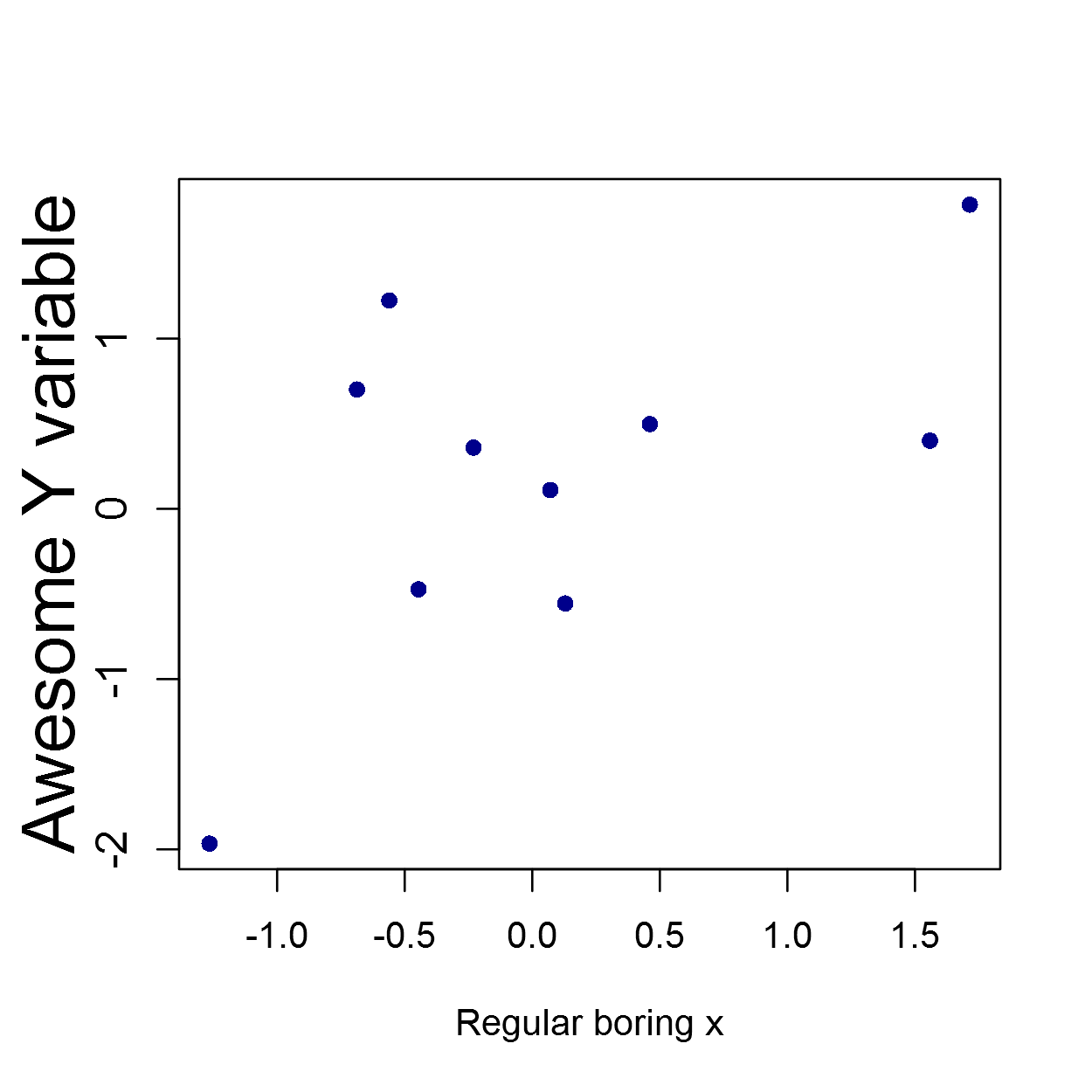
Vous devrez peut-être ajuster l' line= option pour obtenir le positionnement optimal du texte, mais à part ça il est vraiment facile à utiliser.
Je ne sais pas ce que vous faites (utile pour montrer ce que vous avez essayé qui n'a pas fonctionné), mais votre réclamation que cex.axis affecte seulement l'axe des x n'est pas vrai:
set.seed(123)
foo <- data.frame(X = rnorm(10), Y = rnorm(10))
plot(Y ~ X, data = foo, cex.axis = 3)
au moins pour moi:
> sessionInfo()
R version 2.11.1 Patched (2010-08-17 r52767)
Platform: x86_64-unknown-linux-gnu (64-bit)
locale:
[1] LC_CTYPE=en_GB.UTF-8 LC_NUMERIC=C
[3] LC_TIME=en_GB.UTF-8 LC_COLLATE=en_GB.UTF-8
[5] LC_MONETARY=C LC_MESSAGES=en_GB.UTF-8
[7] LC_PAPER=en_GB.UTF-8 LC_NAME=C
[9] LC_ADDRESS=C LC_TELEPHONE=C
[11] LC_MEASUREMENT=en_GB.UTF-8 LC_IDENTIFICATION=C
attached base packages:
[1] grid stats graphics grDevices utils datasets methods
[8] base
other attached packages:
[1] ggplot2_0.8.8 proto_0.3-8 reshape_0.8.3 plyr_1.2.1
loaded via a namespace (and not attached):
[1] digest_0.4.2 tools_2.11.1
Également cex.axis affecte l'étiquetage des marques de graduation. cex.lab est utilisé pour contrôler ce que R appellent les étiquettes axis.
plot(Y ~ X, data = foo, cex.lab = 3)
mais même cela fonctionne pour les axes x et Y.
suite au commentaire de Jens sur l'utilisation barplot(). Découvrez cex.names argument barplot(), qui vous permet de contrôler la barre d'étiquettes:
dat <- rpois (10, 3) noms (dat) < - lettres[1: 10] barplot (dat, cex.noms = 3, cex.axe = 2)
Comme vous l'avez mentionné que cex.axis affectait seulement l'axe des x je présume que vous aviez horiz = TRUE dans votre barplot() appeler ainsi? Comme les étiquettes de barre ne sont pas dessinées avec un axis() appel, l'application de Joris' (sinon, très utile) réponse avec axis() les appels ne serviront à rien situation lorsque vous utilisez barplot()
HTH