Comment remplacer les valeurs de NA par des zéros dans une dataframe R?
14 réponses
Voir mon commentaire dans la réponse @gsk3. Un exemple simple:
> m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
> d <- as.data.frame(m)
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 3 NA 3 7 6 6 10 6 5
2 9 8 9 5 10 NA 2 1 7 2
3 1 1 6 3 6 NA 1 4 1 6
4 NA 4 NA 7 10 2 NA 4 1 8
5 1 2 4 NA 2 6 2 6 7 4
6 NA 3 NA NA 10 2 1 10 8 4
7 4 4 9 10 9 8 9 4 10 NA
8 5 8 3 2 1 4 5 9 4 7
9 3 9 10 1 9 9 10 5 3 3
10 4 2 2 5 NA 9 7 2 5 5
> d[is.na(d)] <- 0
> d
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 3 0 3 7 6 6 10 6 5
2 9 8 9 5 10 0 2 1 7 2
3 1 1 6 3 6 0 1 4 1 6
4 0 4 0 7 10 2 0 4 1 8
5 1 2 4 0 2 6 2 6 7 4
6 0 3 0 0 10 2 1 10 8 4
7 4 4 9 10 9 8 9 4 10 0
8 5 8 3 2 1 4 5 9 4 7
9 3 9 10 1 9 9 10 5 3 3
10 4 2 2 5 0 9 7 2 5 5
il n'est pas nécessaire d'appliquer apply . = )
MODIFIER
vous devriez également jeter un oeil à norm paquet. Il a beaucoup de belles fonctionnalités pour l'analyse de données manquantes. = )
l'hybride dplyr/Base R option: mutate_all(funs(replace(., is.na(.), 0)))) est plus de deux fois plus rapide que la base R d[is.na(d)] <- 0 option. (voir les analyses de référence ci-dessous.)
si vous êtes aux prises avec des images de données massives, data.table est l'option la plus rapide de toutes: 30% moins de temps que dplyr , et 3 fois plus rapide que les approches Base R . Il modifie également les données en place, permettant effectivement vous de travail avec près de deux fois plus de données à la fois.
Un regroupement des autres tidyverse remplacement des approches
Locationally:
- index
mutate_at(c(5:10), funs(replace(., is.na(.), 0))) - référence directe
mutate_at(vars(var5:var10), funs(replace(., is.na(.), 0))) - match fixe
mutate_at(vars(contains("1")), funs(replace(., is.na(.), 0)))- ou à la place de
contains(), essayezends_with(),starts_with()
- ou à la place de
- motif
mutate_at(vars(matches("\d{2}")), funs(replace(., is.na(.), 0)))
conditionnellement:
(changement numérique (colonnes) et laissez la chaîne (colonnes) seul.)
- entiers
mutate_if(is.integer, funs(replace(., is.na(.), 0))) - doubles
mutate_if(is.numeric, funs(replace(., is.na(.), 0))) - cordes
mutate_if(is.character, funs(replace(., is.na(.), 0)))
L'Analyse Complète -
approches testées:
# Base R:
baseR.sbst.rssgn <- function(x) { x[is.na(x)] <- 0; x }
baseR.replace <- function(x) { replace(x, is.na(x), 0) }
baseR.for <- function(x) { for(j in 1:ncol(x))
x[[j]][is.na(x[[j]])] = 0 }
# tidyverse
## dplyr
library(tidyverse)
dplyr_if_else <- function(x) { mutate_all(x, funs(if_else(is.na(.), 0, .))) }
dplyr_coalesce <- function(x) { mutate_all(x, funs(coalesce(., 0))) }
## tidyr
tidyr_replace_na <- function(x) { replace_na(x, as.list(setNames(rep(0, 10), as.list(c(paste0("var", 1:10)))))) }
## hybrid
hybrd.ifelse <- function(x) { mutate_all(x, funs(ifelse(is.na(.), 0, .))) }
hybrd.rplc_all <- function(x) { mutate_all(x, funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.idx<- function(x) { mutate_at(x, c(1:10), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.nse<- function(x) { mutate_at(x, vars(var1:var10), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.stw<- function(x) { mutate_at(x, vars(starts_with("var")), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.ctn<- function(x) { mutate_at(x, vars(contains("var")), funs(replace(., is.na(.), 0))) }
hybrd.rplc_at.mtc<- function(x) { mutate_at(x, vars(matches("\d+")), funs(replace(., is.na(.), 0))) }
hybrd.rplc_if <- function(x) { mutate_if(x, is.numeric, funs(replace(., is.na(.), 0))) }
# data.table
library(data.table)
DT.for.set.nms <- function(x) { for (j in names(x))
set(x,which(is.na(x[[j]])),j,0) }
DT.for.set.sqln <- function(x) { for (j in seq_len(ncol(x)))
set(x,which(is.na(x[[j]])),j,0) }
le code pour cette analyse:
library(microbenchmark)
# 20% NA filled dataframe of 5 Million rows and 10 columns
set.seed(42) # to recreate the exact dataframe
dfN <- as.data.frame(matrix(sample(c(NA, as.numeric(1:4)), 5e6*10, replace = TRUE),
dimnames = list(NULL, paste0("var", 1:10)),
ncol = 10))
# Running 250 trials with each replacement method
# (the functions are excecuted locally - so that the original dataframe remains unmodified in all cases)
perf_results <- microbenchmark(
hybrid.ifelse = hybrid.ifelse(copy(dfN)),
dplyr_if_else = dplyr_if_else(copy(dfN)),
baseR.sbst.rssgn = baseR.sbst.rssgn(copy(dfN)),
baseR.replace = baseR.replace(copy(dfN)),
dplyr_coalesce = dplyr_coalesce(copy(dfN)),
hybrd.rplc_at.nse= hybrd.rplc_at.nse(copy(dfN)),
hybrd.rplc_at.stw= hybrd.rplc_at.stw(copy(dfN)),
hybrd.rplc_at.ctn= hybrd.rplc_at.ctn(copy(dfN)),
hybrd.rplc_at.mtc= hybrd.rplc_at.mtc(copy(dfN)),
hybrd.rplc_at.idx= hybrd.rplc_at.idx(copy(dfN)),
hybrd.rplc_if = hybrd.rplc_if(copy(dfN)),
tidyr_replace_na = tidyr_replace_na(copy(dfN)),
baseR.for = baseR.for(copy(dfN)),
DT.for.set.nms = DT.for.set.nms(copy(dfN)),
DT.for.set.sqln = DT.for.set.sqln(copy(dfN)),
times = 250L
)
résumé des résultats
> perf_results Unit: milliseconds expr min lq mean median uq max neval hybrid.ifelse 5250.5259 5620.8650 5809.1808 5759.3997 5947.7942 6732.791 250 dplyr_if_else 3209.7406 3518.0314 3653.0317 3620.2955 3746.0293 4390.888 250 baseR.sbst.rssgn 1611.9227 1878.7401 1964.6385 1942.8873 2031.5681 2485.843 250 baseR.replace 1559.1494 1874.7377 1946.2971 1920.8077 2002.4825 2516.525 250 dplyr_coalesce 949.7511 1231.5150 1279.3015 1288.3425 1345.8662 1624.186 250 hybrd.rplc_at.nse 735.9949 871.1693 1016.5910 1064.5761 1104.9590 1361.868 250 hybrd.rplc_at.stw 704.4045 887.4796 1017.9110 1063.8001 1106.7748 1338.557 250 hybrd.rplc_at.ctn 723.9838 878.6088 1017.9983 1063.0406 1110.0857 1296.024 250 hybrd.rplc_at.mtc 686.2045 885.8028 1013.8293 1061.2727 1105.7117 1269.949 250 hybrd.rplc_at.idx 696.3159 880.7800 1003.6186 1038.8271 1083.1932 1309.635 250 hybrd.rplc_if 705.9907 889.7381 1000.0113 1036.3963 1083.3728 1338.190 250 tidyr_replace_na 680.4478 973.1395 978.2678 1003.9797 1051.2624 1294.376 250 baseR.for 670.7897 965.6312 983.5775 1001.5229 1052.5946 1206.023 250 DT.for.set.nms 496.8031 569.7471 695.4339 623.1086 861.1918 1067.640 250 DT.for.set.sqln 500.9945 567.2522 671.4158 623.1454 764.9744 1033.463 250
boîte à moustaches des Résultats (sur une échelle log)
# adjust the margins to prepare for better boxplot printing
par(mar=c(8,5,1,1) + 0.1)
# generate boxplot
boxplot(opN, las = 2, xlab = "", ylab = "log(time)[milliseconds]")
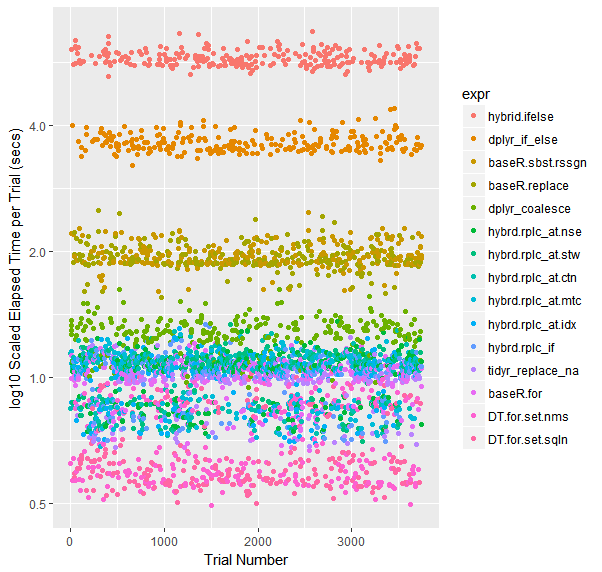
nuage d'Essais codé en couleur (sur une échelle logarithmique)
qplot(y=time/10^9, data=opN, colour=expr) +
labs(y = "log10 Scaled Elapsed Time per Trial (secs)", x = "Trial Number") +
scale_y_log10(breaks=c(1, 2, 4))
note sur les autres grands artistes
quand les ensembles de données deviennent plus grands, Tidyr "s replace_na avait historiquement tiré en avant. Avec la collecte actuelle de points de données 50M à parcourir, il fonctionne presque aussi bien qu'une Base R pour boucle. Je suis curieux de voir ce qui se passe pour les différentes tailles d'dataframes.
exemples supplémentaires pour les variantes de fonctions mutate et summarize _at et _all peuvent être trouvé ici: https://rdrr.io/cran/dplyr/man/summarise_all.html
En outre, j'ai trouvé des démonstrations utiles et des collections d'exemples ici: https://blog.exploratory.io/dplyr-0-5-is-awesome-heres-why-be095fd4eb8a
Attributions et appréciations
avec remerciements spéciaux à:
- Tyler Rinker et Akrun pour démontrer microbenchmark.
- alexis_laz pour m'avoir aidé à comprendre l'utilisation de
local(), et (avec L'aide du patient de Frank, aussi) le rôle que la coercition silencieuse joue dans l'accélération de plusieurs de ces approches. - ArthurYip pour le poke pour ajouter la nouvelle
coalesce()de la fonction et de mettre à jour l'analyse. - Gregor pour le coup de coude de comprendre l'
data.tablefonctionne assez bien pour enfin les inclure dans la gamme. - Base R pour boucle: alexis_laz
- "1519270920 des données".tableau pour boucles: Matt_Dowle
(bien sûr, s'il vous plaît tendez la main et donnez-leur upvotes, aussi si vous trouvez ces approches utiles.)
Note sur mon utilisation de Numerics: si vous avez un ensemble de données entier pur, toutes vos fonctions s'exécuteront plus rapidement. Voir l'œuvre d'alexiz_laz pour plus d'informations. IRL, Je ne me souviens pas avoir rencontré un ensemble de données contenant plus de 10-15% d'entiers, donc j'exécute ces tests sur des images de données entièrement numériques.
pour un vecteur unique:
x <- c(1,2,NA,4,5)
x[is.na(x)] <- 0
pour les données a.frame, faire une fonction à partir de ce qui précède, puis apply il aux colonnes.
, Veuillez fournir un exemple reproductible prochaine fois que détaillé ici:
exemple dplyr:
library(dplyr)
df1 <- df1 %>%
mutate(myCol1 = if_else(is.na(myCol1), 0, myCol1))
Note: cela fonctionne par colonne sélectionnée, si nous devons faire cela pour toute colonne, voir @reidjax réponse de mutate_each .
si nous essayons de remplacer NA s lors de l'exportation, par exemple en écrivant à csv, alors nous pouvons utiliser:
write.csv(data, "data.csv", na = "0")
je sais que la question est déjà résolue, mais le faire de cette façon pourrait être plus utile pour certains:
définit cette fonction:
na.zero <- function (x) {
x[is.na(x)] <- 0
return(x)
}
maintenant chaque fois que vous avez besoin de convertir NA dans un vecteur à zéro, vous pouvez faire:
na.zero(some.vector)
méthode plus générale consistant à utiliser replace() dans la matrice ou le vecteur pour remplacer NA par 0
par exemple:
> x <- c(1,2,NA,NA,1,1)
> x1 <- replace(x,is.na(x),0)
> x1
[1] 1 2 0 0 1 1
il s'agit également d'une alternative à l'utilisation de ifelse() dans dplyr
df = data.frame(col = c(1,2,NA,NA,1,1))
df <- df %>%
mutate(col = replace(col,is.na(col),0))
avec dplyr 0.5.0, vous pouvez utiliser la fonction coalesce qui peut être facilement intégrée dans %>% pipeline en faisant coalesce(vec, 0) . Ceci remplace tous les NAs dans vec par 0:
disons que nous avons une base de données avec NA s:
library(dplyr)
df <- data.frame(v = c(1, 2, 3, NA, 5, 6, 8))
df
# v
# 1 1
# 2 2
# 3 3
# 4 NA
# 5 5
# 6 6
# 7 8
df %>% mutate(v = coalesce(v, 0))
# v
# 1 1
# 2 2
# 3 3
# 4 0
# 5 5
# 6 6
# 7 8
un autre exemple utilisant imputeTS paquet:
library(imputeTS)
na.replace(yourDataframe, 0)
si vous voulez remplacer le NAs dans les variables de facteurs, cela peut être utile:
n <- length(levels(data.vector))+1
data.vector <- as.numeric(data.vector)
data.vector[is.na(data.vector)] <- n
data.vector <- as.factor(data.vector)
levels(data.vector) <- c("level1","level2",...,"leveln", "NAlevel")
il transforme un vecteur de facteur en vecteur numérique et ajoute un autre niveau de facteur numérique artificiel, qui est ensuite transformé à nouveau à un vecteur de facteur avec un" NA-niveau " supplémentaire de votre choix.
aurait commenté le billet de @ianmunoz mais je n'ai pas assez de réputation. Vous pouvez combiner dplyr 's mutate_each et replace pour prendre soin du NA à 0 remplacement. En utilisant le dataframe de la réponse de @aL3xa...
> m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
> d <- as.data.frame(m)
> d
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 8 1 9 6 9 NA 8 9 8
2 8 3 6 8 2 1 NA NA 6 3
3 6 6 3 NA 2 NA NA 5 7 7
4 10 6 1 1 7 9 1 10 3 10
5 10 6 7 10 10 3 2 5 4 6
6 2 4 1 5 7 NA NA 8 4 4
7 7 2 3 1 4 10 NA 8 7 7
8 9 5 8 10 5 3 5 8 3 2
9 9 1 8 7 6 5 NA NA 6 7
10 6 10 8 7 1 1 2 2 5 7
> d %>% mutate_each( funs_( interp( ~replace(., is.na(.),0) ) ) )
V1 V2 V3 V4 V5 V6 V7 V8 V9 V10
1 4 8 1 9 6 9 0 8 9 8
2 8 3 6 8 2 1 0 0 6 3
3 6 6 3 0 2 0 0 5 7 7
4 10 6 1 1 7 9 1 10 3 10
5 10 6 7 10 10 3 2 5 4 6
6 2 4 1 5 7 0 0 8 4 4
7 7 2 3 1 4 10 0 8 7 7
8 9 5 8 10 5 3 5 8 3 2
9 9 1 8 7 6 5 0 0 6 7
10 6 10 8 7 1 1 2 2 5 7
nous utilisons l'évaluation standard (SE) ici, c'est pourquoi nous avons besoin du soulignement sur " funs_ ."Nous utilisons aussi lazyeval ' s interp / ~ et les références . "tout ce avec quoi nous travaillons", c'est-à-dire la base de données. Maintenant, il y a de zéros!
vous pouvez utiliser replace()
par exemple:
> x <- c(-1,0,1,0,NA,0,1,1)
> x1 <- replace(x,5,1)
> x1
[1] -1 0 1 0 1 0 1 1
> x1 <- replace(x,5,mean(x,na.rm=T))
> x1
[1] -1.00 0.00 1.00 0.00 0.29 0.00 1.00 1.00
l'Autre dplyr pipe compatibles en option avec tidyr méthode replace_na qui fonctionne pour plusieurs colonnes:
require(dplyr)
require(tidyr)
m <- matrix(sample(c(NA, 1:10), 100, replace = TRUE), 10)
d <- as.data.frame(m)
myList <- setNames(lapply(vector("list", ncol(d)), function(x) x <- 0), names(d))
df <- d %>% replace_na(myList)
vous pouvez facilement restreindre à des colonnes numériques par exemple:
d$str <- c("string", NA)
myList <- myList[sapply(d, is.numeric)]
df <- d %>% replace_na(myList)
cette fonction simple extraite de Datacamp pourrait aider:
replace_missings <- function(x, replacement) {
is_miss <- is.na(x)
x[is_miss] <- replacement
message(sum(is_miss), " missings replaced by the value ", replacement)
x
}
puis
replace_missings(df, replacement = 0)