Comment puis-je tracer avec 2 axes y différents?
Je voudrais superposer deux diagrammes de dispersion dans R de sorte que chaque ensemble de points ait son propre axe y (différent) (c'est-à-dire dans les positions 2 et 4 sur la figure) mais les points apparaissent superposés sur la même figure.
Est-il possible de le faire avec plot?
Modifier {[8] } Exemple de code montrant le problème
# example code for SO question
y1 <- rnorm(10, 100, 20)
y2 <- rnorm(10, 1, 1)
x <- 1:10
# in this plot y2 is plotted on what is clearly an inappropriate scale
plot(y1 ~ x, ylim = c(-1, 150))
points(y2 ~ x, pch = 2)
6 réponses
Mise à jour : copié le matériel qui était sur le wiki R à http://rwiki.sciviews.org/doku.php?id=tips:graphics-base:2yaxes , lien maintenant cassé: également disponible à partir de la machine wayback
Deux axes y différents sur le même tracé
(certains matériaux à L'origine par Daniel Rajdl 2006/03/31 15: 26)
Veuillez noter qu'il existe très peu de situations où il est approprié d'utiliser deux échelles différentes sur la même parcelle. Il est très facile d'induire en erreur le spectateur de l'image. Vérifiez les deux exemples et commentaires suivants sur ce problème (example1, example2 de Junk Charts), ainsi que cet article de Stephen Few (qui conclut "Je ne peux certainement pas conclure, une fois pour toutes, que les graphiques avec des axes à double échelle ne sont jamais utiles; seulement que je ne peux pas penser à une situation qui les justifie à la lumière d'autres solutions meilleures.") Voir aussi le point # 4 dans Ce dessin animé ...
Si vous sont déterminés, la recette de base est de créer votre premier tracé, définir par(new=TRUE) pour empêcher R d'effacer le périphérique graphique, créer le deuxième tracé avec axes=FALSE (et définir xlab et ylab pour être vide – ann=FALSE devrait également fonctionner), puis utiliser axis(side=4) pour ajouter un nouvel axe sur le côté droit, et mtext(...,side=4) pour ajouter une étiquette Voici un exemple utilisant un peu de données inventées:
set.seed(101)
x <- 1:10
y <- rnorm(10)
## second data set on a very different scale
z <- runif(10, min=1000, max=10000)
par(mar = c(5, 4, 4, 4) + 0.3) # Leave space for z axis
plot(x, y) # first plot
par(new = TRUE)
plot(x, z, type = "l", axes = FALSE, bty = "n", xlab = "", ylab = "")
axis(side=4, at = pretty(range(z)))
mtext("z", side=4, line=3)
twoord.plot() dans le paquet plotrix automatise ce processus, comme le fait doubleYScale() dans le latticeExtra paquet.
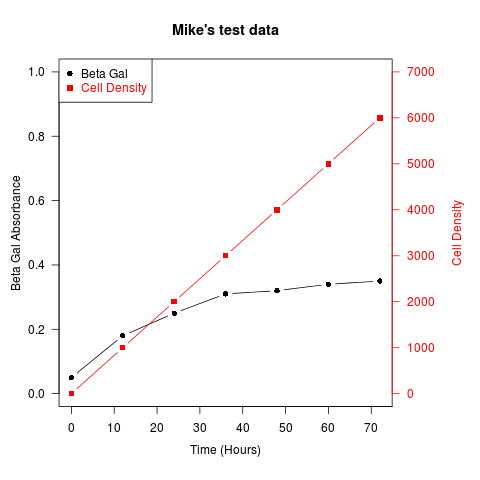
Un autre exemple (adapté d'un message de R mailing list DE Robert W. Baer):
## set up some fake test data
time <- seq(0,72,12)
betagal.abs <- c(0.05,0.18,0.25,0.31,0.32,0.34,0.35)
cell.density <- c(0,1000,2000,3000,4000,5000,6000)
## add extra space to right margin of plot within frame
par(mar=c(5, 4, 4, 6) + 0.1)
## Plot first set of data and draw its axis
plot(time, betagal.abs, pch=16, axes=FALSE, ylim=c(0,1), xlab="", ylab="",
type="b",col="black", main="Mike's test data")
axis(2, ylim=c(0,1),col="black",las=1) ## las=1 makes horizontal labels
mtext("Beta Gal Absorbance",side=2,line=2.5)
box()
## Allow a second plot on the same graph
par(new=TRUE)
## Plot the second plot and put axis scale on right
plot(time, cell.density, pch=15, xlab="", ylab="", ylim=c(0,7000),
axes=FALSE, type="b", col="red")
## a little farther out (line=4) to make room for labels
mtext("Cell Density",side=4,col="red",line=4)
axis(4, ylim=c(0,7000), col="red",col.axis="red",las=1)
## Draw the time axis
axis(1,pretty(range(time),10))
mtext("Time (Hours)",side=1,col="black",line=2.5)
## Add Legend
legend("topleft",legend=c("Beta Gal","Cell Density"),
text.col=c("black","red"),pch=c(16,15),col=c("black","red"))
Des recettes similaires peuvent être utilisées pour superposer des tracés de différents types – tracés à barres, histogrammes, etc..
Une option consiste à faire deux parcelles côte à côte. ggplot2 fournit une bonne option pour cette avec facet_wrap():
dat <- data.frame(x = c(rnorm(100), rnorm(100, 10, 2))
, y = c(rnorm(100), rlnorm(100, 9, 2))
, index = rep(1:2, each = 100)
)
require(ggplot2)
ggplot(dat, aes(x,y)) +
geom_point() +
facet_wrap(~ index, scales = "free_y")
C'est une FAQ. Voici une solution plus ancienne que j'ai fournie presque Il y a six ans à la galerie R Graph
Vous pouvez regarder par exemple la fonction plotVolumeBars() qui combine une échelle absolue et une échelle relative dans un graphique.
Si vous pouvez abandonner les étiquettes échelles/axes, vous pouvez redimensionner les données à (0, 1) intervalle. Cela fonctionne par exemple pour différents trakcs "wiggle" sur les chromosomes, lorsque vous êtes généralement intéressé par les corrélations locales entre les pistes et qu'ils ont des échelles différentes (couverture en milliers, Fst 0-1).
# rescale numeric vector into (0, 1) interval
# clip everything outside the range
rescale <- function(vec, lims=range(vec), clip=c(0, 1)) {
# find the coeficients of transforming linear equation
# that maps the lims range to (0, 1)
slope <- (1 - 0) / (lims[2] - lims[1])
intercept <- - slope * lims[1]
xformed <- slope * vec + intercept
# do the clipping
xformed[xformed < 0] <- clip[1]
xformed[xformed > 1] <- clip[2]
xformed
}
, Puis, ayant un bloc de données chrom, position, coverage et fst colonnes, vous pouvez faire quelque chose comme:
ggplot(d, aes(position)) +
geom_line(aes(y = rescale(fst))) +
geom_line(aes(y = rescale(coverage))) +
facet_wrap(~chrom)
L'avantage de ceci est que vous n'êtes pas limité à deux trakcs.
Je suggère aussi, twoord.stackplot() dans les tracés de paquets plotrix avec plus de deux axes d'ordonnées.
data<-read.table(text=
"e0AL fxAL e0CO fxCO e0BR fxBR anos
51.8 5.9 50.6 6.8 51.0 6.2 1955
54.7 5.9 55.2 6.8 53.5 6.2 1960
57.1 6.0 57.9 6.8 55.9 6.2 1965
59.1 5.6 60.1 6.2 57.9 5.4 1970
61.2 5.1 61.8 5.0 59.8 4.7 1975
63.4 4.5 64.0 4.3 61.8 4.3 1980
65.4 3.9 66.9 3.7 63.5 3.8 1985
67.3 3.4 68.0 3.2 65.5 3.1 1990
69.1 3.0 68.7 3.0 67.5 2.6 1995
70.9 2.8 70.3 2.8 69.5 2.5 2000
72.4 2.5 71.7 2.6 71.1 2.3 2005
73.3 2.3 72.9 2.5 72.1 1.9 2010
74.3 2.2 73.8 2.4 73.2 1.8 2015
75.2 2.0 74.6 2.3 74.2 1.7 2020
76.0 2.0 75.4 2.2 75.2 1.6 2025
76.8 1.9 76.2 2.1 76.1 1.6 2030
77.6 1.9 76.9 2.1 77.1 1.6 2035
78.4 1.9 77.6 2.0 77.9 1.7 2040
79.1 1.8 78.3 1.9 78.7 1.7 2045
79.8 1.8 79.0 1.9 79.5 1.7 2050
80.5 1.8 79.7 1.9 80.3 1.7 2055
81.1 1.8 80.3 1.8 80.9 1.8 2060
81.7 1.8 80.9 1.8 81.6 1.8 2065
82.3 1.8 81.4 1.8 82.2 1.8 2070
82.8 1.8 82.0 1.7 82.8 1.8 2075
83.3 1.8 82.5 1.7 83.4 1.9 2080
83.8 1.8 83.0 1.7 83.9 1.9 2085
84.3 1.9 83.5 1.8 84.4 1.9 2090
84.7 1.9 83.9 1.8 84.9 1.9 2095
85.1 1.9 84.3 1.8 85.4 1.9 2100", header=T)
require(plotrix)
twoord.stackplot(lx=data$anos, rx=data$anos,
ldata=cbind(data$e0AL, data$e0BR, data$e0CO),
rdata=cbind(data$fxAL, data$fxBR, data$fxCO),
lcol=c("black","red", "blue"),
rcol=c("black","red", "blue"),
ltype=c("l","o","b"),
rtype=c("l","o","b"),
lylab="Años de Vida", rylab="Hijos x Mujer",
xlab="Tiempo",
main="Mortalidad/Fecundidad:1950–2100",
border="grey80")
legend("bottomright", c(paste("Proy:",
c("A. Latina", "Brasil", "Colombia"))), cex=1,
col=c("black","red", "blue"), lwd=2, bty="n",
lty=c(1,1,2), pch=c(NA,1,1) )