Comment puis-je superposer deux diagrammes de dispersion dense pour voir les contours de chacun dans R ou Matlab?
Voir cet exemple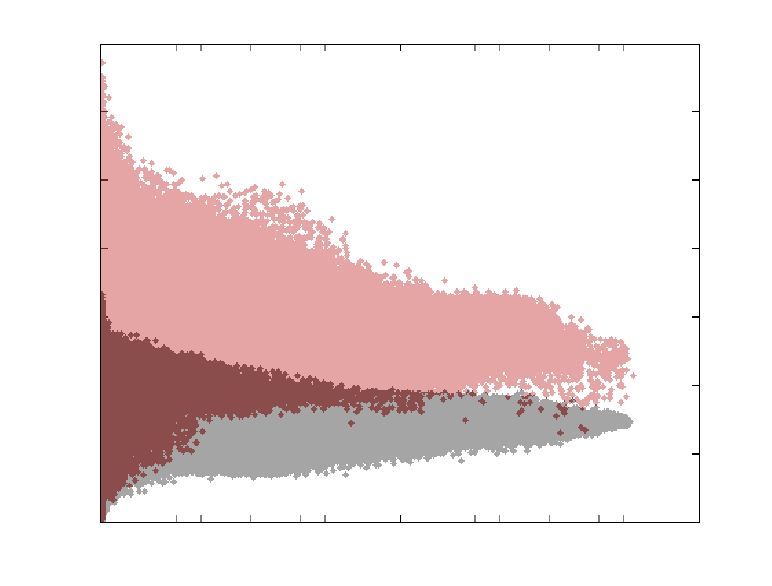
ceci a été créé dans matlab en faisant deux diagrammes de dispersion indépendamment, en créant des images de chacun, puis en utilisant l'imagesc pour les attirer dans la même figure et enfin en réglant l'alpha de l'image supérieure à 0.5.
je voudrais le faire en R ou matlab sans utiliser d'images, car la création d'une image ne préserve pas l'information de l'échelle de l'axe, et je ne peux pas non plus superposer une grille (par exemple en utilisant 'grid on' dans matlab). Idéalement, je wold comme pour le faire correctement dans matlab, mais serait également heureux avec une solution en R. Il semble que cela devrait être possible, mais je ne peux pas pour la vie de moi de le comprendre.
donc en général, je voudrais être capable de définir l'alpha d'un objet entier tracé (c.-à-d. d'une poignée de parcelle matlab dans le jargon matlab...)
Merci,
Ben.
modifier: les données dans l'exemple ci-dessus sont en fait 2D. Les points tracés proviennent d'une simulation par ordinateur. Chaque point représente 'amplitude' (Y-axis) (une propriété émergente spécifique à la simulation que j'exécute), tracée en fonction de 'performance' (x-axis).
EDIT 2: Il y a 1796400 points dans chaque ensemble de données.
4 réponses
en utilisant ggplot2 vous pouvez ajouter deux geom_point's et les rendre transparents en utilisant le alpha paramètre. ggplot2 sla ajoute de la transparence, et je pense que c'est ce que vous voulez. Ça devrait marcher, même si je n'ai pas vérifié.
dat = data.frame(x = runif(1000), y = runif(1000), cat = rep(c("A","B"), each = 500))
ggplot(aes(x = x, y = y, color = cat), data = dat) + geom_point(alpha = 0.3)
ggplot2 est génial!
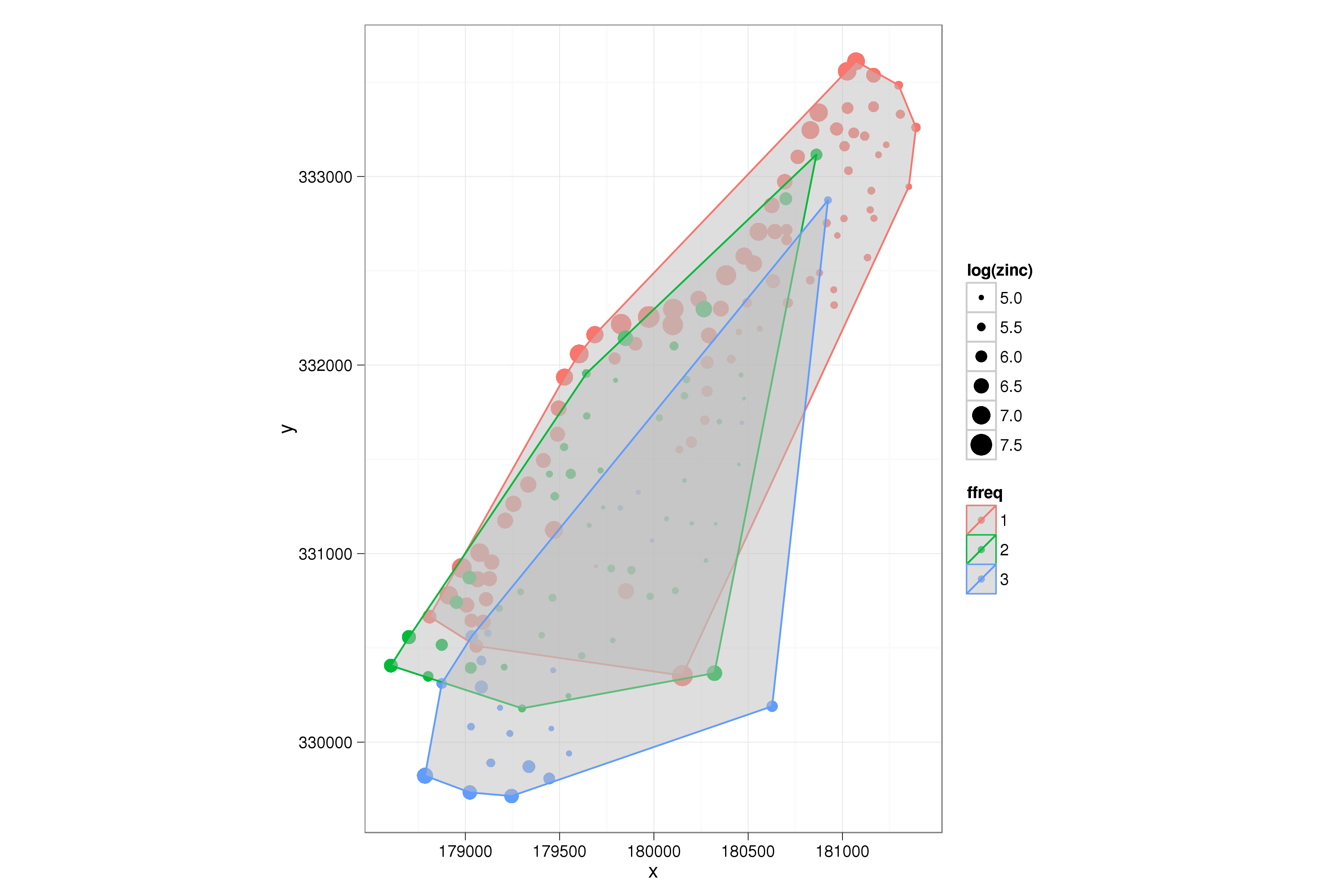
ceci est un exemple de calcul et de dessin d'une coque convexe:
library(automap)
library(ggplot2)
library(plyr)
loadMeuse()
theme_set(theme_bw())
meuse = as.data.frame(meuse)
chull_per_soil = ddply(meuse, .(soil),
function(sub) sub[chull(sub$x, sub$y),c("x","y")])
ggplot(aes(x = x, y = y), data = meuse) +
geom_point(aes(size = log(zinc), color = ffreq)) +
geom_polygon(aes(color = soil), data = chull_per_soil, fill = NA) +
coord_equal()
ce qui conduit à l'illustration suivante:
vous pouvez d'abord exporter les deux ensembles de données en images bitmap, les réimporter, ajouter de la transparence:
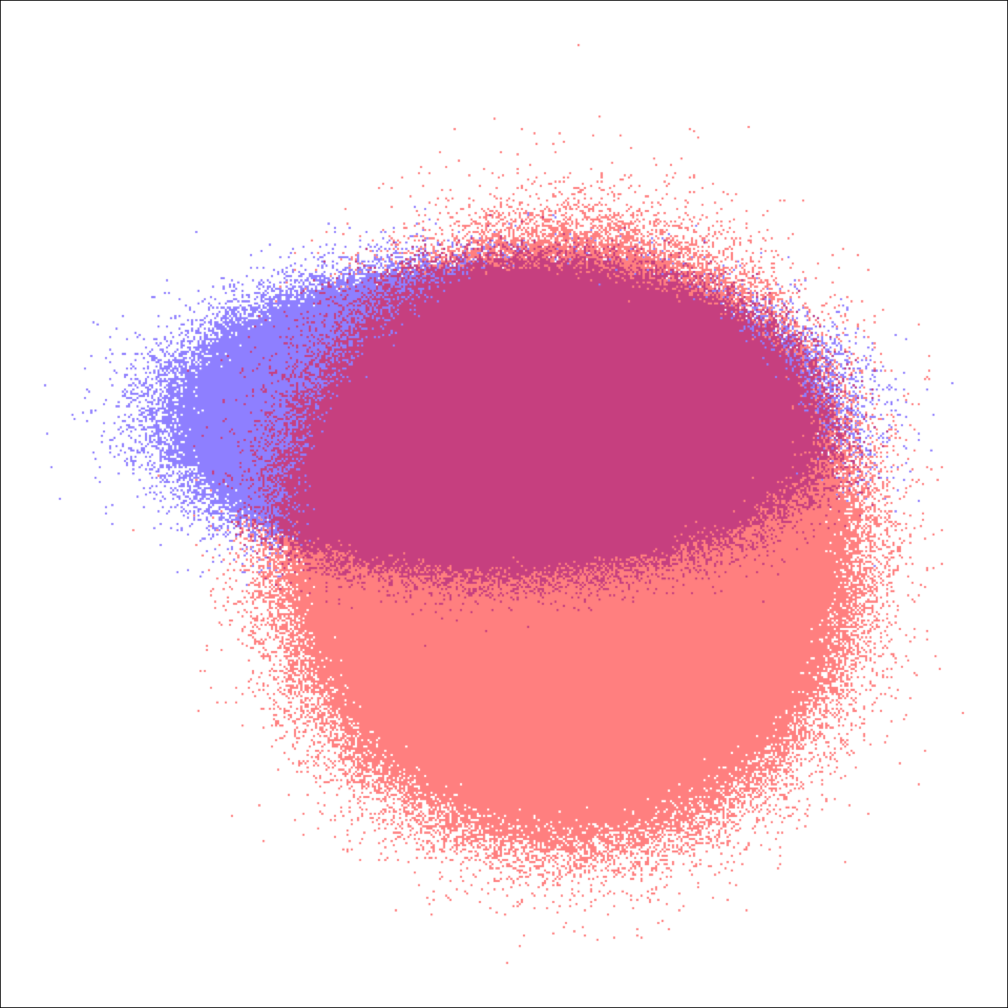
library(grid)
N <- 1e7 # Warning: slow
d <- data.frame(x1=rnorm(N),
x2=rnorm(N, 0.8, 0.9),
y=rnorm(N, 0.8, 0.2),
z=rnorm(N, 0.2, 0.4))
v <- with(d, dataViewport(c(x1,x2),c(y, z)))
png("layer1.png", bg="transparent")
with(d, grid.points(x1,y, vp=v,default="native",pch=".",gp=gpar(col="blue")))
dev.off()
png("layer2.png", bg="transparent")
with(d, grid.points(x2,z, vp=v,default="native",pch=".",gp=gpar(col="red")))
dev.off()
library(png)
i1 <- readPNG("layer1.png", native=FALSE)
i2 <- readPNG("layer2.png", native=FALSE)
ghostize <- function(r, alpha=0.5)
matrix(adjustcolor(rgb(r[,,1],r[,,2],r[,,3],r[,,4]), alpha.f=alpha), nrow=dim(r)[1])
grid.newpage()
grid.rect(gp=gpar(fill="white"))
grid.raster(ghostize(i1))
grid.raster(ghostize(i2))
vous pouvez ajouter ces couches, par exemple, ggplot2.
Utilisez la capacité de transparence des descriptions de couleurs. Vous pouvez définir une couleur comme une séquence de quatre mots de 2 octets: muddy <- "#888888FF". Les trois premières paires fixent les couleurs RGB (00 à FF); la dernière paire fixe le niveau de transparence.
AFAIK, votre meilleure option avec Matlab est de faire votre propre fonction d'intrigue. Les points scatter plot n'ont malheureusement pas encore d'attribut de transparence donc vous ne pouvez pas l'affecter. Toutefois, si vous créez, disons, plus grossièrement, un tas de boucles qui attirent beaucoup de petits cercles, vous pouvez facilement leur donner une valeur alpha et obtenir un ensemble transparent de points de données.