Fonctions de regroupement (tapply, by, aggregate) et la famille d'application
chaque fois que je veux faire quelque chose "map"py in R, j'essaie habituellement d'utiliser une fonction dans la famille apply .
cependant, je n'ai jamais tout à fait compris les différences entre eux -- comment { sapply , lapply , etc.} appliquer la fonction à l'entrée/entrée groupée, à quoi ressemblera la sortie, ou même ce que l'entrée peut être -- donc je les passe souvent en revue jusqu'à ce que j'obtienne ce que je veux.
est-ce que quelqu'un peut expliquer comment utiliser qui un quand?
ma compréhension actuelle (probablement incorrecte/incomplète) est...
-
sapply(vec, f): l'entrée est un vecteur. la sortie est un vecteur / matrice, où l'élémentiestf(vec[i]), vous donnant une matrice sifa une sortie multi-éléments -
lapply(vec, f): commesapply, mais la sortie est une liste? -
apply(matrix, 1/2, f): en entrée une matrice. la sortie est un vecteur, où l'élémentiest f (ligne/colonne i de la matrice) -
tapply(vector, grouping, f): sortie est une matrice / tableau, où un élément dans la matrice / tableau est la valeur defà un groupementgdu vecteur, etgest poussé aux noms de ligne / colonne -
by(dataframe, grouping, f): quegsoit un groupement. appliquerfà chaque colonne du groupe/dataframe. jolie impression de la le regroupement et la valeur defà chaque colonne. -
aggregate(matrix, grouping, f): semblable àby, mais au lieu d'Imprimer la sortie, aggregate colle tout dans une base de données.
question secondaire: je n'ai toujours pas appris le plyr ou le remodelage -- est-ce que plyr ou reshape remplacerait tout cela entièrement?
9 réponses
r a beaucoup de fonctions *apply qui sont habilement décrites dans les fichiers d'aide (par exemple ?apply ). Il y en a assez, cependant, pour que les utilisateurs débutants puissent avoir de la difficulté à décider lequel est approprié à leur situation ou même à se les rappeler tous. Ils peuvent avoir le sentiment général que "je devrais utiliser une fonction * apply ici", mais il peut être difficile de les garder tous droits au début.
malgré le fait (noté dans d'autres réponses) que la plupart des la fonctionnalité de la famille * apply est couverte par le très populaire paquet plyr , les fonctions de base restent utiles et valent la peine d'être connues.
cette réponse est destinée à agir comme une sorte de signpost pour les nouveaux utilisateurs pour les aider à les diriger vers la fonction correcte *appliquer pour leur problème particulier. Notez que c'est et non destiné à simplement régurgiter ou remplacer la documentation r! L'espoir est que cette réponse aide vous décidez quelle fonction * apply convient à votre situation et c'est à vous de la rechercher davantage. À une exception près, les écarts de rendement ne seront pas abordés.
-
appliquer - quand vous voulez appliquer une fonction aux lignes ou aux colonnes d'une matrice (et des analogues de dimensions plus élevées); généralement déconseillé pour les bases de données car il sera d'abord contraint à une matrice.
# Two dimensional matrix M <- matrix(seq(1,16), 4, 4) # apply min to rows apply(M, 1, min) [1] 1 2 3 4 # apply max to columns apply(M, 2, max) [1] 4 8 12 16 # 3 dimensional array M <- array( seq(32), dim = c(4,4,2)) # Apply sum across each M[*, , ] - i.e Sum across 2nd and 3rd dimension apply(M, 1, sum) # Result is one-dimensional [1] 120 128 136 144 # Apply sum across each M[*, *, ] - i.e Sum across 3rd dimension apply(M, c(1,2), sum) # Result is two-dimensional [,1] [,2] [,3] [,4] [1,] 18 26 34 42 [2,] 20 28 36 44 [3,] 22 30 38 46 [4,] 24 32 40 48Si vous voulez ligne/colonne ou sommes pour une matrice 2D, assurez-vous de enquêtez sur le très optimisé, lightning-quick
colMeans,rowMeans,colSums,rowSums. -
lapply - quand vous voulez appliquer une fonction à chaque élément d'un la liste tour et d'obtenir une liste de retour.
C'est la bête de somme de beaucoup d'autres * apply fonction. Peel retournez leur code et vous trouverez souvent
lapplyci-dessous.x <- list(a = 1, b = 1:3, c = 10:100) lapply(x, FUN = length) $a [1] 1 $b [1] 3 $c [1] 91 lapply(x, FUN = sum) $a [1] 1 $b [1] 6 $c [1] 5005 -
sapply - quand vous voulez appliquer une fonction à chaque élément d'un liste à son tour, mais vous voulez un vecteur plutôt que d'une liste.
si vous tapez
unlist(lapply(...)), arrêtez et considérezsapply.x <- list(a = 1, b = 1:3, c = 10:100) # Compare with above; a named vector, not a list sapply(x, FUN = length) a b c 1 3 91 sapply(x, FUN = sum) a b c 1 6 5005dans les utilisations plus avancées de
sapplyil tentera de contraindre le suite à un tableau multidimensionnel, le cas échéant. Par exemple, si notre fonction renvoie des vecteurs de même longueur,sapplyles utilisera comme colonnes d'une matrice:sapply(1:5,function(x) rnorm(3,x))si notre fonction retourne une matrice bidimensionnelle,
sapplyfera essentiellement la même chose, traitant chaque matrice retournée comme un seul vecteur long:sapply(1:5,function(x) matrix(x,2,2))Sauf si nous spécifions
simplify = "array", auquel cas il utilisera les matrices individuelles pour construire un tableau multidimensionnel:sapply(1:5,function(x) matrix(x,2,2), simplify = "array")chacun de ces comportements dépend évidemment de notre fonction retournant des vecteurs ou des matrices de la même longueur ou dimension.
-
vapply - quand vous voulez utiliser
sapplymais peut-être besoin de sors un peu plus de vitesse de ton code.pour
vapply, vous donnez essentiellement un exemple de ce genre de chose votre fonction reviendra, ce qui peut vous faire gagner du temps. valeurs à ajuster dans un vecteur atomique unique.x <- list(a = 1, b = 1:3, c = 10:100) #Note that since the advantage here is mainly speed, this # example is only for illustration. We're telling R that # everything returned by length() should be an integer of # length 1. vapply(x, FUN = length, FUN.VALUE = 0L) a b c 1 3 91 -
mapply - pour quand vous avez plusieurs structures de données (par ex. vecteurs, listes) et vous voulez appliquer une fonction aux 1er éléments de chacun, et puis la 2ème éléments de chacun, etc., le fait de contraindre le résultat à un vecteur / tableau comme dans
sapply.c'est MultiVariable dans le sens où votre fonction doit accepter arguments multiples.
#Sums the 1st elements, the 2nd elements, etc. mapply(sum, 1:5, 1:5, 1:5) [1] 3 6 9 12 15 #To do rep(1,4), rep(2,3), etc. mapply(rep, 1:4, 4:1) [[1]] [1] 1 1 1 1 [[2]] [1] 2 2 2 [[3]] [1] 3 3 [[4]] [1] 4 -
Carte - "1519550920 Un" wrapper
mapplyavecSIMPLIFY = FALSE, de sorte qu'il est garanti pour revenir une liste.Map(sum, 1:5, 1:5, 1:5) [[1]] [1] 3 [[2]] [1] 6 [[3]] [1] 9 [[4]] [1] 12 [[5]] [1] 15 -
rapply - Pour lorsque vous souhaitez appliquer une fonction à chaque élément d'un liste imbriquée de la structure, de manière récursive.
pour vous donner une idée à quel point
rapplyest rare, je l'ai oublié lors de la première affichage de cette réponse! Évidemment, je suis sûr que beaucoup de gens l'utilisent, mais YMMV.rapplyest le mieux illustré par une fonction définie par l'utilisateur à appliquer:# Append ! to string, otherwise increment myFun <- function(x){ if(is.character(x)){ return(paste(x,"!",sep="")) } else{ return(x + 1) } } #A nested list structure l <- list(a = list(a1 = "Boo", b1 = 2, c1 = "Eeek"), b = 3, c = "Yikes", d = list(a2 = 1, b2 = list(a3 = "Hey", b3 = 5))) # Result is named vector, coerced to character rapply(l, myFun) # Result is a nested list like l, with values altered rapply(l, myFun, how="replace") -
tapply - Pour lorsque vous souhaitez appliquer une fonction de sous-ensembles d'un le vecteur et les sous-ensembles sont définis par un autre vecteur, généralement un facteur.
le mouton noir de la *appliquer la famille, en quelque sorte. Le fichier d'aide de l'utilisation de l'expression "Ragged array" peut être un peu déroutant , mais il est en fait tout à fait simple.
d'Un vecteur:
x <- 1:20Un facteur (de la même longueur!) définition des groupes:
y <- factor(rep(letters[1:5], each = 4))additionner les valeurs de
xdans chaque sous-groupe défini pary:tapply(x, y, sum) a b c d e 10 26 42 58 74des exemples plus complexes peuvent être traités lorsque les sous-groupes sont définis par des combinaisons uniques d'une liste de plusieurs facteurs.
tapplyest semblable dans l'esprit à la split-apply-combinent des fonctions qui être fréquent (aggregate,by,ave,ddply, etc.) D'où son statut de mouton noir.
sur la note latérale, voici comment les diverses fonctions plyr correspondent aux fonctions de base *apply (de l'intro au document plyr de la page Web http://had.co.nz/plyr / )
Base function Input Output plyr function
---------------------------------------
aggregate d d ddply + colwise
apply a a/l aaply / alply
by d l dlply
lapply l l llply
mapply a a/l maply / mlply
replicate r a/l raply / rlply
sapply l a laply
l'un des objectifs de plyr est de fournir des conventions d'appellation cohérentes pour chacune des fonctions, en encodant les types de données d'entrée et de sortie dans le nom de la fonction. Il fournit également la stabilité de la puissance, qui la sortie de dlply() peut facilement passer à ldply() pour produire une sortie utile, etc.
sur le plan conceptuel, l'apprentissage plyr n'est pas plus difficile que de comprendre les fonctions de base *apply .
plyr et reshape fonctions ont remplacé presque toutes ces fonctions dans mon utilisation quotidienne. Mais, aussi de L'Intro à Plyr document:
fonctions connexes
tapplyetsweepn'ont pas de fonction correspondante dansplyr, et restent utiles.mergeest utile pour combiner les résumés avec les données originales.
de la diapositive 21 de http://www.slideshare.net/hadley/plyr-one-data-analytic-strategy :
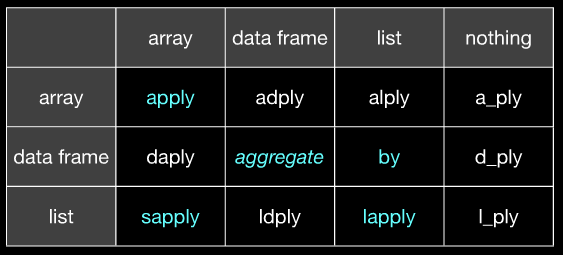
(J'espère qu'il est clair que apply correspond à aaply et aggregate de @Hadley correspond à ddply etc. La diapositive 20 du même slideshare clarifiera si vous ne l'obtenez pas de cette image.)
(sur la gauche de l'entrée, sur le dessus est production)
d'Abord commencer par Joran est excellente réponse -- douteux tout peut mieux que.
alors la mnémonique suivante peut aider à se rappeler les distinctions entre chacun. Tandis que certains sont évidents, d'autres peuvent être moins --- pour ceux-ci, vous trouverez la justification dans les discussions de Joran.
Mnémoniques
-
lapplyest un liste , qui agit sur une liste ou un vecteur et renvoie une liste. -
sapplyest un simplelapply(fonction par défaut de retour d'un vecteur ou d'une matrice lorsque cela est possible) -
vapplyis a verified apply (permet de préciser le type d'objet retourné) -
rapplyest un récursive s'appliquent pour les listes imbriquées, c'est à dire les listes dans les listes -
tapplyest un marqué appliquer où les étiquettes d'identifier les sous-ensembles -
applyest générique : applique une fonction à une matrice de lignes ou de colonnes (ou, plus généralement, aux dimensions d'un tableau)
construire le fond droit
si l'utilisation de la famille apply se sent encore un peu étranger à vous, alors il se peut que vous manquiez un point de vue clé.
ces deux articles peuvent vous aider. Ils fournissent le contexte nécessaire pour motiver les "techniques de programmation fonctionnelle qui sont fournis par la apply famille de fonctions.
les utilisateurs de Lisp reconnaîtront immédiatement le paradigme. Si vous n'êtes pas familier avec le Lisp, une fois que vous obtenez votre tête autour de la FP, vous aurez gagné un puissant point de vue de L'utilisation dans R -- et apply fera beaucoup plus de sens.
- Advanced R: Functional Programming , par Hadley Wickham
- de Simples de la Programmation Fonctionnelle dans R , par Michael Barton
depuis que je me suis rendu compte que (les réponses très excellentes) de ce manque de by et aggregate explications. Voici ma contribution.
par
la fonction by , comme indiqué dans la documentation peut être cependant, comme un" wrapper "pour tapply . La puissance de by apparaît lorsque nous voulons calculer une tâche que tapply ne peut pas gérer. Un exemple est ce code:
ct <- tapply(iris$Sepal.Width , iris$Species , summary )
cb <- by(iris$Sepal.Width , iris$Species , summary )
cb
iris$Species: setosa
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.300 3.200 3.400 3.428 3.675 4.400
--------------------------------------------------------------
iris$Species: versicolor
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.000 2.525 2.800 2.770 3.000 3.400
--------------------------------------------------------------
iris$Species: virginica
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.200 2.800 3.000 2.974 3.175 3.800
ct
$setosa
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.300 3.200 3.400 3.428 3.675 4.400
$versicolor
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.000 2.525 2.800 2.770 3.000 3.400
$virginica
Min. 1st Qu. Median Mean 3rd Qu. Max.
2.200 2.800 3.000 2.974 3.175 3.800
si nous imprimons ces deux objets, ct et cb , nous avons" essentiellement "les mêmes résultats et les seules différences sont dans la façon dont ils sont affichés et les différents attributs class , respectivement by pour cb et array pour ct .
comme je l'ai dit, la puissance de by se produit quand nous ne pouvons pas utiliser tapply ; le code suivant est un exemple:
tapply(iris, iris$Species, summary )
Error in tapply(iris, iris$Species, summary) :
arguments must have same length
r dit que les arguments doivent avoir les mêmes longueurs, dire "nous voulons calculer le summary de toutes les variables dans iris le long du facteur Species ": mais R ne peut pas le faire parce qu'il ne sait pas comment gérer.
avec la by Fonction R envoyer une méthode spécifique pour data frame classe et puis laisser la summary fonction fonctionne même si la longueur du premier argument (et le type aussi) sont différents.
bywork <- by(iris, iris$Species, summary )
bywork
iris$Species: setosa
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.300 Min. :2.300 Min. :1.000 Min. :0.100 setosa :50
1st Qu.:4.800 1st Qu.:3.200 1st Qu.:1.400 1st Qu.:0.200 versicolor: 0
Median :5.000 Median :3.400 Median :1.500 Median :0.200 virginica : 0
Mean :5.006 Mean :3.428 Mean :1.462 Mean :0.246
3rd Qu.:5.200 3rd Qu.:3.675 3rd Qu.:1.575 3rd Qu.:0.300
Max. :5.800 Max. :4.400 Max. :1.900 Max. :0.600
--------------------------------------------------------------
iris$Species: versicolor
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.900 Min. :2.000 Min. :3.00 Min. :1.000 setosa : 0
1st Qu.:5.600 1st Qu.:2.525 1st Qu.:4.00 1st Qu.:1.200 versicolor:50
Median :5.900 Median :2.800 Median :4.35 Median :1.300 virginica : 0
Mean :5.936 Mean :2.770 Mean :4.26 Mean :1.326
3rd Qu.:6.300 3rd Qu.:3.000 3rd Qu.:4.60 3rd Qu.:1.500
Max. :7.000 Max. :3.400 Max. :5.10 Max. :1.800
--------------------------------------------------------------
iris$Species: virginica
Sepal.Length Sepal.Width Petal.Length Petal.Width Species
Min. :4.900 Min. :2.200 Min. :4.500 Min. :1.400 setosa : 0
1st Qu.:6.225 1st Qu.:2.800 1st Qu.:5.100 1st Qu.:1.800 versicolor: 0
Median :6.500 Median :3.000 Median :5.550 Median :2.000 virginica :50
Mean :6.588 Mean :2.974 Mean :5.552 Mean :2.026
3rd Qu.:6.900 3rd Qu.:3.175 3rd Qu.:5.875 3rd Qu.:2.300
Max. :7.900 Max. :3.800 Max. :6.900 Max. :2.500
cela fonctionne en effet et le résultat est très surprenant. C'est un objet de la classe by que Species (disons, pour chacun d'eux) calcule la summary de chaque variable.
notez que si le premier argument est un data frame , la fonction dispatchée doit avoir une méthode pour cette classe d'objets. Par exemple, si nous utilisons ce code avec la fonction mean , nous aurons ce code qui n'a aucun sens:
by(iris, iris$Species, mean)
iris$Species: setosa
[1] NA
-------------------------------------------
iris$Species: versicolor
[1] NA
-------------------------------------------
iris$Species: virginica
[1] NA
Warning messages:
1: In mean.default(data[x, , drop = FALSE], ...) :
argument is not numeric or logical: returning NA
2: In mean.default(data[x, , drop = FALSE], ...) :
argument is not numeric or logical: returning NA
3: In mean.default(data[x, , drop = FALSE], ...) :
argument is not numeric or logical: returning NA
agrégat
aggregate peut être considéré comme une autre façon d'utiliser tapply si nous l'utilisons de cette manière.
at <- tapply(iris$Sepal.Length , iris$Species , mean)
ag <- aggregate(iris$Sepal.Length , list(iris$Species), mean)
at
setosa versicolor virginica
5.006 5.936 6.588
ag
Group.1 x
1 setosa 5.006
2 versicolor 5.936
3 virginica 6.588
Les deux différences sont que le deuxième argument de aggregate doit être une liste de tapply peut (pas obligatoire) être une liste et que la sortie de aggregate est un bloc de données, tandis que celui de tapply est un array .
la puissance de aggregate est qu'il peut manipuler facilement des sous-ensembles des données avec subset argument et qu'il a des méthodes pour ts objets et formula ainsi.
ces éléments rendent aggregate plus facile à utiliser que tapply dans certaines situations.
Voici quelques exemples (disponibles dans la documentation):
ag <- aggregate(len ~ ., data = ToothGrowth, mean)
ag
supp dose len
1 OJ 0.5 13.23
2 VC 0.5 7.98
3 OJ 1.0 22.70
4 VC 1.0 16.77
5 OJ 2.0 26.06
6 VC 2.0 26.14
nous pouvons obtenir la même chose avec tapply mais la syntaxe est légèrement plus dure et la sortie (dans certaines circonstances) moins lisible:
att <- tapply(ToothGrowth$len, list(ToothGrowth$dose, ToothGrowth$supp), mean)
att
OJ VC
0.5 13.23 7.98
1 22.70 16.77
2 26.06 26.14
il y a d'autres moments où nous ne pouvons pas utiliser by ou tapply et nous devons utiliser aggregate .
ag1 <- aggregate(cbind(Ozone, Temp) ~ Month, data = airquality, mean)
ag1
Month Ozone Temp
1 5 23.61538 66.73077
2 6 29.44444 78.22222
3 7 59.11538 83.88462
4 8 59.96154 83.96154
5 9 31.44828 76.89655
nous ne pouvons pas obtenir le résultat précédent avec tapply en un seul appel mais nous devons calculer la moyenne le long de Month pour chaque élément et puis les combiner (notez aussi que nous devons appeler le na.rm = TRUE , parce que les méthodes formula de la fonction aggregate ont par défaut le na.action = na.omit ):
ta1 <- tapply(airquality$Ozone, airquality$Month, mean, na.rm = TRUE)
ta2 <- tapply(airquality$Temp, airquality$Month, mean, na.rm = TRUE)
cbind(ta1, ta2)
ta1 ta2
5 23.61538 65.54839
6 29.44444 79.10000
7 59.11538 83.90323
8 59.96154 83.96774
9 31.44828 76.90000
alors qu'avec by nous ne pouvons tout simplement pas atteindre que, en fait, l'appel de fonction suivant renvoie une erreur (mais très probablement il est lié à la fonction fournie, mean ):
by(airquality[c("Ozone", "Temp")], airquality$Month, mean, na.rm = TRUE)
D'autres fois les résultats sont les mêmes et les différences sont juste dans la classe (et alors comment il est montré / imprimé et pas seulement -- exemple, comment le sous-traiter) objet:
byagg <- by(airquality[c("Ozone", "Temp")], airquality$Month, summary)
aggagg <- aggregate(cbind(Ozone, Temp) ~ Month, data = airquality, summary)
le code précédent atteint le même but et les mêmes résultats, à certains moments ce que l'outil à utiliser est juste une question de goûts et de besoins personnels; les deux objets précédents ont des besoins très différents en termes de subsetting.
Il ya beaucoup de grandes réponses qui traitent des différences dans les cas d'utilisation pour chaque fonction. Aucune des réponses ne traite des différences de rendement. C'est une cause raisonnable diverses fonctions attend divers input et produit divers output, mais la plupart d'entre eux ont un objectif commun général d'évaluer par série/groupes. Ma réponse sera axée sur la performance. En raison de ci-dessus la création d'entrée à partir des vecteurs est inclus dans le timing, aussi la fonction apply n'est pas mesurer.
j'ai testé deux fonctions différentes sum et length à la fois. Le Volume testé est de 50 m à l'entrée et de 50 K à la sortie. J'ai également inclus deux paquets actuellement populaires qui n'étaient pas largement utilisés au moment où la question a été posée, data.table et dplyr . Les deux sont certainement la peine de regarder si vous visez une bonne performance.
library(dplyr)
library(data.table)
set.seed(123)
n = 5e7
k = 5e5
x = runif(n)
grp = sample(k, n, TRUE)
timing = list()
# sapply
timing[["sapply"]] = system.time({
lt = split(x, grp)
r.sapply = sapply(lt, function(x) list(sum(x), length(x)), simplify = FALSE)
})
# lapply
timing[["lapply"]] = system.time({
lt = split(x, grp)
r.lapply = lapply(lt, function(x) list(sum(x), length(x)))
})
# tapply
timing[["tapply"]] = system.time(
r.tapply <- tapply(x, list(grp), function(x) list(sum(x), length(x)))
)
# by
timing[["by"]] = system.time(
r.by <- by(x, list(grp), function(x) list(sum(x), length(x)), simplify = FALSE)
)
# aggregate
timing[["aggregate"]] = system.time(
r.aggregate <- aggregate(x, list(grp), function(x) list(sum(x), length(x)), simplify = FALSE)
)
# dplyr
timing[["dplyr"]] = system.time({
df = data_frame(x, grp)
r.dplyr = summarise(group_by(df, grp), sum(x), n())
})
# data.table
timing[["data.table"]] = system.time({
dt = setnames(setDT(list(x, grp)), c("x","grp"))
r.data.table = dt[, .(sum(x), .N), grp]
})
# all output size match to group count
sapply(list(sapply=r.sapply, lapply=r.lapply, tapply=r.tapply, by=r.by, aggregate=r.aggregate, dplyr=r.dplyr, data.table=r.data.table),
function(x) (if(is.data.frame(x)) nrow else length)(x)==k)
# sapply lapply tapply by aggregate dplyr data.table
# TRUE TRUE TRUE TRUE TRUE TRUE TRUE
# print timings
as.data.table(sapply(timing, `[[`, "elapsed"), keep.rownames = TRUE
)[,.(fun = V1, elapsed = V2)
][order(-elapsed)]
# fun elapsed
#1: aggregate 109.139
#2: by 25.738
#3: dplyr 18.978
#4: tapply 17.006
#5: lapply 11.524
#6: sapply 11.326
#7: data.table 2.686
il est peut-être intéressant de mentionner ave . ave est le cousin amical de tapply . Il renvoie des résultats sous une forme que vous pouvez brancher directement dans votre base de données.
dfr <- data.frame(a=1:20, f=rep(LETTERS[1:5], each=4))
means <- tapply(dfr$a, dfr$f, mean)
## A B C D E
## 2.5 6.5 10.5 14.5 18.5
## great, but putting it back in the data frame is another line:
dfr$m <- means[dfr$f]
dfr$m2 <- ave(dfr$a, dfr$f, FUN=mean) # NB argument name FUN is needed!
dfr
## a f m m2
## 1 A 2.5 2.5
## 2 A 2.5 2.5
## 3 A 2.5 2.5
## 4 A 2.5 2.5
## 5 B 6.5 6.5
## 6 B 6.5 6.5
## 7 B 6.5 6.5
## ...
il n'y a rien dans le paquet de base qui fonctionne comme ave pour les bases de données entières (comme by est comme tapply pour les bases de données). Mais vous pouvez le caresser:
dfr$foo <- ave(1:nrow(dfr), dfr$f, FUN=function(x) {
x <- dfr[x,]
sum(x$m*x$m2)
})
dfr
## a f m m2 foo
## 1 1 A 2.5 2.5 25
## 2 2 A 2.5 2.5 25
## 3 3 A 2.5 2.5 25
## ...
malgré toutes les grandes réponses ici, il y a 2 autres fonctions de base qui méritent d'être mentionnées, la fonction utile outer et l'obscure eapply fonction
extérieur
outer est une fonction très utile cachée comme une plus banale. Si vous lisez l'aide pour outer sa description dit:
The outer product of the arrays X and Y is the array A with dimension
c(dim(X), dim(Y)) where element A[c(arrayindex.x, arrayindex.y)] =
FUN(X[arrayindex.x], Y[arrayindex.y], ...).
qui donne l'impression que c'est utile seulement pour les choses de type algèbre linéaire. Cependant, il peut être utilisé comme mapply appliquer une fonction à deux vecteurs d'entrées. La différence est que mapply appliquera la fonction aux deux premiers éléments, puis aux deux autres, etc., tandis que outer appliquera la fonction à chaque combinaison d'un élément du premier vecteur et d'un élément du second. Par exemple:
A<-c(1,3,5,7,9)
B<-c(0,3,6,9,12)
mapply(FUN=pmax, A, B)
> mapply(FUN=pmax, A, B)
[1] 1 3 6 9 12
outer(A,B, pmax)
> outer(A,B, pmax)
[,1] [,2] [,3] [,4] [,5]
[1,] 1 3 6 9 12
[2,] 3 3 6 9 12
[3,] 5 5 6 9 12
[4,] 7 7 7 9 12
[5,] 9 9 9 9 12
j'ai personnellement utilisé cette quand j'ai un vecteur de valeurs et d'un vectorielle des conditions et souhaitent voir quelles valeurs répondent à quelles conditions.
eapply
eapply est comme lapply sauf que plutôt que d'appliquer une fonction à chaque élément d'une liste, il applique une fonction à chaque élément d'un environnement. Par exemple, si vous voulez trouver une liste des fonctions définies par l'utilisateur dans l'environnement mondial:
A<-c(1,3,5,7,9)
B<-c(0,3,6,9,12)
C<-list(x=1, y=2)
D<-function(x){x+1}
> eapply(.GlobalEnv, is.function)
$A
[1] FALSE
$B
[1] FALSE
$C
[1] FALSE
$D
[1] TRUE
franchement je n'utilise pas très beaucoup, mais si vous construisez beaucoup de paquets ou Créez beaucoup d'environnements, cela peut s'avérer pratique.
j'ai récemment découvert la fonction plutôt utile sweep et l'ajouter ici par souci d'exhaustivité:
sweep
l'idée de base est de balayer à travers une rangée de tableaux - ou la colonne-sage et retourner un tableau modifié. Un exemple le montrera clairement (source: datacamp ):
disons que vous avez une matrice et que vous voulez standardiser it colonne-wise:
dataPoints <- matrix(4:15, nrow = 4)
# Find means per column with `apply()`
dataPoints_means <- apply(dataPoints, 2, mean)
# Find standard deviation with `apply()`
dataPoints_sdev <- apply(dataPoints, 2, sd)
# Center the points
dataPoints_Trans1 <- sweep(dataPoints, 2, dataPoints_means,"-")
print(dataPoints_Trans1)
## [,1] [,2] [,3]
## [1,] -1.5 -1.5 -1.5
## [2,] -0.5 -0.5 -0.5
## [3,] 0.5 0.5 0.5
## [4,] 1.5 1.5 1.5
# Return the result
dataPoints_Trans1
## [,1] [,2] [,3]
## [1,] -1.5 -1.5 -1.5
## [2,] -0.5 -0.5 -0.5
## [3,] 0.5 0.5 0.5
## [4,] 1.5 1.5 1.5
# Normalize
dataPoints_Trans2 <- sweep(dataPoints_Trans1, 2, dataPoints_sdev, "/")
# Return the result
dataPoints_Trans2
## [,1] [,2] [,3]
## [1,] -1.1618950 -1.1618950 -1.1618950
## [2,] -0.3872983 -0.3872983 -0.3872983
## [3,] 0.3872983 0.3872983 0.3872983
## [4,] 1.1618950 1.1618950 1.1618950
NB: pour cet exemple simple, le même résultat peut bien sûr être obtenu plus facilement en
apply(dataPoints, 2, scale)