Ajustement d'une distribution de Weibull à L'aide de Scipy
j'essaie de recréer la distribution de vraisemblance maximale, je peux déjà le faire dans Matlab et R, mais maintenant je veux utiliser scipy. En particulier, j'aimerais estimer les paramètres de distribution de Weibull pour mon ensemble de données.
j'ai essayé ceci:
import scipy.stats as s
import numpy as np
import matplotlib.pyplot as plt
def weib(x,n,a):
return (a / n) * (x / n)**(a - 1) * np.exp(-(x / n)**a)
data = np.loadtxt("stack_data.csv")
(loc, scale) = s.exponweib.fit_loc_scale(data, 1, 1)
print loc, scale
x = np.linspace(data.min(), data.max(), 1000)
plt.plot(x, weib(x, loc, scale))
plt.hist(data, data.max(), normed=True)
plt.show()
Et d'obtenir ceci:
(2.5827280639441961, 3.4955032285727947)
et une distribution qui ressemble à ceci:
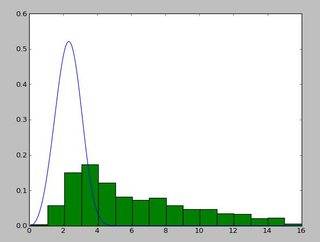
j'ai utilisé le exponweib après avoir lu ce http://www.johndcook.com/distributions_scipy.html . J'ai également essayé les autres fonctions Weibull en scipy (juste au cas où!).
dans Matlab (en utilisant L'outil D'ajustement de Distribution - voir la capture d'écran) et dans R (en utilisant à la fois la fonction de bibliothèque de masse fitdistr et le paquet GAMLSS) j'obtiens des Paramètres A (loc) et b (scale) plus comme 1.58463497 5.93030013. Je crois que les trois méthodes utilisez la méthode du maximum de vraisemblance pour ajuster la distribution.
j'ai posté mes données ici si vous souhaitez avoir un aller! Et pour être complet, J'utilise Python 2.7.5, Scipy 0.12.0, R 2.15.2 et Matlab 2012b.
pourquoi j'obtiens un résultat différent!?
8 réponses
mon avis est que vous voulez estimer le paramètre de forme et l'échelle de la distribution de Weibull tout en maintenant l'emplacement fixe. Fixation loc suppose que les valeurs de vos données et de la distribution sont positifs avec une limite inférieure à zéro.
floc=0 maintient l'emplacement fixé à zéro, f0=1 garde le premier paramètre de forme de l'exponentielle, weibull fixé à un.
>>> stats.exponweib.fit(data, floc=0, f0=1)
[1, 1.8553346917584836, 0, 6.8820748596850905]
>>> stats.weibull_min.fit(data, floc=0)
[1.8553346917584836, 0, 6.8820748596850549]
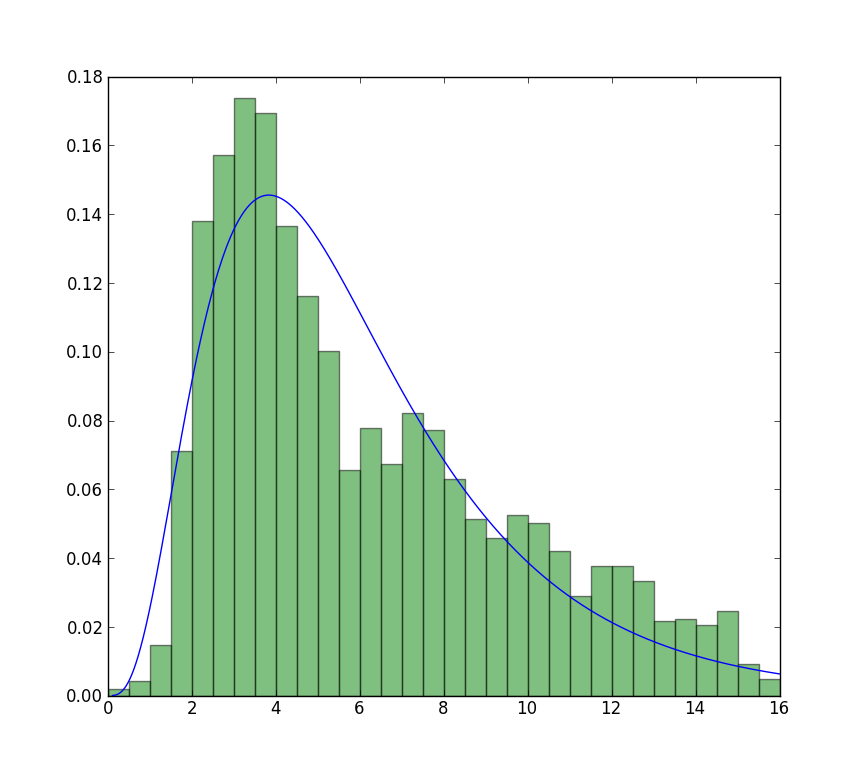
L'ajustement par rapport à la l'histogramme semble correct, mais pas très bon. Les estimations des paramètres sont un peu plus élevées que celles de R et matlab.
mise à Jour
le plus proche que je peux obtenir de la parcelle qui est maintenant disponible est avec ajustement illimité, mais en utilisant des valeurs de départ. L'intrigue est encore moins retentissante. Note les valeurs de fit qui n'ont pas de f devant sont utilisées comme valeurs de départ.
>>> from scipy import stats
>>> import matplotlib.pyplot as plt
>>> plt.plot(data, stats.exponweib.pdf(data, *stats.exponweib.fit(data, 1, 1, scale=02, loc=0)))
>>> _ = plt.hist(data, bins=np.linspace(0, 16, 33), normed=True, alpha=0.5);
>>> plt.show()
il est facile de vérifier quel résultat est le vrai MLE, juste besoin d'une fonction simple pour calculer log vraisemblance:
>>> def wb2LL(p, x): #log-likelihood
return sum(log(stats.weibull_min.pdf(x, p[1], 0., p[0])))
>>> adata=loadtxt('/home/user/stack_data.csv')
>>> wb2LL(array([6.8820748596850905, 1.8553346917584836]), adata)
-8290.1227946678173
>>> wb2LL(array([5.93030013, 1.57463497]), adata)
-8410.3327470347667
le résultat de fit méthode de exponweib et R fitdistr (@Warren) est meilleur et a une plus grande probabilité logarithmique. Il est plus probable que ce soit le vrai MLE. Il n'est pas surprenant que le résultat de GAMLSS soit différent. Il s'agit d'un modèle statistique complètement différent: modèle additif généralisé.
toujours pas convaincu? Nous pouvons dessiner un tracé de limite de confiance 2D autour de MLE, voir le livre de Meeker et Escobar pour plus de détails). 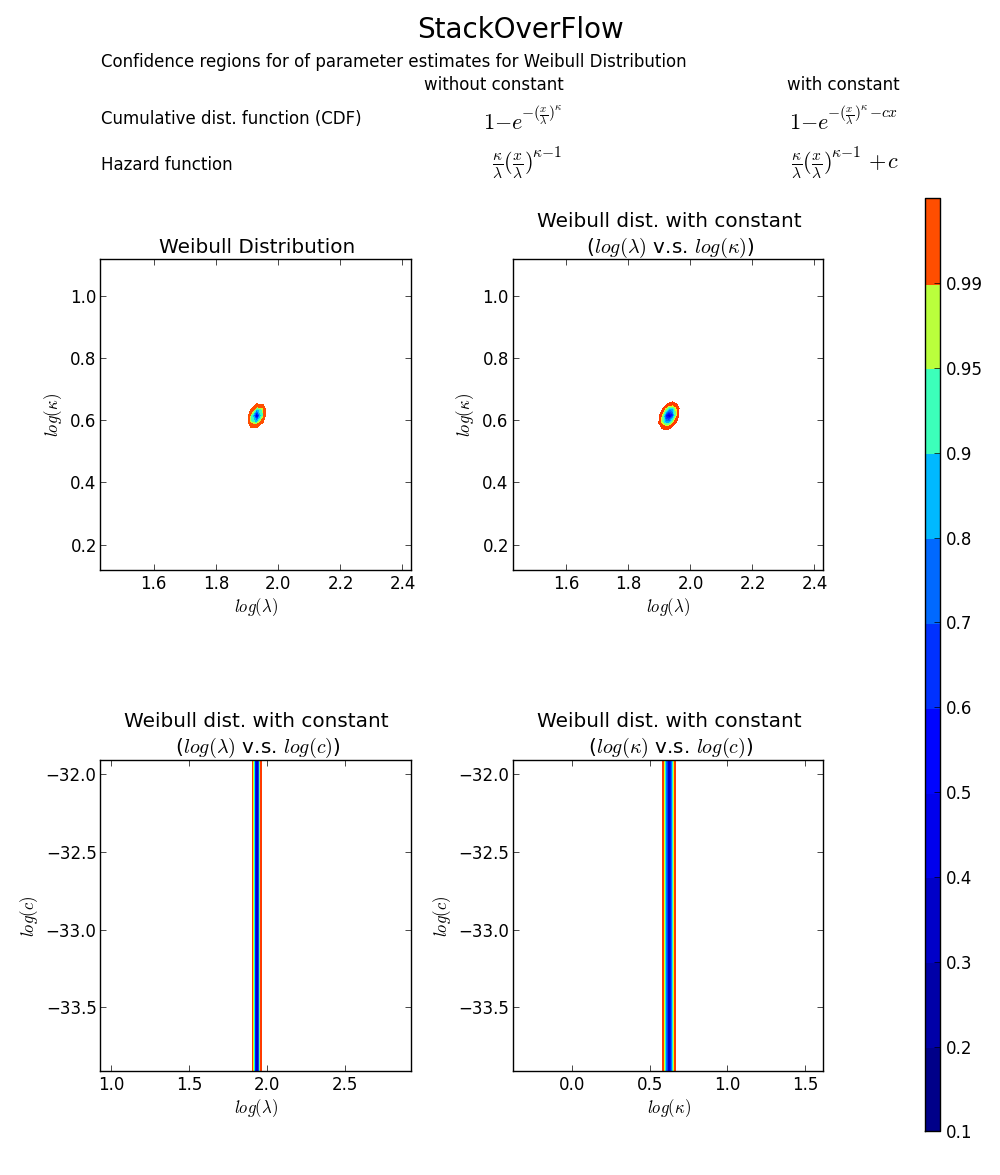
vérifie à nouveau que array([6.8820748596850905, 1.8553346917584836]) est la bonne réponse car la loglikelihood est inférieure à tout autre point dans l'espace paramètre. Note:
>>> log(array([6.8820748596850905, 1.8553346917584836]))
array([ 1.92892018, 0.61806511])
BTW1, MLE ajustement peut ne pas apparaît pour l'ajustement de l'histogramme de la distribution étroitement. Une façon facile de penser à MLE est que MLE est le paramètre estimé le plus probable compte tenu des données observées. Il n'a pas besoin de bien ajuster visuellement l'histogramme, ce sera quelque chose qui minimisera l'erreur carrée moyenne.
BTW2, vos données semblent être leptokurtiques et asymétriques à gauche, ce qui signifie que la distribution de Weibull pourrait ne pas correspondre bien à vos données. Essayez, par exemple Gompertz-logistique, qui améliore log-probabilité d'un autre d'environ 100.
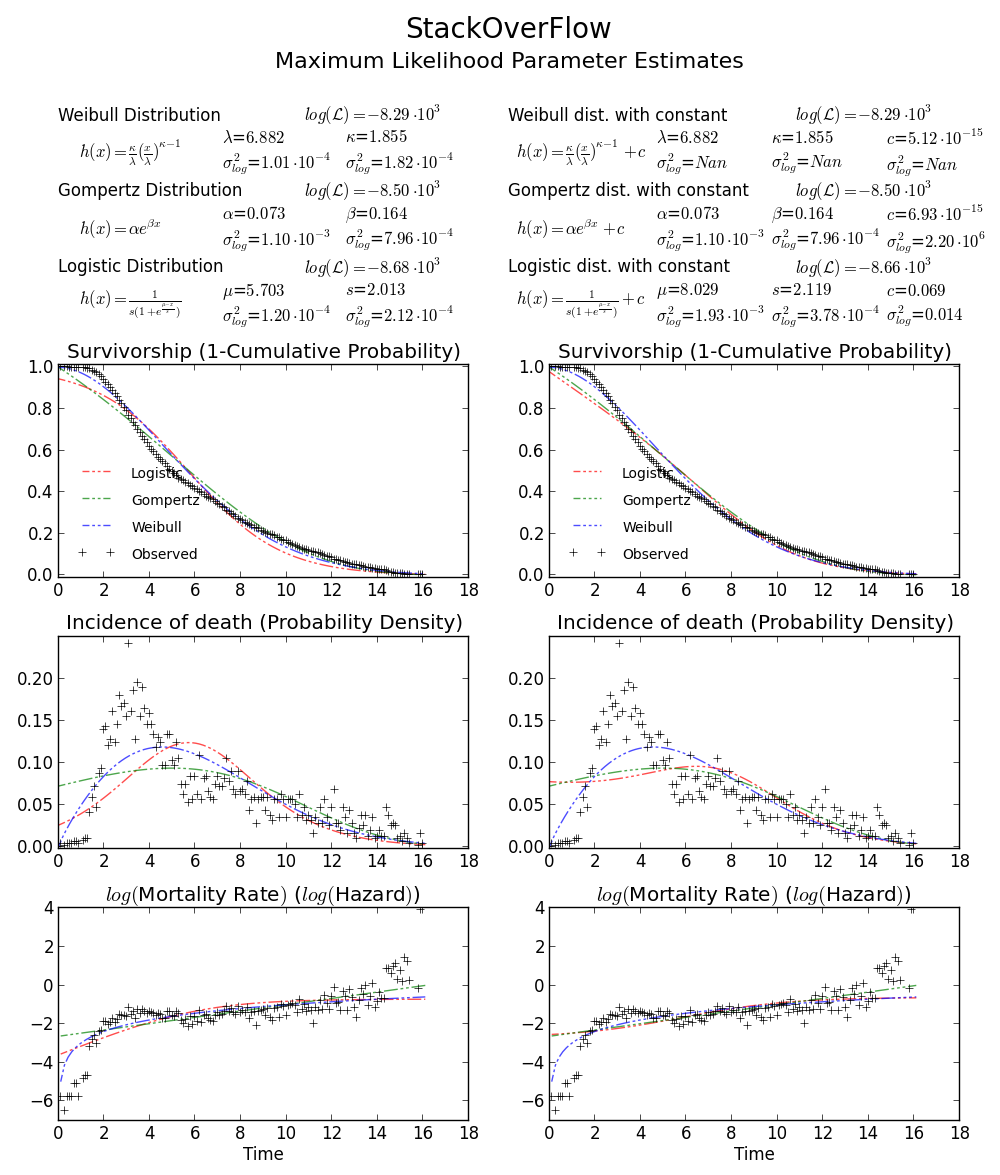
 Acclamations!
Acclamations!
j'étais curieux au sujet de votre question et, malgré cela n'est pas une réponse, il compare le Matlab résultat avec votre résultat et avec le résultat en utilisant leastsq , qui a montré la meilleure corrélation avec les données Données données:
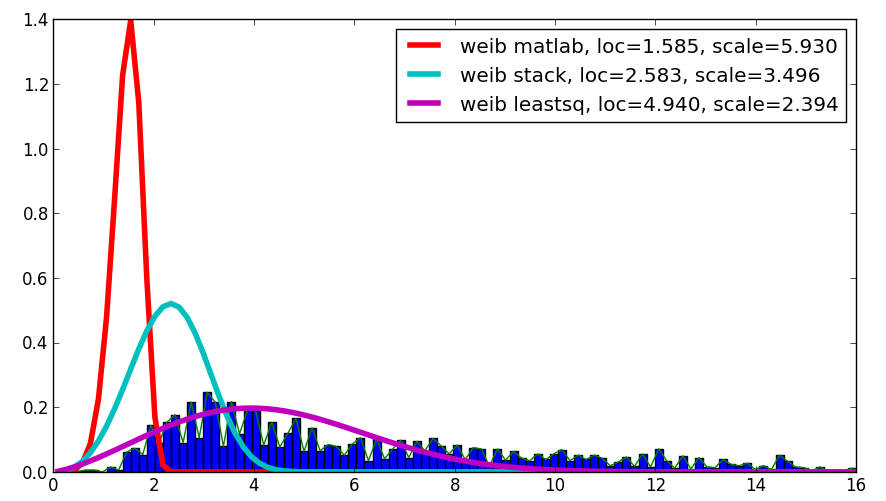
le code est le suivant:
import scipy.stats as s
import numpy as np
import matplotlib.pyplot as plt
import numpy.random as mtrand
from scipy.integrate import quad
from scipy.optimize import leastsq
## my distribution (Inverse Normal with shape parameter mu=1.0)
def weib(x,n,a):
return (a / n) * (x / n)**(a-1) * np.exp(-(x/n)**a)
def residuals(p,x,y):
integral = quad( weib, 0, 16, args=(p[0],p[1]) )[0]
penalization = abs(1.-integral)*100000
return y - weib(x, p[0],p[1]) + penalization
#
data = np.loadtxt("stack_data.csv")
x = np.linspace(data.min(), data.max(), 100)
n, bins, patches = plt.hist(data,bins=x, normed=True)
binsm = (bins[1:]+bins[:-1])/2
popt, pcov = leastsq(func=residuals, x0=(1.,1.), args=(binsm,n))
loc, scale = 1.58463497, 5.93030013
plt.plot(binsm,n)
plt.plot(x, weib(x, loc, scale),
label='weib matlab, loc=%1.3f, scale=%1.3f' % (loc, scale), lw=4.)
loc, scale = s.exponweib.fit_loc_scale(data, 1, 1)
plt.plot(x, weib(x, loc, scale),
label='weib stack, loc=%1.3f, scale=%1.3f' % (loc, scale), lw=4.)
plt.plot(x, weib(x,*popt),
label='weib leastsq, loc=%1.3f, scale=%1.3f' % tuple(popt), lw=4.)
plt.legend(loc='upper right')
plt.show()
je sais que c'est un vieux post, mais je viens de faire face à un problème similaire et ce fil m'a aidé à le résoudre. J'ai pensé que ma solution pourrait être utile pour d'autres comme moi:
# Fit Weibull function, some explanation below
params = stats.exponweib.fit(data, floc=0, f0=1)
shape = params[1]
scale = params[3]
print 'shape:',shape
print 'scale:',scale
#### Plotting
# Histogram first
values,bins,hist = plt.hist(data,bins=51,range=(0,25),normed=True)
center = (bins[:-1] + bins[1:]) / 2.
# Using all params and the stats function
plt.plot(center,stats.exponweib.pdf(center,*params),lw=4,label='scipy')
# Using my own Weibull function as a check
def weibull(u,shape,scale):
'''Weibull distribution for wind speed u with shape parameter k and scale parameter A'''
return (shape / scale) * (u / scale)**(shape-1) * np.exp(-(u/scale)**shape)
plt.plot(center,weibull(center,shape,scale),label='Wind analysis',lw=2)
plt.legend()
Quelques informations supplémentaires qui m'ont aidé à comprendre:
Scipy fonction Weibull peut prendre quatre paramètres d'entrée: (a,c),loc et scale. Vous voulez corriger la loc et le premier paramètre de forme (a), cela est fait avec floc=0,f0=1. L'ajustement vous donnera alors des paramètres c et scale, où c correspond au paramètre de forme de la distribution de Weibull à deux paramètres (souvent utilisée dans l'analyse des données sur le vent) et l'échelle correspond à son facteur d'échelle.
De docs:
exponweib.pdf(x, a, c) =
a * c * (1-exp(-x**c))**(a-1) * exp(-x**c)*x**(c-1)
Si a est 1, alors
exponweib.pdf(x, a, c) =
c * (1-exp(-x**c))**(0) * exp(-x**c)*x**(c-1)
= c * (1) * exp(-x**c)*x**(c-1)
= c * x **(c-1) * exp(-x**c)
A partir de là, la relation avec la fonction Weibull "analyse du vent" devrait être plus claire
j'ai eu le MÊME PROBLÈME, MAIS j'ai trouvé que le réglage loc=0 dans exponweib.fit amorçait la pompe pour l'optimisation. C'est tout ce dont on avait besoin de la réponse de @user333700 . Je ne pouvais pas charger vos données -- votre lien de données pointe vers une image, pas des données. J'ai donc fait un test sur mes données à la place:
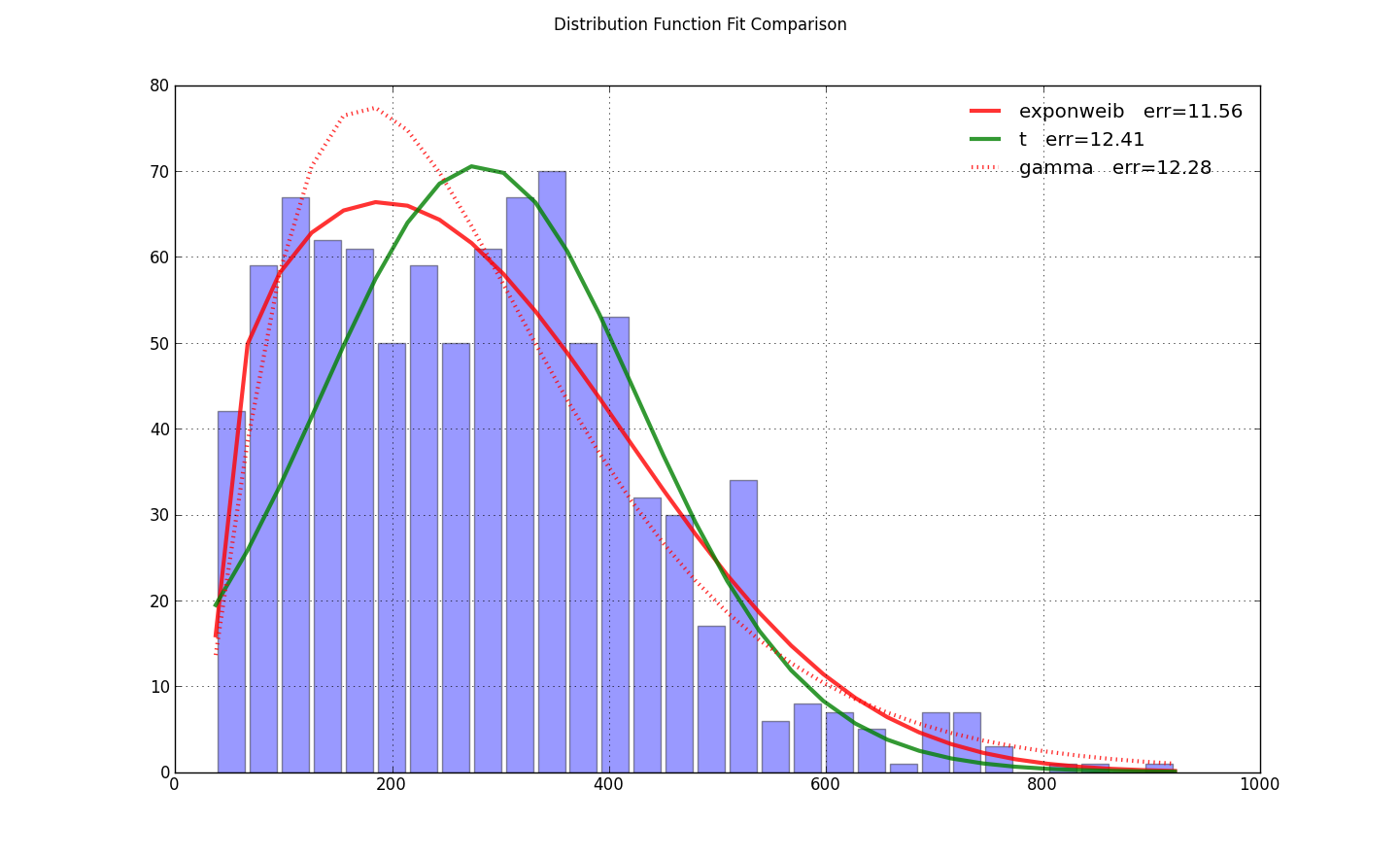
import scipy.stats as ss
import matplotlib.pyplot as plt
import numpy as np
N=30
counts, bins = np.histogram(x, bins=N)
bin_width = bins[1]-bins[0]
total_count = float(sum(counts))
f, ax = plt.subplots(1, 1)
f.suptitle(query_uri)
ax.bar(bins[:-1]+bin_width/2., counts, align='center', width=.85*bin_width)
ax.grid('on')
def fit_pdf(x, name='lognorm', color='r'):
dist = getattr(ss, name) # params = shape, loc, scale
# dist = ss.gamma # 3 params
params = dist.fit(x, loc=0) # 1-day lag minimum for shipping
y = dist.pdf(bins, *params)*total_count*bin_width
sqerror_sum = np.log(sum(ci*(yi - ci)**2. for (ci, yi) in zip(counts, y)))
ax.plot(bins, y, color, lw=3, alpha=0.6, label='%s err=%3.2f' % (name, sqerror_sum))
return y
colors = ['r-', 'g-', 'r:', 'g:']
for name, color in zip(['exponweib', 't', 'gamma'], colors): # 'lognorm', 'erlang', 'chi2', 'weibull_min',
y = fit_pdf(x, name=name, color=color)
ax.legend(loc='best', frameon=False)
plt.show()
il y a eu quelques réponses à cela déjà ici et dans d'autres endroits. likt dans distribution de Weibull et les données dans la même figure (avec numpy et scipy)
il m'a fallu encore un certain temps pour trouver un exemple de jouet propre donc j'ai pensé qu'il serait utile de poster.
from scipy import stats
import matplotlib.pyplot as plt
#input for pseudo data
N = 10000
Kappa_in = 1.8
Lambda_in = 10
a_in = 1
loc_in = 0
#Generate data from given input
data = stats.exponweib.rvs(a=a_in,c=Kappa_in, loc=loc_in, scale=Lambda_in, size = N)
#The a and loc are fixed in the fit since it is standard to assume they are known
a_out, Kappa_out, loc_out, Lambda_out = stats.exponweib.fit(data, f0=a_in,floc=loc_in)
#Plot
bins = range(51)
fig = plt.figure()
ax = fig.add_subplot(1, 1, 1)
ax.plot(bins, stats.exponweib.pdf(bins, a=a_out,c=Kappa_out,loc=loc_out,scale = Lambda_out))
ax.hist(data, bins = bins , normed=True, alpha=0.5)
ax.annotate("Shape: $k = %.2f$ \n Scale: $\lambda = %.2f$"%(Kappa_out,Lambda_out), xy=(0.7, 0.85), xycoords=ax.transAxes)
plt.show()
l'ordre de la ldc et de l'échelle est foiré dans le code:
plt.plot(x, weib(x, scale, loc))
le paramètre d'échelle doit passer en premier.
dans la fonction fit, il y a 3 paramètres à considérer:
-
les paramètres de forme: dans ce cas, nous avons deux paramètres de forme, qui peuvent être fixés selon f0 et f1. (Essayez par vous-même!). Habituellement, le nom du paramètre est indiqué par f%d, où d est le numéro de la forme.
-
le paramètre de localisation: utilisez floc pour corriger cela. Si vous ne pas fixer floc, la moyenne des données est délivrée comme loc.
-
le paramètre d'échelle: utilisez fscale pour corriger cela.
le retour de tout fit sort dans cet ordre.
suivant les lignes de @Peter9192, j'ai trouvé le meilleur ajustement pour un CDF Weibull de ~20-30 échantillons de données en utilisant le suivant:
_,gamma,_alpha=scipy.stats.exponweib.fit(data,floc=0,f0=1)
la formule pour CDF est:
1-np.exp(-np.power(x/alpha,gamma))
Les valeurs pour données j'ai estimé à l'aide d'un K-M estimateur de la méthode, correspondant à une distribution de Weibull m'a donné de bonnes valeurs.
pour fixer a comme 1, Je n'a pas loc=0, scale=1 comme la meilleure méthode comme vous pouvez clairement voir dans les 4 valeurs de paramètre retournées. Deuxièmement, en utilisant gamma, alpha de celui-ci n'a pas donné la moyenne de Weibull correcte.
enfin, j'ai confirmé quelle méthode fonctionne le mieux en calculant la moyenne de la Distribution de Weibull:
Mean=alpha*scipy.special.gamma(1+(1/gamma))
Les valeurs que j'ai obtenu correspondait à ma demande.
vous pouvez consulter les formules mean & CDF ici pour référence: https://en.m.wikipedia.org/wiki/Weibull_distribution