Échantillonnage pondéré plus rapide sans remplacement
L'échantillonnage par défaut decette question a conduit à un nouveau paquet R:
wrswoR
R sans remplacement par sample.int semble nécessiter un temps d'exécution quadratique, par exemple lorsqu'on utilise des poids tirés d'une distribution uniforme. Cela est lent pour les échantillons de grande taille. Est-ce que quelqu'un connaît une implémentation plus rapide qui serait utilisable à partir de R ? Deux options sont "Echantillonnage de rejet avec remplacement "(voir cette question sur les statistiques.sx) et l'algorithme de Wong et Easton (1980) (avec une implémentation Python dans un StackOverflow answer ).
merci à Ben Bolker pour l'allusion à la fonction C qui est appelée en interne lorsque sample.int est appelé avec replace=F et des poids non uniformes: ProbSampleNoReplace . En effet, le code montre deux imbriqués for les boucles (ligne 420 ff de random.c ).
voici le code pour analyser empiriquement le temps d'exécution:
library(plyr)
sample.int.test <- function(n, p) {
sample.int(2 * n, n, replace=F, prob=p); NULL }
times <- ldply(
1:7,
function(i) {
n <- 1024 * (2 ** i)
p <- runif(2 * n)
data.frame(
n=n,
user=system.time(sample.int.test(n, p), gcFirst=T)['user.self'])
},
.progress='text'
)
times
library(ggplot2)
ggplot(times, aes(x=n, y=user/n)) + geom_point() + scale_x_log10() +
ylab('Time per unit (s)')
# Output:
n user
1 2048 0.008
2 4096 0.028
3 8192 0.100
4 16384 0.408
5 32768 1.645
6 65536 6.604
7 131072 26.558
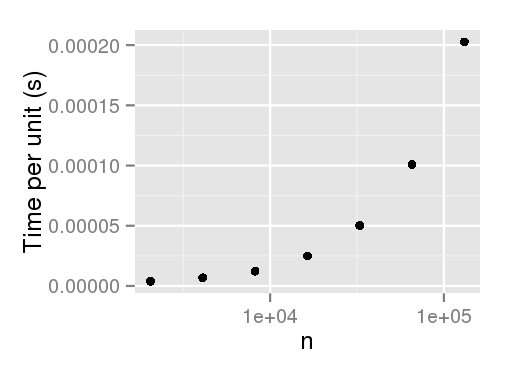
EDIT : merci à Arun de souligner que l'échantillonnage non pondéré ne semble pas avoir cette pénalité de performance.
3 réponses
mise à jour:
Un Rcpp la mise en œuvre de Efraimidis & Spirakis algorithme (merci à @Hemmo, @Dinrem, @krlmlr et @rtlgrmpf ):
library(inline)
library(Rcpp)
src <-
'
int num = as<int>(size), x = as<int>(n);
Rcpp::NumericVector vx = Rcpp::clone<Rcpp::NumericVector>(x);
Rcpp::NumericVector pr = Rcpp::clone<Rcpp::NumericVector>(prob);
Rcpp::NumericVector rnd = rexp(x) / pr;
for(int i= 0; i<vx.size(); ++i) vx[i] = i;
std::partial_sort(vx.begin(), vx.begin() + num, vx.end(), Comp(rnd));
vx = vx[seq(0, num - 1)] + 1;
return vx;
'
incl <-
'
struct Comp{
Comp(const Rcpp::NumericVector& v ) : _v(v) {}
bool operator ()(int a, int b) { return _v[a] < _v[b]; }
const Rcpp::NumericVector& _v;
};
'
funFast <- cxxfunction(signature(n = "Numeric", size = "integer", prob = "numeric"),
src, plugin = "Rcpp", include = incl)
# See the bottom of the answer for comparison
p <- c(995/1000, rep(1/1000, 5))
n <- 100000
system.time(print(table(replicate(funFast(6, 3, p), n = n)) / n))
1 2 3 4 5 6
1.00000 0.39996 0.39969 0.39973 0.40180 0.39882
user system elapsed
3.93 0.00 3.96
# In case of:
# Rcpp::IntegerVector vx = Rcpp::clone<Rcpp::IntegerVector>(x);
# i.e. instead of NumericVector
1 2 3 4 5 6
1.00000 0.40150 0.39888 0.39925 0.40057 0.39980
user system elapsed
1.93 0.00 2.03
ancienne version:
essayons quelques approches possibles:
Simple rejet de l'échantillonnage avec remplacement. c'est une fonction beaucoup plus simple que sample.int.rej offert par @krlmlr, c.-à-d. la taille de l'échantillon est toujours égale à n . Comme nous le verrons, il est encore très rapide en supposant une distribution uniforme des poids, mais extrêmement lent dans une autre situation.
fastSampleReject <- function(all, n, w){
out <- numeric(0)
while(length(out) < n)
out <- unique(c(out, sample(all, n, replace = TRUE, prob = w)))
out[1:n]
}
L'algorithme par Wong et Easton (1980) . Voici une implémentation de cette version de Python. C'est stable et il se peut que je manque quelque chose., mais il est beaucoup plus lent par rapport à d'autres fonctions.
fastSample1980 <- function(all, n, w){
tws <- w
for(i in (length(tws) - 1):0)
tws[1 + i] <- sum(tws[1 + i], tws[1 + 2 * i + 1],
tws[1 + 2 * i + 2], na.rm = TRUE)
out <- numeric(n)
for(i in 1:n){
gas <- tws[1] * runif(1)
k <- 0
while(gas > w[1 + k]){
gas <- gas - w[1 + k]
k <- 2 * k + 1
if(gas > tws[1 + k]){
gas <- gas - tws[1 + k]
k <- k + 1
}
}
wgh <- w[1 + k]
out[i] <- all[1 + k]
w[1 + k] <- 0
while(1 + k >= 1){
tws[1 + k] <- tws[1 + k] - wgh
k <- floor((k - 1) / 2)
}
}
out
}
Rcpp mise en œuvre de l'algorithme par Wong et Easton. peut-être il peut être optimisé encore plus puisque c'est ma première fonction utilisable Rcpp , mais de toute façon il fonctionne bien.
library(inline)
library(Rcpp)
src <-
'
Rcpp::NumericVector weights = Rcpp::clone<Rcpp::NumericVector>(w);
Rcpp::NumericVector tws = Rcpp::clone<Rcpp::NumericVector>(w);
Rcpp::NumericVector x = Rcpp::NumericVector(all);
int k, num = as<int>(n);
Rcpp::NumericVector out(num);
double gas, wgh;
if((weights.size() - 1) % 2 == 0){
tws[((weights.size()-1)/2)] += tws[weights.size()-1] + tws[weights.size()-2];
}
else
{
tws[floor((weights.size() - 1)/2)] += tws[weights.size() - 1];
}
for (int i = (floor((weights.size() - 1)/2) - 1); i >= 0; i--){
tws[i] += (tws[2 * i + 1]) + (tws[2 * i + 2]);
}
for(int i = 0; i < num; i++){
gas = as<double>(runif(1)) * tws[0];
k = 0;
while(gas > weights[k]){
gas -= weights[k];
k = 2 * k + 1;
if(gas > tws[k]){
gas -= tws[k];
k += 1;
}
}
wgh = weights[k];
out[i] = x[k];
weights[k] = 0;
while(k > 0){
tws[k] -= wgh;
k = floor((k - 1) / 2);
}
tws[0] -= wgh;
}
return out;
'
fun <- cxxfunction(signature(all = "numeric", n = "integer", w = "numeric"),
src, plugin = "Rcpp")
maintenant quelques résultats:
times1 <- ldply(
1:6,
function(i) {
n <- 1024 * (2 ** i)
p <- runif(2 * n) # Uniform distribution
p <- p/sum(p)
data.frame(
n=n,
user=c(system.time(sample.int.test(n, p), gcFirst=T)['user.self'],
system.time(weighted_Random_Sample(1:(2*n), p, n), gcFirst=T)['user.self'],
system.time(fun(1:(2*n), n, p), gcFirst=T)['user.self'],
system.time(sample.int.rej(2*n, n, p), gcFirst=T)['user.self'],
system.time(fastSampleReject(1:(2*n), n, p), gcFirst=T)['user.self'],
system.time(fastSample1980(1:(2*n), n, p), gcFirst=T)['user.self']),
id=c("Base", "Reservoir", "Rcpp", "Rejection", "Rejection simple", "1980"))
},
.progress='text'
)
times2 <- ldply(
1:6,
function(i) {
n <- 1024 * (2 ** i)
p <- runif(2 * n - 1)
p <- p/sum(p)
p <- c(0.999, 0.001 * p) # Special case
data.frame(
n=n,
user=c(system.time(sample.int.test(n, p), gcFirst=T)['user.self'],
system.time(weighted_Random_Sample(1:(2*n), p, n), gcFirst=T)['user.self'],
system.time(fun(1:(2*n), n, p), gcFirst=T)['user.self'],
system.time(sample.int.rej(2*n, n, p), gcFirst=T)['user.self'],
system.time(fastSampleReject(1:(2*n), n, p), gcFirst=T)['user.self'],
system.time(fastSample1980(1:(2*n), n, p), gcFirst=T)['user.self']),
id=c("Base", "Reservoir", "Rcpp", "Rejection", "Rejection simple", "1980"))
},
.progress='text'
)
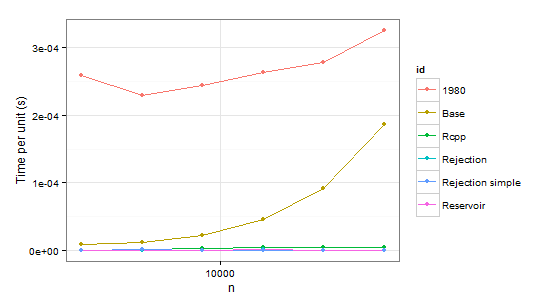
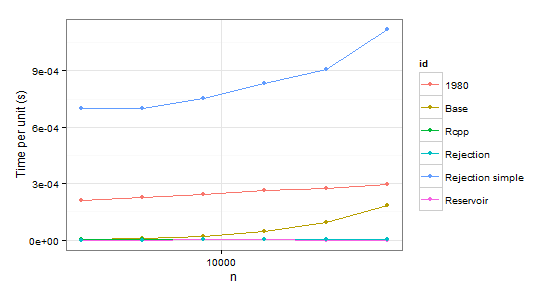
arrange(times1, id)
n user id
1 2048 0.53 1980
2 4096 0.94 1980
3 8192 2.00 1980
4 16384 4.32 1980
5 32768 9.10 1980
6 65536 21.32 1980
7 2048 0.02 Base
8 4096 0.05 Base
9 8192 0.18 Base
10 16384 0.75 Base
11 32768 2.99 Base
12 65536 12.23 Base
13 2048 0.00 Rcpp
14 4096 0.01 Rcpp
15 8192 0.03 Rcpp
16 16384 0.07 Rcpp
17 32768 0.14 Rcpp
18 65536 0.31 Rcpp
19 2048 0.00 Rejection
20 4096 0.00 Rejection
21 8192 0.00 Rejection
22 16384 0.02 Rejection
23 32768 0.02 Rejection
24 65536 0.03 Rejection
25 2048 0.00 Rejection simple
26 4096 0.01 Rejection simple
27 8192 0.00 Rejection simple
28 16384 0.01 Rejection simple
29 32768 0.00 Rejection simple
30 65536 0.05 Rejection simple
31 2048 0.00 Reservoir
32 4096 0.00 Reservoir
33 8192 0.00 Reservoir
34 16384 0.02 Reservoir
35 32768 0.03 Reservoir
36 65536 0.05 Reservoir
arrange(times2, id)
n user id
1 2048 0.43 1980
2 4096 0.93 1980
3 8192 2.00 1980
4 16384 4.36 1980
5 32768 9.08 1980
6 65536 19.34 1980
7 2048 0.01 Base
8 4096 0.04 Base
9 8192 0.18 Base
10 16384 0.75 Base
11 32768 3.11 Base
12 65536 12.04 Base
13 2048 0.01 Rcpp
14 4096 0.02 Rcpp
15 8192 0.03 Rcpp
16 16384 0.08 Rcpp
17 32768 0.15 Rcpp
18 65536 0.33 Rcpp
19 2048 0.00 Rejection
20 4096 0.00 Rejection
21 8192 0.02 Rejection
22 16384 0.02 Rejection
23 32768 0.05 Rejection
24 65536 0.08 Rejection
25 2048 1.43 Rejection simple
26 4096 2.87 Rejection simple
27 8192 6.17 Rejection simple
28 16384 13.68 Rejection simple
29 32768 29.74 Rejection simple
30 65536 73.32 Rejection simple
31 2048 0.00 Reservoir
32 4096 0.00 Reservoir
33 8192 0.02 Reservoir
34 16384 0.02 Reservoir
35 32768 0.02 Reservoir
36 65536 0.04 Reservoir
évidemment nous pouvons rejeter la fonction 1980 parce qu'elle est plus lente que Base dans les deux cas. Rejection simple est en difficulté aussi quand il ya une seule probabilité 0,999 dans le second cas.
donc il reste Rejection , Rcpp , Reservoir . La dernière étape est de vérifier si les valeurs sont correctes. Pour en être sûr, nous utiliserons sample comme point de repère (également pour éliminer la confusion sur les probabilités qui ne doivent pas coïncider avec p en raison de l'échantillonnage sans remplacement).
p <- c(995/1000, rep(1/1000, 5))
n <- 100000
system.time(print(table(replicate(sample(1:6, 3, repl = FALSE, prob = p), n = n))/n))
1 2 3 4 5 6
1.00000 0.39992 0.39886 0.40088 0.39711 0.40323 # Benchmark
user system elapsed
1.90 0.00 2.03
system.time(print(table(replicate(sample.int.rej(2*3, 3, p), n = n))/n))
1 2 3 4 5 6
1.00000 0.40007 0.40099 0.39962 0.40153 0.39779
user system elapsed
76.02 0.03 77.49 # Slow
system.time(print(table(replicate(weighted_Random_Sample(1:6, p, 3), n = n))/n))
1 2 3 4 5 6
1.00000 0.49535 0.41484 0.36432 0.36338 0.36211 # Incorrect
user system elapsed
3.64 0.01 3.67
system.time(print(table(replicate(fun(1:6, 3, p), n = n))/n))
1 2 3 4 5 6
1.00000 0.39876 0.40031 0.40219 0.40039 0.39835
user system elapsed
4.41 0.02 4.47
remarquez quelques petites choses ici. Pour quelque raison weighted_Random_Sample renvoie des valeurs incorrectes (Je ne l'ai pas regardé du tout, mais il fonctionne correctement en supposant une distribution uniforme). sample.int.rej est très lent dans les échantillonnages répétés.
en conclusion, il semble que Rcpp soit le choix optimal en cas de échantillonnage répété alors que sample.int.rej est un peu plus rapide autrement et aussi plus facile à utiliser.
j'ai décidé de creuser dans certains des commentaires et j'ai trouvé le papier Efraimidis & Spirakis fascinant (merci à @Hemmo d'avoir trouvé la référence). L'idée générale dans le document est la suivante: créer une clé en générant un nombre uniforme aléatoire et en l'élevant à la puissance d'un sur le poids de chaque élément. Ensuite, vous prenez simplement les valeurs clés les plus élevées comme échantillon. Cela fonctionne brillamment!
weighted_Random_Sample <- function(
.data,
.weights,
.n
){
key <- runif(length(.data)) ^ (1 / .weights)
return(.data[order(key, decreasing=TRUE)][1:.n])
}
si vous définissez '.n' étant la longueur de '.données "(qui devrait toujours être la longueur de".poids ('), il s'agit en fait d'une permutation pondérée du réservoir, mais la méthode fonctionne bien tant pour l'échantillonnage que pour la permutation.
Update : je devrais probablement mentionner que la fonction ci-dessus s'attend à ce que les poids soient supérieurs à zéro. Sinon key <- runif(length(.data)) ^ (1 / .weights) ne sera pas commandé correctement.
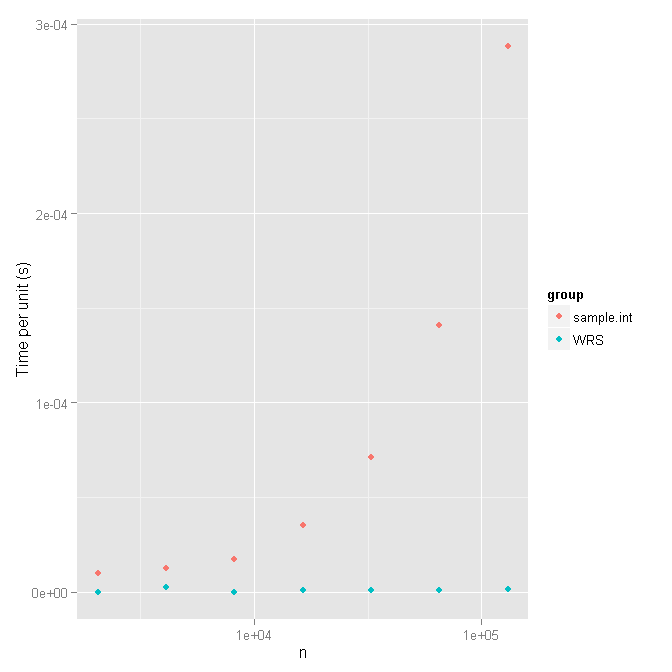
juste pour les coups de pied, j'ai également utilisé le test scénario dans le PO pour comparer les deux fonctions.
set.seed(1)
times_WRS <- ldply(
1:7,
function(i) {
n <- 1024 * (2 ** i)
p <- runif(2 * n)
n_Set <- 1:(2 * n)
data.frame(
n=n,
user=system.time(weighted_Random_Sample(n_Set, p, n), gcFirst=T)['user.self'])
},
.progress='text'
)
sample.int.test <- function(n, p) {
sample.int(2 * n, n, replace=F, prob=p); NULL }
times_sample.int <- ldply(
1:7,
function(i) {
n <- 1024 * (2 ** i)
p <- runif(2 * n)
data.frame(
n=n,
user=system.time(sample.int.test(n, p), gcFirst=T)['user.self'])
},
.progress='text'
)
times_WRS$group <- "WRS"
times_sample.int$group <- "sample.int"
library(ggplot2)
ggplot(rbind(times_WRS, times_sample.int) , aes(x=n, y=user/n, col=group)) + geom_point() + scale_x_log10() + ylab('Time per unit (s)')
et voici les temps:
times_WRS
# n user
# 1 2048 0.00
# 2 4096 0.01
# 3 8192 0.00
# 4 16384 0.01
# 5 32768 0.03
# 6 65536 0.06
# 7 131072 0.16
times_sample.int
# n user
# 1 2048 0.02
# 2 4096 0.05
# 3 8192 0.14
# 4 16384 0.58
# 5 32768 2.33
# 6 65536 9.23
# 7 131072 37.79
Permettez-moi de lancer ma propre mise en œuvre d'une approche plus rapide basée sur Echantillonnage de rejet avec remplacement . L'idée est la suivante:
-
Générer un échantillon avec de remplacement qui est "un peu" plus grande que la taille demandée
-
jetez les valeurs dupliquées
-
si les valeurs sont insuffisantes ont été tirées, appeler la même procédure récursivement avec ajustée
n,sizeetprobparamètres -
Remapper le retour de l'index à l'origine indices
de quelle taille doit-on prélever un échantillon? En supposant une distribution uniforme, le résultat est le nombre prévu d'essais pour voir x valeurs uniques sur N valeurs totales . C'est une différence d' deux nombres harmoniques (h_n et H_{n - size}). Les premiers nombres harmoniques sont tabulés, sinon une approximation utilisant le logarithme naturel est utilisée. (Ce n'est qu'un chiffre approximatif, pas besoin d'être trop précis ici. Maintenant, pour une distribution non uniforme, le nombre prévu d'articles à dessiner ne peut être que plus grand, donc nous ne ferons pas trop d'échantillons. En outre, le nombre d'échantillons est limité par deux fois la taille de la population -- je suppose que c'est plus rapide pour avoir quelques appels récursifs que l'échantillonnage jusqu'à O (N ln n) articles.
le code est disponible dans le paquet R wrswoR dans la routine sample.int.rej en sample_int_rej.R . Installation avec:
library(devtools)
install_github('wrswoR', 'muelleki')
Il semble que cela fonctionne "assez rapide", mais pas formel d'exécution des tests ont été réalisés encore. En outre, le paquet est testé en Ubuntu seulement. J'apprécie vos commentaires.