Estimer L'autocorrélation en utilisant Python
j'aimerais effectuer une autocorrélation sur le signal ci-dessous. Le temps entre deux points consécutifs est de 2,5 ms (ou un taux de répétition de 400Hz).
c'est l'équation pour estimer l'autoacrrélation que je voudrais utiliser (tiré de http://en.wikipedia.org/wiki/Autocorrelation , Estimation de section):
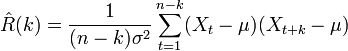
Quelle est la méthode la plus simple pour trouver l'autocorrélation estimée de mes données en python? Y a-t-il quelque chose de similaire à numpy.correlate que je peux utiliser?
ou dois-je simplement calculer la moyenne et la variance?
Edit:
avec l'aide de unutbu , j'ai écrit:
from numpy import *
import numpy as N
import pylab as P
fn = 'data.txt'
x = loadtxt(fn,unpack=True,usecols=[1])
time = loadtxt(fn,unpack=True,usecols=[0])
def estimated_autocorrelation(x):
n = len(x)
variance = x.var()
x = x-x.mean()
r = N.correlate(x, x, mode = 'full')[-n:]
#assert N.allclose(r, N.array([(x[:n-k]*x[-(n-k):]).sum() for k in range(n)]))
result = r/(variance*(N.arange(n, 0, -1)))
return result
P.plot(time,estimated_autocorrelation(x))
P.xlabel('time (s)')
P.ylabel('autocorrelation')
P.show()
5 réponses
Je ne pense pas qu'il y ait une fonction de NumPy pour ce calcul particulier. Voici comment je l'écrirais:
def estimated_autocorrelation(x):
"""
/q/estimate-autocorrelation-using-python-49624/"""
n = len(x)
variance = x.var()
x = x-x.mean()
r = np.correlate(x, x, mode = 'full')[-n:]
assert np.allclose(r, np.array([(x[:n-k]*x[-(n-k):]).sum() for k in range(n)]))
result = r/(variance*(np.arange(n, 0, -1)))
return result
l'affirmation est là pour vérifier le calcul et pour documenter son intention.
lorsque vous êtes sûr que cette fonction se comporte comme prévu, vous pouvez commenter la déclaration assert , ou exécuter votre script avec python -O . (Le drapeau -O dit à Python d'ignorer les affirmations d'assert.)
j'ai pris une partie du code de la fonction pandas autocorrelation_plot (). J'ai vérifié les réponses avec R et les valeurs correspondent exactement.
import numpy
def acf(series):
n = len(series)
data = numpy.asarray(series)
mean = numpy.mean(data)
c0 = numpy.sum((data - mean) ** 2) / float(n)
def r(h):
acf_lag = ((data[:n - h] - mean) * (data[h:] - mean)).sum() / float(n) / c0
return round(acf_lag, 3)
x = numpy.arange(n) # Avoiding lag 0 calculation
acf_coeffs = map(r, x)
return acf_coeffs
le paquet statsmodels ajoute une fonction d'autocorrélation qui utilise en interne np.correlate (selon la documentation statsmodels ).
la méthode que j'ai écrite à partir de ma dernière édition est maintenant plus rapide que même scipy.statstools.acf avec fft=True jusqu'à ce que la taille de l'échantillon devient très grande.
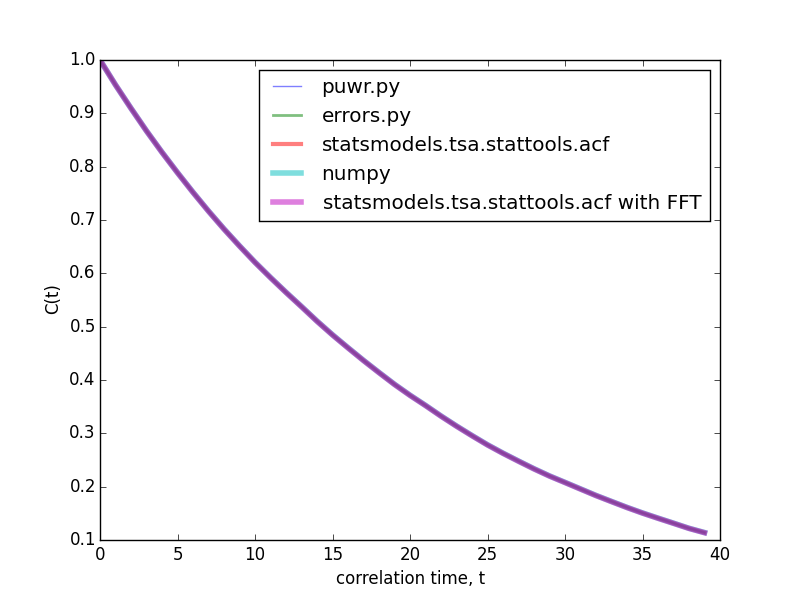
Erreur d'analyse Si vous voulez régler des préjugés et très précis d'erreur des estimations: Regardez mon code ici , qui met en œuvre les ce document par Ulli Wolff ( ou d'origine par l'université du wisconsin dans Matlab )
Fonctions Testées
-
a = correlatedData(n=10000)est d'une routine trouvée ici -
gamma()est du même endroit quecorrelated_data() -
acorr()est ma fonction ci-dessous -
estimated_autocorrelationse trouve dans une autre réponse -
acf()est defrom statsmodels.tsa.stattools import acf
%timeit a0, junk, junk = gamma(a, f=0) # puwr.py
%timeit a1 = [acorr(a, m, i) for i in range(l)] # my own
%timeit a2 = acf(a) # statstools
%timeit a3 = estimated_autocorrelation(a) # numpy
%timeit a4 = acf(a, fft=True) # stats FFT
## -- End pasted text --
100 loops, best of 3: 7.18 ms per loop
100 loops, best of 3: 2.15 ms per loop
10 loops, best of 3: 88.3 ms per loop
10 loops, best of 3: 87.6 ms per loop
100 loops, best of 3: 3.33 ms per loop
Edit... J'ai vérifié à nouveau en gardant l=40 et en changeant n=10000 en n=200000 échantillons les méthodes FFT commencent à obtenir un peu de traction et statsmodels mise en œuvre FFT contours juste... (l'ordre est le même)
## -- End pasted text --
10 loops, best of 3: 86.2 ms per loop
10 loops, best of 3: 69.5 ms per loop
1 loops, best of 3: 16.2 s per loop
1 loops, best of 3: 16.3 s per loop
10 loops, best of 3: 52.3 ms per loop
Edit 2: j'ai changé ma routine et re-testé contre la FFT pour n=10000 et n=20000
a = correlatedData(n=200000); b=correlatedData(n=10000)
m = a.mean(); rng = np.arange(40); mb = b.mean()
%timeit a1 = map(lambda t:acorr(a, m, t), rng)
%timeit a1 = map(lambda t:acorr.acorr(b, mb, t), rng)
%timeit a4 = acf(a, fft=True)
%timeit a4 = acf(b, fft=True)
10 loops, best of 3: 73.3 ms per loop # acorr below
100 loops, best of 3: 2.37 ms per loop # acorr below
10 loops, best of 3: 79.2 ms per loop # statstools with FFT
100 loops, best of 3: 2.69 ms per loop # statstools with FFT
mise en Œuvre
def acorr(op_samples, mean, separation, norm = 1):
"""autocorrelation of a measured operator with optional normalisation
the autocorrelation is measured over the 0th axis
Required Inputs
op_samples :: np.ndarray :: the operator samples
mean :: float :: the mean of the operator
separation :: int :: the separation between HMC steps
norm :: float :: the autocorrelation with separation=0
"""
return ((op_samples[:op_samples.size-separation] - mean)*(op_samples[separation:]- mean)).ravel().mean() / norm
4x speedup peut être atteint ci-dessous. Vous devez faire attention de passer seulement op_samples=a.copy() car il va modifier le tableau a par a-=mean sinon:
op_samples -= mean
return (op_samples[:op_samples.size-separation]*op_samples[separation:]).ravel().mean() / norm
Contrôle De Santé
Exemple D'Analyse D'Erreur
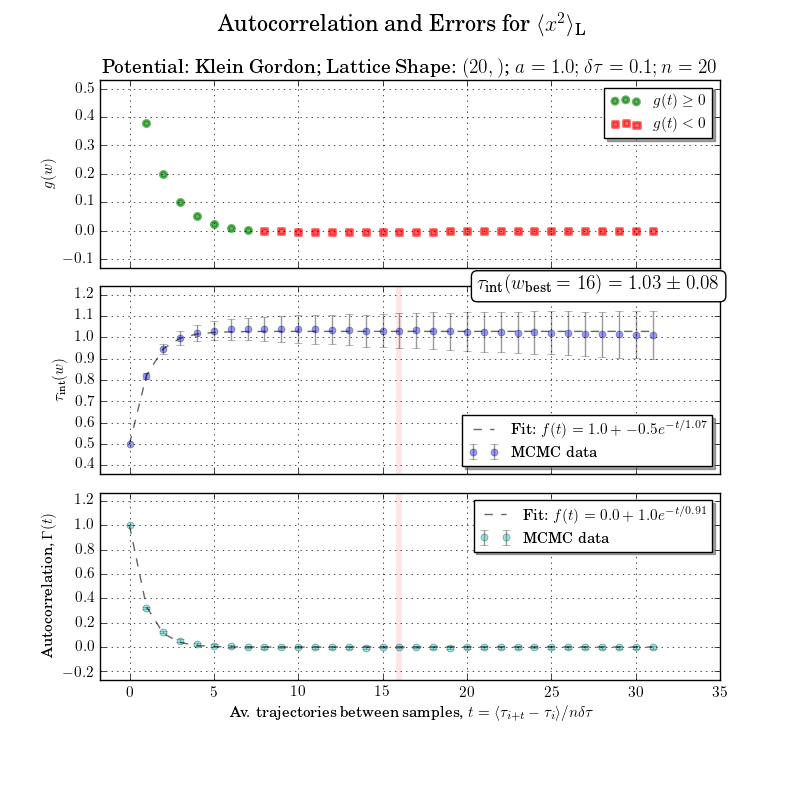
C'est un peu hors de portée, mais je ne peux pas être pris la peine de refaire le figure sans calcul du temps d'autocorrélation intégré ou de la fenêtre d'intégration. Les autocorrélations avec les erreurs sont claires dans le graphique du bas
j'ai trouvé cela obtenu les résultats attendus avec juste un léger changement:
def estimated_autocorrelation(x):
n = len(x)
variance = x.var()
x = x-x.mean()
r = N.correlate(x, x, mode = 'full')
result = r/(variance*n)
return result
test contre les résultats d'autocorrélation D'Excel.