Équivalent de la fonction qualité cluster de Matlab?
MATLAB has a nice fonction de la silhouette pour aider à évaluer le nombre de grappes de K-means. Est-il un équivalent pour Python Numpy/Scipy ainsi?
3 réponses
je vous présente ci-dessous un exemple silhouette mise en œuvre dans les deux MATLAB et Python/Numpy (gardez à l'esprit que je suis plus à l'aise dans MATLAB):
1) MATLAB
function s = mySilhouette(X, IDX)
%# X : matrix of size N-by-p, data where rows are instances
%# IDX: vector of size N, cluster index of each instance (starting from 1)
%# s : vector of size N, silhouette score value of each instance
N = size(X,1); %# number of instances
K = numel(unique(IDX)); %# number of clusters
%# compute pairwise distance matrix
D = squareform( pdist(X,'euclidean').^2 );
%# indices belonging to each cluster
kIndices = accumarray(IDX, 1:N, [K 1], @(x){sort(x)});
%# compute a,b,s for each instance
%# a(i): average distance from i to all other data within the same cluster.
%# b(i): lowest average dist from i to the data of another single cluster
a = zeros(N,1);
b = zeros(N,1);
for i=1:N
ind = kIndices{IDX(i)}; ind = ind(ind~=i);
a(i) = mean( D(i,ind) );
b(i) = min( cellfun(@(ind) mean(D(i,ind)), kIndices([1:K]~=IDX(i))) );
end
s = (b-a) ./ max(a,b);
end
Pour émuler le tracé de la silhouette fonction dans MATLAB, nous groupons les valeurs de silhouette par groupe, trions à l'intérieur de chacun, puis traçons les barres horizontalement. MATLAB ajoute NaNs pour séparer les barres des différents clusters, j'ai trouvé plus facile de simplement colorier le code bars:
%# sample data
load fisheriris
X = meas;
N = size(X,1);
%# cluster and compute silhouette score
K = 3;
[IDX,C] = kmeans(X, K, 'distance','sqEuclidean');
s = mySilhouette(X, IDX);
%# plot
[~,ord] = sortrows([IDX s],[1 -2]);
indices = accumarray(IDX(ord), 1:N, [K 1], @(x){sort(x)});
ytick = cellfun(@(ind) (min(ind)+max(ind))/2, indices);
ytickLabels = num2str((1:K)','%d'); %#'
h = barh(1:N, s(ord),'hist');
set(h, 'EdgeColor','none', 'CData',IDX(ord))
set(gca, 'CLim',[1 K], 'CLimMode','manual')
set(gca, 'YDir','reverse', 'YTick',ytick, 'YTickLabel',ytickLabels)
xlabel('Silhouette Value'), ylabel('Cluster')
%# compare against SILHOUETTE
figure, silhouette(X,IDX)
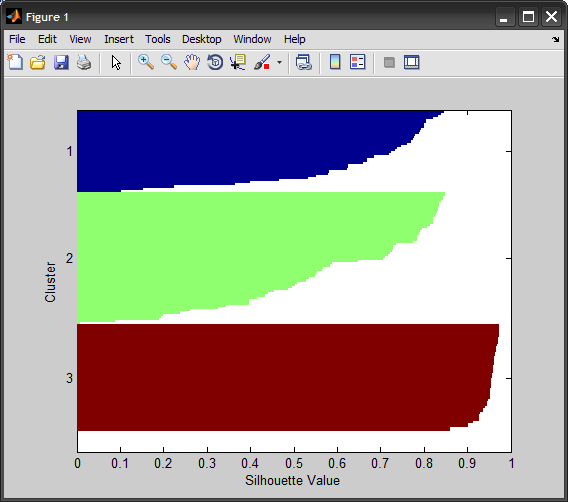
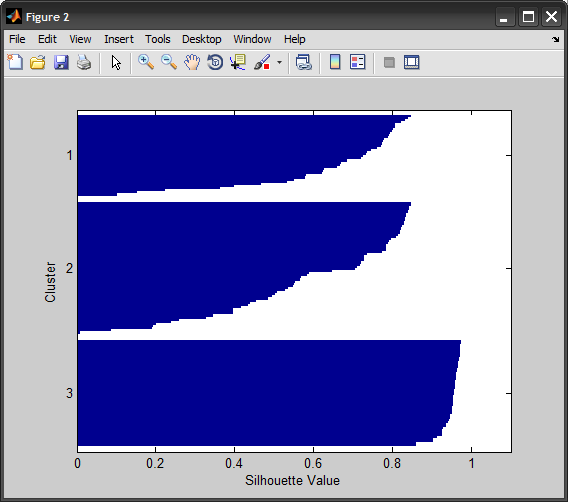
2) Python
Et voici ce que j'ai trouvé en Python:
import numpy as np
from scipy.cluster.vq import kmeans2
from scipy.spatial.distance import pdist, squareform
from sklearn import datasets
import matplotlib.pyplot as plt
from matplotlib import cm
def silhouette(X, cIDX):
"""
Computes the silhouette score for each instance of a clustered dataset,
which is defined as:
s(i) = (b(i)-a(i)) / max{a(i),b(i)}
with:
-1 <= s(i) <= 1
Args:
X : A M-by-N array of M observations in N dimensions
cIDX : array of len M containing cluster indices (starting from zero)
Returns:
s : silhouette value of each observation
"""
N = X.shape[0] # number of instances
K = len(np.unique(cIDX)) # number of clusters
# compute pairwise distance matrix
D = squareform(pdist(X))
# indices belonging to each cluster
kIndices = [np.flatnonzero(cIDX==k) for k in range(K)]
# compute a,b,s for each instance
a = np.zeros(N)
b = np.zeros(N)
for i in range(N):
# instances in same cluster other than instance itself
a[i] = np.mean( [D[i][ind] for ind in kIndices[cIDX[i]] if ind!=i] )
# instances in other clusters, one cluster at a time
b[i] = np.min( [np.mean(D[i][ind])
for k,ind in enumerate(kIndices) if cIDX[i]!=k] )
s = (b-a)/np.maximum(a,b)
return s
def main():
# load Iris dataset
data = datasets.load_iris()
X = data['data']
# cluster and compute silhouette score
K = 3
C, cIDX = kmeans2(X, K)
s = silhouette(X, cIDX)
# plot
order = np.lexsort((-s,cIDX))
indices = [np.flatnonzero(cIDX[order]==k) for k in range(K)]
ytick = [(np.max(ind)+np.min(ind))/2 for ind in indices]
ytickLabels = ["%d" % x for x in range(K)]
cmap = cm.jet( np.linspace(0,1,K) ).tolist()
clr = [cmap[i] for i in cIDX[order]]
fig = plt.figure()
ax = fig.add_subplot(111)
ax.barh(range(X.shape[0]), s[order], height=1.0,
edgecolor='none', color=clr)
ax.set_ylim(ax.get_ylim()[::-1])
plt.yticks(ytick, ytickLabels)
plt.xlabel('Silhouette Value')
plt.ylabel('Cluster')
plt.show()
if __name__ == '__main__':
main()
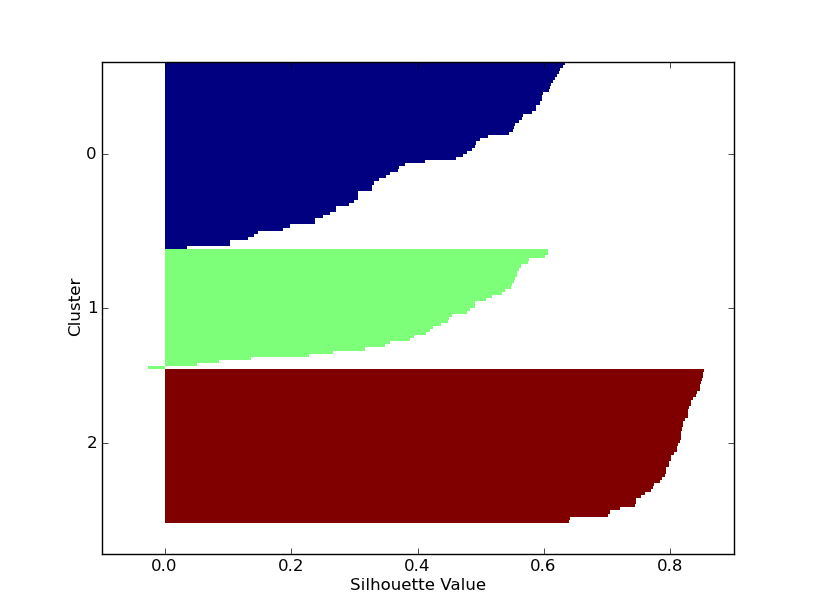
mise à Jour:
comme d'autres l'ont noté, scikit-learn a depuis ajouté son propre silhouette métrique mise en oeuvre. Pour l'utiliser dans le code ci-dessus, remplacez l'appel à l'personnalisé silhouette fonction:
from sklearn.metrics import silhouette_samples
...
#s = silhouette(X, cIDX)
s = silhouette_samples(X, cIDX) # <-- scikit-learn function
...
le reste du code peut encore être utilisé comme-est de générer exactement la même parcelle.
j'ai cherché, mais je ne trouve pas de fonction de silhouette en forme de pépin, j'ai même cherché dans pylab et matplotlib. Je pense que tu devras le mettre en œuvre toi-même.
je peux point vous à http://orange.biolab.si/trac/browser/trunk/orange/orngClustering.py?rev=7462. Il a quelques fonctions qui implémentent une silhouette fonction.
J'espère que cela vous aidera.
C'est un peu tard, mais pour ce qu'il vaut, il semble que scikits-apprendre implémente maintenant une silhouette fonction. Voir leur page de documentation ou afficher les code source directement.