Obtenez efficacement des indices de bins d'histogramme en Python
Question Courte
J'ai une grande image 10000x10000 éléments, que je range dans quelques centaines de secteurs/bacs différents. J'ai ensuite besoin d'effectuer un calcul itératif sur les valeurs contenues dans chaque bac.
Comment extraire les indices de chaque bac pour effectuer efficacement mon calcul en utilisant les valeurs des bacs?
Ce que je cherche est une solution qui évite le goulot d'étranglement d'avoir à sélectionner chaque fois ind == j de mon grand tableau. Est-il un moyen de obtenir directement, en une seule fois, les indices des éléments appartenant à chaque bin?
Explication Détaillée
1. Solution Simple
Une façon de réaliser ce dont j'ai besoin est d'utiliser un code comme celui-ci (Voir par exemple cette réponse connexe), où je numérise mes valeurs, puis j'ai une boucle j sélectionnant des indices numérisés égaux à j comme ci-dessous
import numpy as np
# This function func() is just a place mark for a much more complicated function.
# I am aware that my problem could be easily speed up in the specific case of
# of the sum() function, but I am looking for a general solution to the problem.
def func(x):
y = np.sum(x)
return y
vals = np.random.random(1e8)
nbins = 100
bins = np.linspace(0, 1, nbins+1)
ind = np.digitize(vals, bins)
result = [func(vals[ind == j]) for j in range(1, nbins)]
Ce que je cherche est une solution qui évite le goulot d'étranglement d'avoir à sélectionner à chaque fois ind == j de mon grand tableau. Est-il possible d'obtenir directement, en une seule fois, les indices des éléments appartenant à chaque bin?
2. Utilisation de binned_statistics
L'approche ci-dessus s'avère être la même implémentée dans scipy.statistique.binned_statistic, pour le cas général d'une fonction définie par l'utilisateur. En utilisant Scipy directement, une sortie identique peut être obtenue avec
import numpy as np
from scipy.stats import binned_statistics
vals = np.random.random(1e8)
results = binned_statistic(vals, vals, statistic=func, bins=100, range=[0, 1])[0]
3. Utilisation de labeled_comprehension
Une autre alternative de Scipy est d'utiliser scipy.ndimage.mesure.labeled_comprehension . En utilisant cette fonction, l'exemple ci-dessus deviendrait
import numpy as np
from scipy.ndimage import labeled_comprehension
vals = np.random.random(1e8)
nbins = 100
bins = np.linspace(0, 1, nbins+1)
ind = np.digitize(vals, bins)
result = labeled_comprehension(vals, ind, np.arange(1, nbins), func, float, 0)
Malheureusement aussi ce formulaire est inefficace et en particulier il n'a aucun avantage de vitesse sur mon exemple original.
4. Comparaison avec la langue IDL
Pour clarifier davantage, ce que je cherche est une fonctionnalité équivalente au mot-clé REVERSE_INDICES dans la fonction HISTOGRAM du langage IDL ici. Cette fonctionnalité très utile peut-elle être efficacement répliqué en Python?
Plus précisément, en utilisant le langage IDL, l'exemple ci-dessus pourrait être écrit comme
vals = randomu(s, 1e8)
nbins = 100
bins = [0:1:1./nbins]
h = histogram(vals, MIN=bins[0], MAX=bins[-2], NBINS=nbins, REVERSE_INDICES=r)
result = dblarr(nbins)
for j=0, nbins-1 do begin
jbins = r[r[j]:r[j+1]-1] ; Selects indices of bin j
result[j] = func(vals[jbins])
endfor
L'implémentation IDL ci-dessus est environ 10 fois plus rapide que celle Numpy, en raison du fait que les indices des bins n'ont pas besoin d'être sélectionnés pour chaque bac. Et la différence de vitesse en faveur de L'implémentation IDL augmente avec le nombre de bacs.
5 réponses
J'ai trouvé qu'un constructeur particulier de matrice clairsemée peut atteindre le résultat souhaité très efficacement. C'est un peu obscur mais nous pouvons en abuser à cette fin. La fonction ci-dessous peut être utilisée de la même manière que scipy.statistique.binned_statistic mais peut être plus rapide
import numpy as np
from scipy.sparse import csr_matrix
def binned_statistic(x, values, func, nbins, range):
'''The usage is nearly the same as scipy.stats.binned_statistic'''
N = len(values)
r0, r1 = range
digitized = (float(nbins)/(r1 - r0)*(x - r0)).astype(int)
S = csr_matrix((values, [digitized, np.arange(N)]), shape=(nbins, N))
return [func(group) for group in np.split(S.data, S.indptr[1:-1])]
J'ai évité np.digitize car il n'utilise pas le fait que tous les bacs sont de largeur égale et donc lents, mais la méthode que j'ai utilisée à la place peut ne pas gérer parfaitement tous les cas de bord.
Je suppose que le binning, fait dans l'exemple avec digitize, ne peut pas être modifié. C'est une façon d'aller, où vous faire le tri une fois pour toutes.
vals = np.random.random(1e4)
nbins = 100
bins = np.linspace(0, 1, nbins+1)
ind = np.digitize(vals, bins)
new_order = argsort(ind)
ind = ind[new_order]
ordered_vals = vals[new_order]
# slower way of calculating first_hit (first version of this post)
# _,first_hit = unique(ind,return_index=True)
# faster way:
first_hit = searchsorted(ind,arange(1,nbins-1))
first_hit.sort()
#example of using the data:
for j in range(nbins-1):
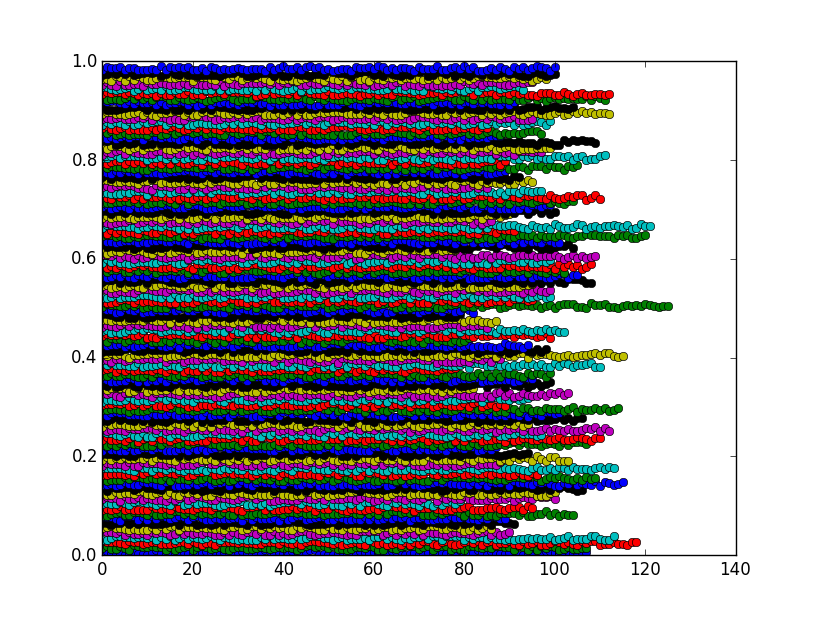
#I am using a plotting function for your f, to show that they cluster
plot(ordered_vals[first_hit[j]:first_hit[j+1]],'o')
La figure montre que les bacs sont en fait des grappes comme prévu:
Vous pouvez réduire de moitié le temps de calcul en triant d'abord le tableau, puis en utilisant np.searchsorted.
vals = np.random.random(1e8)
vals.sort()
nbins = 100
bins = np.linspace(0, 1, nbins+1)
ind = np.digitize(vals, bins)
results = [func(vals[np.searchsorted(ind,j,side='left'):
np.searchsorted(ind,j,side='right')])
for j in range(1,nbins)]
En utilisant 1e8 comme cas de test, je passe de 34 secondes de calcul à environ 17.
Une solution efficace utilise le paquetnumpy_indexed (avertissement: je suis son auteur):
import numpy_indexed as npi
npi.group_by(ind).split(vals)
Pandas a un code de regroupement très rapide (je pense qu'il est écrit en C), donc si cela ne vous dérange pas de charger la bibliothèque, vous pouvez le faire:
import pandas as pd
pdata=pd.DataFrame({'vals':vals,'ind':ind})
resultsp = pdata.groupby('ind').sum().values
Ou plus généralement :
pdata=pd.DataFrame({'vals':vals,'ind':ind})
resultsp = pdata.groupby('ind').agg(func).values
Bien que ce dernier soit plus lent pour les fonctions d'agrégation standard (comme somme, moyenne, etc)