Moyen efficace de rbind données.cadres avec différentes colonnes
J'ai une liste de trames de données avec un ensemble différent de colonnes, je voudrais les combiner par lignes en une seule trame de données. J'utilise plyr::rbind.fill pour le faire. Je cherche quelque chose qui le ferait plus efficacement.
Similaire à la réponse donnée ici
require(plyr)
set.seed(45)
sample.fun <- function() {
nam <- sample(LETTERS, sample(5:15))
val <- data.frame(matrix(sample(letters, length(nam)*10,replace=TRUE),nrow=10))
setNames(val, nam)
}
ll <- replicate(1e4, sample.fun())
rbind.fill(ll)
3 réponses
UPDATE: Voircette réponse mise à jour à la place.
UPDATE (eddi): ceci a maintenant été implémenté dans version 1.8.11 comme argument fill pour rbind. Par exemple:
DT1 = data.table(a = 1:2, b = 1:2)
DT2 = data.table(a = 3:4, c = 1:2)
rbind(DT1, DT2, fill = TRUE)
# a b c
#1: 1 1 NA
#2: 2 2 NA
#3: 3 NA 1
#4: 4 NA 2
FR # 4790 Ajouté maintenant-rbind.remplir (de plyr) comme fonctionnalité pour fusionner la liste des données.images/données.tableaux
Remarque 1:
Cette solution utilise la fonction rbindlist de data.table pour "rbind" liste de données.tables et pour cela, assurez-vous de utilisez la version 1.8.9 à cause de Ce bogue dans les versions .
Remarque 2:
rbindlist lors de la liaison des listes de données.images/données.les tables, à partir de Maintenant, conserveront le type de données de la première colonne. Autrement dit, si une colonne dans les premières données.frame est un caractère et la même colonne dans les 2ème données.frame est "factor", alors, rbindlist aura comme conséquence que cette colonne est un caractère. Donc, si vos données.frame se composait de toutes les colonnes de caractères, alors, votre solution avec cette méthode sera identique à la méthode plyr. Si non, les valeurs seront toujours les mêmes, mais certaines colonnes seront caractère au lieu de facteur. Vous devrez vous convertir en" facteur " après. espérons que ce comportement changera à l'avenir.
Et maintenant, voici en utilisant data.table (et la comparaison de benchmarking avec rbind.fill de plyr):
require(data.table)
rbind.fill.DT <- function(ll) {
# changed sapply to lapply to return a list always
all.names <- lapply(ll, names)
unq.names <- unique(unlist(all.names))
ll.m <- rbindlist(lapply(seq_along(ll), function(x) {
tt <- ll[[x]]
setattr(tt, 'class', c('data.table', 'data.frame'))
data.table:::settruelength(tt, 0L)
invisible(alloc.col(tt))
tt[, c(unq.names[!unq.names %chin% all.names[[x]]]) := NA_character_]
setcolorder(tt, unq.names)
}))
}
rbind.fill.PLYR <- function(ll) {
rbind.fill(ll)
}
require(microbenchmark)
microbenchmark(t1 <- rbind.fill.DT(ll), t2 <- rbind.fill.PLYR(ll), times=10)
# Unit: seconds
# expr min lq median uq max neval
# t1 <- rbind.fill.DT(ll) 10.8943 11.02312 11.26374 11.34757 11.51488 10
# t2 <- rbind.fill.PLYR(ll) 121.9868 134.52107 136.41375 184.18071 347.74724 10
# for comparison change t2 to data.table
setattr(t2, 'class', c('data.table', 'data.frame'))
data.table:::settruelength(t2, 0L)
invisible(alloc.col(t2))
setcolorder(t2, unique(unlist(sapply(ll, names))))
identical(t1, t2) # [1] TRUE
Il convient de noter que plyr rbind.fill dépasse cette solution data.table jusqu'à une taille de liste d'environ 500.
Benchmarking terrain:
Voici l'intrigue sur les courses avec la longueur de la liste des données.cadres avec seq(1000, 10000, by=1000). J'ai utilisé microbenchmark avec 10 représentants sur chacune de ces différentes longueurs de liste.
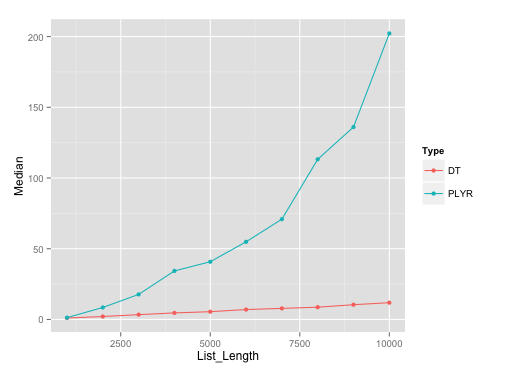
Analyse comparative de l'essentiel:
Voici l'essentiel de l'analyse comparative , au cas où quelqu'un voudrait reproduire les résultats.
Maintenant que rbindlist (et rbind) pour data.table a amélioré la fonctionnalité et la vitesse avec les modifications/commits récents dans v1.9.3 (version de développement), et dplyr a une version plus rapide de plyr's rbind.fill, nommé rbind_all, cette réponse semble un peu trop dépassée.
Voici l'entrée de nouvelles pertinente pour rbindlist:
o 'rbindlist' gains 'use.names' and 'fill' arguments and is now implemented entirely in C. Closes #5249
-> use.names by default is FALSE for backwards compatibility (doesn't bind by
names by default)
-> rbind(...) now just calls rbindlist() internally, except that 'use.names'
is TRUE by default, for compatibility with base (and backwards compatibility).
-> fill by default is FALSE. If fill is TRUE, use.names has to be TRUE.
-> At least one item of the input list has to have non-null column names.
-> Duplicate columns are bound in the order of occurrence, like base.
-> Attributes that might exist in individual items would be lost in the bound result.
-> Columns are coerced to the highest SEXPTYPE, if they are different, if/when possible.
-> And incredibly fast ;).
-> Documentation updated in much detail. Closes DR #5158.
Donc, j'ai comparé les versions plus récentes (et plus rapides) sur des données relativement plus grandes ci-dessous.
Nouvelle Référence:
Nous allons créez un total de 10 000 données.tables avec des colonnes allant de 200-300 avec le nombre total de colonnes après la liaison à 500.
Fonctions pour créer des données:
require(data.table) ## 1.9.3 commit 1267
require(dplyr) ## commit 1504 devel
set.seed(1L)
names = paste0("V", 1:500)
foo <- function() {
cols = sample(200:300, 1)
data = setDT(lapply(1:cols, function(x) sample(10)))
setnames(data, sample(names)[1:cols])
}
n = 10e3L
ll = vector("list", n)
for (i in 1:n) {
.Call("Csetlistelt", ll, i, foo())
}
Et voici les horaires:
## Updated timings on data.table v1.9.5 - three consecutive runs:
system.time(ans1 <- rbindlist(ll, fill=TRUE))
# user system elapsed
# 1.993 0.106 2.107
system.time(ans1 <- rbindlist(ll, fill=TRUE))
# user system elapsed
# 1.644 0.092 1.744
system.time(ans1 <- rbindlist(ll, fill=TRUE))
# user system elapsed
# 1.297 0.088 1.389
## dplyr's rbind_all - Timings for three consecutive runs
system.time(ans2 <- rbind_all(ll))
# user system elapsed
# 9.525 0.121 9.761
# user system elapsed
# 9.194 0.112 9.370
# user system elapsed
# 8.665 0.081 8.780
identical(ans1, setDT(ans2)) # [1] TRUE
Il y a encore quelque chose à gagner si vous parallélisez à la fois rbind.fill et rbindlist.
Les résultats sont faits avec data.table version 1.8.8 car la version 1.8.9 a été briquée quand je l'ai essayé avec la fonction parallélisée. Les résultats ne sont donc pas identiques entre data.table et plyr, mais ils sont identiques dans la solution data.table ou plyr. Sens parallèle plyr correspond à l'incomparable plyr, et vice versa.
Voici Le benchmark / scripts. Le parallel.rbind.fill.DT semble horrible, mais c'est le plus rapide que j'ai pu tirer.
require(plyr)
require(data.table)
require(ggplot2)
require(rbenchmark)
require(parallel)
# data.table::rbindlist solutions
rbind.fill.DT <- function(ll) {
all.names <- lapply(ll, names)
unq.names <- unique(unlist(all.names))
rbindlist(lapply(seq_along(ll), function(x) {
tt <- ll[[x]]
setattr(tt, 'class', c('data.table', 'data.frame'))
data.table:::settruelength(tt, 0L)
invisible(alloc.col(tt))
tt[, c(unq.names[!unq.names %chin% all.names[[x]]]) := NA_character_]
setcolorder(tt, unq.names)
}))
}
parallel.rbind.fill.DT <- function(ll, cluster=NULL){
all.names <- lapply(ll, names)
unq.names <- unique(unlist(all.names))
if(is.null(cluster)){
ll.m <- rbindlist(lapply(seq_along(ll), function(x) {
tt <- ll[[x]]
setattr(tt, 'class', c('data.table', 'data.frame'))
data.table:::settruelength(tt, 0L)
invisible(alloc.col(tt))
tt[, c(unq.names[!unq.names %chin% all.names[[x]]]) := NA_character_]
setcolorder(tt, unq.names)
}))
}else{
cores <- length(cluster)
sequ <- as.integer(seq(1, length(ll), length.out = cores+1))
Call <- paste(paste("list", seq(cores), sep=""), " = ll[", c(1, sequ[2:cores]+1), ":", sequ[2:(cores+1)], "]", sep="", collapse=", ")
ll <- eval(parse(text=paste("list(", Call, ")")))
rbindlist(clusterApply(cluster, ll, function(ll, unq.names){
rbindlist(lapply(seq_along(ll), function(x, ll, unq.names) {
tt <- ll[[x]]
setattr(tt, 'class', c('data.table', 'data.frame'))
data.table:::settruelength(tt, 0L)
invisible(alloc.col(tt))
tt[, c(unq.names[!unq.names %chin% colnames(tt)]) := NA_character_]
setcolorder(tt, unq.names)
}, ll=ll, unq.names=unq.names))
}, unq.names=unq.names))
}
}
# plyr::rbind.fill solutions
rbind.fill.PLYR <- function(ll) {
rbind.fill(ll)
}
parallel.rbind.fill.PLYR <- function(ll, cluster=NULL, magicConst=400){
if(is.null(cluster) | ceiling(length(ll)/magicConst) < length(cluster)){
rbind.fill(ll)
}else{
cores <- length(cluster)
sequ <- as.integer(seq(1, length(ll), length.out = ceiling(length(ll)/magicConst)))
Call <- paste(paste("list", seq(cores), sep=""), " = ll[", c(1, sequ[2:(length(sequ)-1)]+1), ":", sequ[2:length(sequ)], "]", sep="", collapse=", ")
ll <- eval(parse(text=paste("list(", Call, ")")))
rbind.fill(parLapply(cluster, ll, rbind.fill))
}
}
# Function to generate sample data of varying list length
set.seed(45)
sample.fun <- function() {
nam <- sample(LETTERS, sample(5:15))
val <- data.frame(matrix(sample(letters, length(nam)*10,replace=TRUE),nrow=10))
setNames(val, nam)
}
ll <- replicate(10000, sample.fun())
cl <- makeCluster(4, type="SOCK")
clusterEvalQ(cl, library(data.table))
clusterEvalQ(cl, library(plyr))
benchmark(t1 <- rbind.fill.PLYR(ll),
t2 <- rbind.fill.DT(ll),
t3 <- parallel.rbind.fill.PLYR(ll, cluster=cl, 400),
t4 <- parallel.rbind.fill.DT(ll, cluster=cl),
replications=5)
stopCluster(cl)
# Results for rbinding 10000 dataframes
# done with 4 cores, i5 3570k and 16gb memory
# test reps elapsed relative
# rbind.fill.PLYR 5 321.80 16.682
# rbind.fill.DT 5 26.10 1.353
# parallel.rbind.fill.PLYR 5 28.00 1.452
# parallel.rbind.fill.DT 5 19.29 1.000
# checking are results equal
t1 <- as.matrix(t1)
t2 <- as.matrix(t2)
t3 <- as.matrix(t3)
t4 <- as.matrix(t4)
t1 <- t1[order(t1[, 1], t1[, 2]), ]
t2 <- t2[order(t2[, 1], t2[, 2]), ]
t3 <- t3[order(t3[, 1], t3[, 2]), ]
t4 <- t4[order(t4[, 1], t4[, 2]), ]
identical(t2, t4) # TRUE
identical(t1, t3) # TRUE
identical(t1, t2) # FALSE, mismatch between plyr and data.table
Comme vous pouvez le voir parallesizing rbind.fill l'a rendu comparable à data.table, et vous pourriez obtenir une augmentation marginale de la vitesse en parallesizing data.table même avec ce bas d'un nombre de dataframe.