Construire une matrice de contiguïté dans MATLAB
tenir compte d'un ensemble de points disposés sur une grille de taille N-par-M. J'essaie de construire la matrice de voisinage de telle sorte que les points voisins sont connectés.
Par exemple, dans une grille 3x3 avec un graphique:
1-2-3
| | |
4-5-6
| | |
7-8-9
nous devrions avoir la matrice de contiguïté correspondante:
+---+------------------------------------------------------+
| | 1 2 3 4 5 6 7 8 9 |
+---+------------------------------------------------------+
| 1 | 0 1 0 1 0 0 0 0 0 |
| 2 | 1 0 1 0 1 0 0 0 0 |
| 3 | 0 1 0 0 0 1 0 0 0 |
| 4 | 1 0 0 0 1 0 1 0 0 |
| 5 | 0 1 0 1 0 1 0 1 0 |
| 6 | 0 0 1 0 1 0 0 0 1 |
| 7 | 0 0 0 1 0 0 0 1 0 |
| 8 | 0 0 0 0 1 0 1 0 1 |
| 9 | 0 0 0 0 0 1 0 1 0 |
+---+------------------------------------------------------+
en bonus, la solution devrait fonctionner pour les points voisins connectés 4 et 8, c'est-à-dire:
o o o o
o X o vs. o X o
o o o o
le code que j'ai donc loin:
N = 3; M = 3;
adj = zeros(N*M);
for i=1:N
for j=1:M
k = sub2ind([N M],i,j);
if i>1
ii=i-1; jj=j;
adj(k,sub2ind([N M],ii,jj)) = 1;
end
if i<N
ii=i+1; jj=j;
adj(k,sub2ind([N M],ii,jj)) = 1;
end
if j>1
ii=i; jj=j-1;
adj(k,sub2ind([N M],ii,jj)) = 1;
end
if j<M
ii=i; jj=j+1;
adj(k,sub2ind([N M],ii,jj)) = 1;
end
end
end
comment cela peut-il être amélioré pour éviter toute la boucle?
6 réponses
si vous remarquez, il y a un motif distinct dans les matrices de contiguïté que vous créez. Plus précisément, ils sont symétriques et bandes. Vous pouvez profiter de ce fait pour créer facilement vos matrices en utilisant le diag fonction (ou spdiags fonction de si vous voulez faire une matrice creuse). Voici comment vous pouvez créer la matrice de contiguïté pour chaque cas, en utilisant votre matrice d'échantillon ci-dessus comme exemple:
4-connectés voisins:
mat = [1 2 3; 4 5 6; 7 8 9]; % Sample matrix
[r, c] = size(mat); % Get the matrix size
diagVec1 = repmat([ones(c-1, 1); 0], r, 1); % Make the first diagonal vector
% (for horizontal connections)
diagVec1 = diagVec1(1:end-1); % Remove the last value
diagVec2 = ones(c*(r-1), 1); % Make the second diagonal vector
% (for vertical connections)
adj = diag(diagVec1, 1)+diag(diagVec2, c); % Add the diagonals to a zero matrix
adj = adj+adj.'; % Add the matrix to a transposed copy of
% itself to make it symmetric
Et vous obtiendrez le tableau suivant:
adj =
0 1 0 1 0 0 0 0 0
1 0 1 0 1 0 0 0 0
0 1 0 0 0 1 0 0 0
1 0 0 0 1 0 1 0 0
0 1 0 1 0 1 0 1 0
0 0 1 0 1 0 0 0 1
0 0 0 1 0 0 0 1 0
0 0 0 0 1 0 1 0 1
0 0 0 0 0 1 0 1 0
8-connecté voisins:
mat = [1 2 3; 4 5 6; 7 8 9]; % Sample matrix
[r, c] = size(mat); % Get the matrix size
diagVec1 = repmat([ones(c-1, 1); 0], r, 1); % Make the first diagonal vector
% (for horizontal connections)
diagVec1 = diagVec1(1:end-1); % Remove the last value
diagVec2 = [0; diagVec1(1:(c*(r-1)))]; % Make the second diagonal vector
% (for anti-diagonal connections)
diagVec3 = ones(c*(r-1), 1); % Make the third diagonal vector
% (for vertical connections)
diagVec4 = diagVec2(2:end-1); % Make the fourth diagonal vector
% (for diagonal connections)
adj = diag(diagVec1, 1)+... % Add the diagonals to a zero matrix
diag(diagVec2, c-1)+...
diag(diagVec3, c)+...
diag(diagVec4, c+1);
adj = adj+adj.'; % Add the matrix to a transposed copy of
% itself to make it symmetric
Et vous obtiendrez le tableau suivant:
adj =
0 1 0 1 1 0 0 0 0
1 0 1 1 1 1 0 0 0
0 1 0 0 1 1 0 0 0
1 1 0 0 1 0 1 1 0
1 1 1 1 0 1 1 1 1
0 1 1 0 1 0 0 1 1
0 0 0 1 1 0 0 1 0
0 0 0 1 1 1 1 0 1
0 0 0 0 1 1 0 1 0
juste pour le plaisir, voici une solution pour construire la matrice de contiguïté en calculant la distance entre toutes les paires de points sur la grille (pas la manière la plus efficace évidemment)
N = 3; M = 3; %# grid size
CONNECTED = 8; %# 4-/8- connected points
%# which distance function
if CONNECTED == 4, distFunc = 'cityblock';
elseif CONNECTED == 8, distFunc = 'chebychev'; end
%# compute adjacency matrix
[X Y] = meshgrid(1:N,1:M);
X = X(:); Y = Y(:);
adj = squareform( pdist([X Y], distFunc) == 1 );
et voici un peu de code pour visualiser la matrice de contiguïté et le graphe des points connectés:
%# plot adjacency matrix
subplot(121), spy(adj)
%# plot connected points on grid
[xx yy] = gplot(adj, [X Y]);
subplot(122), plot(xx, yy, 'ks-', 'MarkerFaceColor','r')
axis([0 N+1 0 M+1])
%# add labels
[X Y] = meshgrid(1:N,1:M);
X = reshape(X',[],1) + 0.1; Y = reshape(Y',[],1) + 0.1;
text(X, Y(end:-1:1), cellstr(num2str((1:N*M)')) )
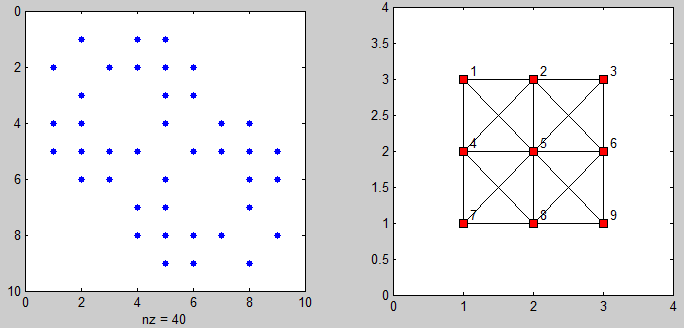
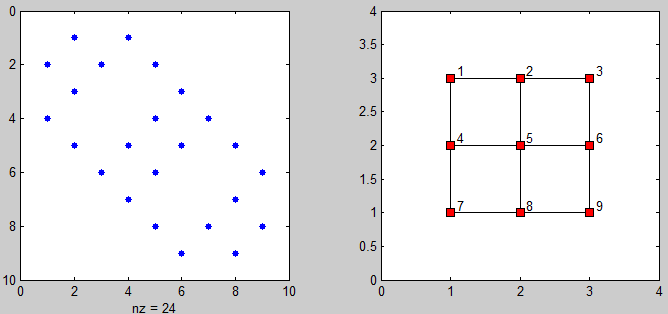
je viens de trouver cette question en cherchant le même problème. Cependant, aucune des solutions proposées n'a fonctionné pour moi en raison de la taille du problème qui nécessitait l'utilisation de types de matrice épars. Voici ma solution qui fonctionne sur des instances à grande échelle:
function W = getAdjacencyMatrix(I)
[m, n] = size(I);
I_size = m*n;
% 1-off diagonal elements
V = repmat([ones(m-1,1); 0],n, 1);
V = V(1:end-1); % remove last zero
% n-off diagonal elements
U = ones(m*(n-1), 1);
% get the upper triangular part of the matrix
W = sparse(1:(I_size-1), 2:I_size, V, I_size, I_size)...
+ sparse(1:(I_size-m),(m+1):I_size, U, I_size, I_size);
% finally make W symmetric
W = W + W';
Votre code actuel ne semble pas si mal. D'une façon ou d'une autre, vous devez itérer sur tous les paires de voisins. Si vous avez vraiment besoin d'optimiser le code, je dirais:
- boucle sur le nœud indices i, où
1 <= i <= (N*M) - n'utilisez pas sub2ind () pour l'efficacité, les voisins de node i sont simpy
[i-M, i+1, i+M, i-1]dans le sens des aiguilles d'une montre
notez que pour obtenir toutes les paires de noeuds de voisinage:
- il suffit de calculer les "bons" voisins (i.e. bords horizontaux) pour les noeuds
i % M != 0(puisque Matlab n'est pas basé sur 0 mais sur 1) - vous n'avez qu'à calculer "au-dessus" des voisins (c.-à-d. les bords verticaux) pour les noeuds
i > M - il existe une règle similaire pour les bords diagonaux
ceci donnerait une boucle simple (mais le même nombre D'itérations N*M), n'appelle pas sub2ind (), et n'a que deux instructions if dans la boucle.
Viens de tomber sur cette question. J'ai un beau travail m-fonction (lien: sparse_adj_matrix.m) qui est assez général.
La fonction peut aussi connecter des noeuds plus loin que radius = 1.
Voici la signature de la fonction:
% Construct sparse adjacency matrix (provides ii and jj indices into the
% matrix)
%
% Usage:
% [ii jj] = sparse_adj_matrix(sz, r, p)
%
% inputs:
% sz - grid size (determine the number of variables n=prod(sz), and the
% geometry/dimensionality)
% r - the radius around each point for which edges are formed
% p - in what p-norm to measure the r-ball, can be 1,2 or 'inf'
%
% outputs
% ii, jj - linear indices into adjacency matrix (for each pair (m,n)
% there is also the pair (n,m))
%
% How to construct the adjacency matrix?
% >> A = sparse(ii, jj, ones(1,numel(ii)), prod(sz), prod(sz));
%
%
% Example:
% >> [ii jj] = sparse_adj_matrix([10 20], 1, inf);
% construct indices for 200x200 adjacency matrix for 8-connect graph over a
% grid of 10x20 nodes.
% To visualize the graph:
% >> [r c]=ndgrid(1:10,1:20);
% >> A = sparse(ii, jj, 1, 200, 200);;
% >> gplot(A, [r(:) c(:)]);
pour chaque noeud dans le graphe ajouter une connexion à droite et une vers le bas. Vérifiez que vous n'exagérez pas votre grille. Considérons la fonction suivante qui construit la matrice de contiguïté.
function adj = AdjMatrixLattice4( N, M )
% Size of adjacency matrix
MN = M*N;
adj = zeros(MN,MN);
% number nodes as such
% [1]---[2]-- .. --[M]
% | | |
% [M+1]-[M+2]- .. -[2*M]
% : : :
% [] [] .. [M*N]
for i=1:N
for j=1:N
A = M*(i-1)+j; %Node # for (i,j) node
if(j<N)
B = M*(i-1)+j+1; %Node # for node to the right
adj(A,B) = 1;
adj(B,A) = 1;
end
if(i<M)
B = M*i+j; %Node # for node below
adj(A,B) = 1;
adj(B,A) = 1;
end
end
end
end
l'Exemple ci-dessus AdjMatrixLattice4(3,3)=
0 1 0 1 0 0 0 0 0
1 0 1 0 1 0 0 0 0
0 1 0 0 0 1 0 0 0
1 0 0 0 1 0 1 0 0
0 1 0 1 0 1 0 1 0
0 0 1 0 1 0 0 0 1
0 0 0 1 0 0 0 1 0
0 0 0 0 1 0 1 0 1
0 0 0 0 0 1 0 1 0