Couleurs perdues dans le diagramme de dispersion 3D de Matplotlib
pour un projet, je fais quelques diagrammes de dispersion 3D avec les trois projections correspondantes sous elle. J'utilise différentes couleurs pour indiquer un quatrième paramètre. D'abord je trace des données avec une certaine couleur et ensuite je surclasse cela avec d'autres données avec une couleur différente, de sorte qu'à la fin l'ordre est tel que je peux voir tout ce que je veux:
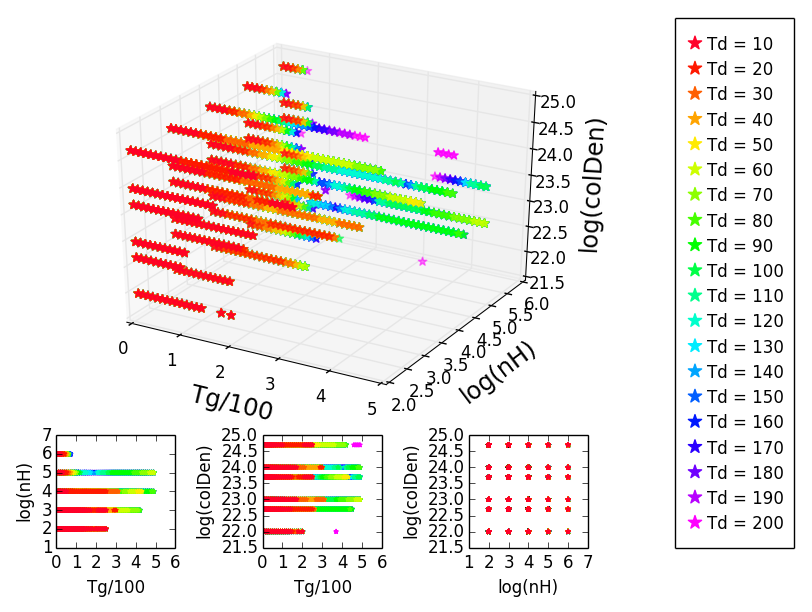
au début cela a bien fonctionné, mais quand j'essaie de faire la même chose avec des données légèrement différentes, les couleurs se gâtent. Les couleurs montrées dans les projections sont les bonnes, mais certaines d'entre elles sont absentes dans le graphique 3D de sorte qu'elles ne correspondent plus:
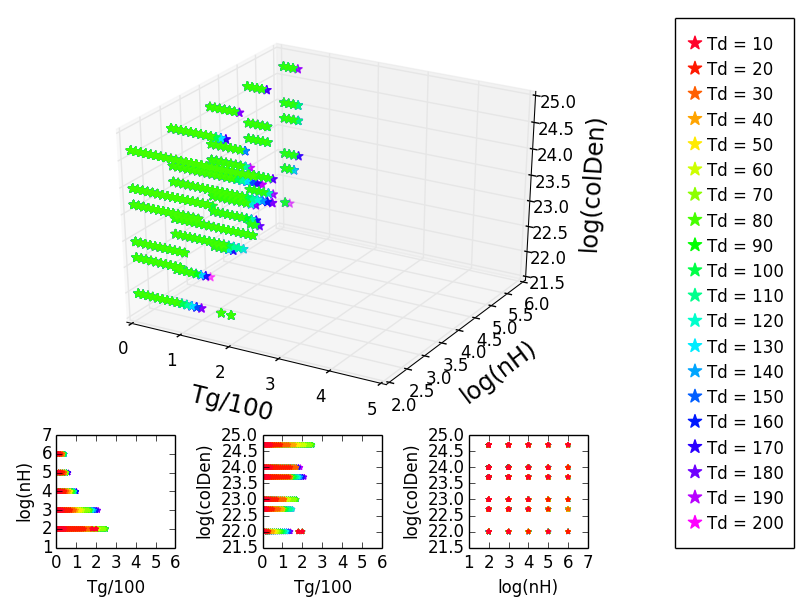
quand je tourne l'intrigue 3D d'une manière amusante, les couleurs sont récupérées et je peux les voir comme elles étaient censées être:
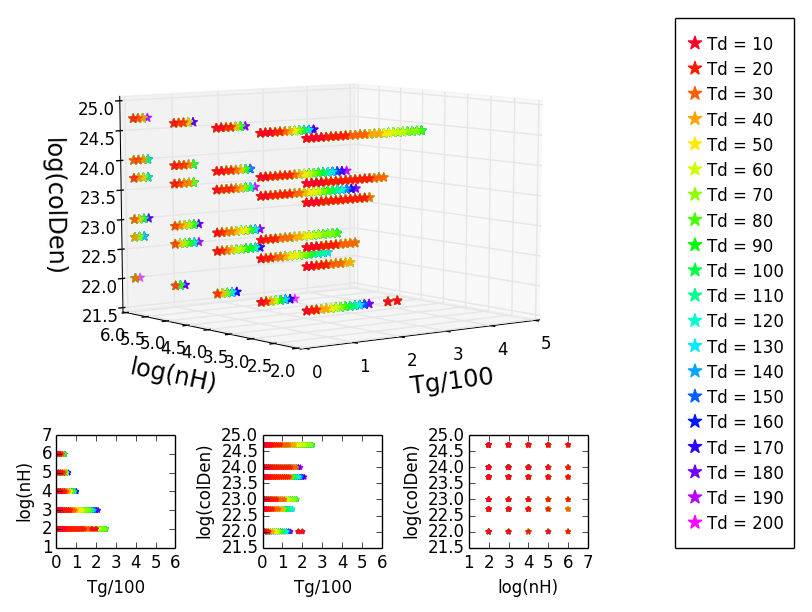
cependant, je ne veux pas d'un tracé en 3D cela tourne d'une manière amusante, parce que les haches se gâtent et il est impossible de le lire correctement comme cela.
j'ai trouvé une solution au problème ici: complot en 3d de la dispersion matplotlib . Ça dit que je dois remplacer ma hache.nuages de points(X,Y) avec ax.plot (X,Y,'o'). Lorsque je fais ceci, les couleurs sont affichées de la manière dont ils étaient censés être, mais l'intrigue est beaucoup plus problématique et la plus laide de cette façon. Fondamentalement, je veux juste être capable de le faire avec un nuage de points.
personne Ne sait comment résoudre ce problème?
voici un exemple minimum de mon code, pour seulement deux couleurs:
from mpl_toolkits.mplot3d import art3d
import numpy as np
from mpl_toolkits.mplot3d import Axes3D
import matplotlib.pyplot as plt
from matplotlib import gridspec
art3d.zalpha = lambda *args:args[0]
numcols = 20
percentage = 50
def load(Td, pc):
T = np.load(str(pc) + 'pctTemperaturesTd=' + str(Td) + '.npy')
D = np.load(str(pc) + 'pctDensitiesTd=' + str(Td) + '.npy')
CD = np.load(str(pc) + 'pctColDensitiesTd=' + str(Td) + '.npy')
return T, D, CD
def colors(ax):
colors = np.zeros((numcols, 4))
cm = plt.get_cmap('gist_rainbow')
ax.set_color_cycle([cm(1.*i/numcols) for i in range(numcols)])
for i in range(numcols):
color = cm(1.*i/numcols)
colors[i,:] = color
return colors
# LOAD DATA
T10, D10, CD10 = load(10, percentage)
T200, D200, CD200 = load(200, percentage)
# 3D PLOT
fig = plt.figure(1)
gs = gridspec.GridSpec(4, 4)
ax = fig.add_subplot(gs[:-1,:-1], projection='3d')
colours = colors(ax)
ax.plot(T200/100., np.log10(D200), np.log10(CD200), '*', markersize=10,color=colours[10], mec = colours[10], label='Td = 200', alpha=1)
ax.plot(T10/100., np.log10(D10), np.log10(CD10), '*', markersize=10,color=colours[0], mec = colours[0], label='Td = 10', alpha=1)
ax.set_xlabel('nTg/100', fontsize='x-large')
ax.set_ylabel('nlog(nH)', fontsize='x-large')
ax.set_zlabel('nlog(colDen)', fontsize='x-large')
ax.set_xlim(0,5)
#ax.set_zlim(0,)
ax.set_ylim(2,6)
# PROJECTIONS
# Tg, nH
ax2 = fig.add_subplot(gs[3,0])
ax2.scatter(T200/100., np.log10(D200), marker='*', s=10, color=colours[10], label='Td = 200', alpha=1, edgecolor=colours[10])
ax2.scatter(T10/100., np.log10(D10), marker='*', s=10, color=colours[0], label='Td = 10', alpha=1, edgecolor=colours[0])
ax2.set_xlabel('Tg/100')
ax2.set_ylabel('log(nH)')
ax2.set_xlim(0,6)
# Tg, colDen
ax3 = fig.add_subplot(gs[3,1])
ax3.scatter(T200/100., np.log10(CD200), marker='*', s=10, color=colours[10], label='Td = 200', alpha=1, edgecolor=colours[10])
ax3.scatter(T10/100., np.log10(CD10), marker='*', s=10, color=colours[0], label='Td = 10', alpha=1, edgecolor=colours[0])
ax3.set_xlabel('Tg/100')
ax3.set_ylabel('log(colDen)')
ax3.set_xlim(0,6)
# nH, colDen
ax4 = fig.add_subplot(gs[3,2])
ax4.scatter(np.log10(D200), np.log10(CD200), marker='*', s=10, color=colours[10], label='Td = 200', alpha=1, edgecolor=colours[10])
ax4.scatter(np.log10(D10), np.log10(CD10), marker='*', s=10, color=colours[0], label='Td = 10', alpha=1, edgecolor=colours[0])
ax4.set_xlabel('log(nH)')
ax4.set_ylabel('log(colDen)')
# LEGEND
legend = fig.add_subplot(gs[:,3])
text = ['Td = 10', 'Td = 20', 'Td = 30', 'Td = 40', 'Td = 50', 'Td = 60', 'Td = 70', 'Td = 80', 'Td = 90', 'Td = 100', 'Td = 110', 'Td = 120', 'Td = 130', 'Td = 140', 'Td = 150', 'Td = 160', 'Td = 170', 'Td = 180', 'Td = 190', 'Td = 200']
array = np.arange(0,2,0.1)
for i in range(len(array)):
legend.scatter(0, i, marker='*', s=100, c=colours[numcols-i-1], edgecolor=colours[numcols-i-1])
legend.text(0.3, i-0.25, text[numcols-i-1])
legend.set_xlim(-0.5, 2.5)
legend.set_ylim(0-1, i+1)
legend.axes.get_xaxis().set_visible(False)
legend.axes.get_yaxis().set_visible(False)
gs.tight_layout(fig)
plt.show()