Coloriser les amas dans un Dendogram avec ggplot2
Didzis Elferts a montré comment tracer un dendogramme en utilisant ggplot2 et ggdendro:
dendrogramme horizontal en R avec étiquettes
voici le code:
labs = paste("sta_",1:50,sep="") #new labels
rownames(USArrests)<-labs #set new row names
hc <- hclust(dist(USArrests), "ave")
library(ggplot2)
library(ggdendro)
#convert cluster object to use with ggplot
dendr <- dendro_data(hc, type="rectangle")
#your own labels are supplied in geom_text() and label=label
ggplot() +
geom_segment(data=segment(dendr), aes(x=x, y=y, xend=xend, yend=yend)) +
geom_text(data=label(dendr), aes(x=x, y=y, label=label, hjust=0), size=3) +
coord_flip() + scale_y_reverse(expand=c(0.2, 0)) +
theme(axis.line.y=element_blank(),
axis.ticks.y=element_blank(),
axis.text.y=element_blank(),
axis.title.y=element_blank(),
panel.background=element_rect(fill="white"),
panel.grid=element_blank())
personne Ne sait, comment coloriser les différents groupes? Par exemple, vous voulez avoir 2 Clusters (k=2) colorisée?
4 réponses
la solution serait de tracer l'objet cluster avec plot() puis utilisez la fonction rect.hclust() dessiner des bordures autour de l'amas (nombre de clusters est définie avec le paramètre k=). Si le résultat de rect.hclust() est sauvegardé en tant qu'objet il va faire liste d'observation où chaque élément de liste contient des observations appartenant à chaque cluster.
plot(hc)
gg<-rect.hclust(hc,k=2)
maintenant cette liste peut être convertie en dataframe où la colonne clust contient des noms pour les clusters (dans cet exemple, deux groupes) - les noms sont répété selon la longueur des elemets de la liste.
clust.gr<-data.frame(num=unlist(gg),
clust=rep(c("Clust1","Clust2"),times=sapply(gg,length)))
head(clust.gr)
num clust
sta_1 1 Clust1
sta_2 2 Clust1
sta_3 3 Clust1
sta_5 5 Clust1
sta_8 8 Clust1
sta_9 9 Clust1
nouvelle base de données fusionnée avec label()dendr objet (dendro_data() résultat).
text.df<-merge(label(dendr),clust.gr,by.x="label",by.y="row.names")
head(text.df)
label x y num clust
1 sta_1 8 0 1 Clust1
2 sta_10 28 0 10 Clust2
3 sta_11 41 0 11 Clust2
4 sta_12 31 0 12 Clust2
5 sta_13 10 0 13 Clust1
6 sta_14 37 0 14 Clust2
lors du tracé de l'utilisation du dendrogramme text.df pour ajouter des étiquettes avec geom_text() et utilisez la colonneclust pour les couleurs.
ggplot() +
geom_segment(data=segment(dendr), aes(x=x, y=y, xend=xend, yend=yend)) +
geom_text(data=text.df, aes(x=x, y=y, label=label, hjust=0,color=clust), size=3) +
coord_flip() + scale_y_reverse(expand=c(0.2, 0)) +
theme(axis.line.y=element_blank(),
axis.ticks.y=element_blank(),
axis.text.y=element_blank(),
axis.title.y=element_blank(),
panel.background=element_rect(fill="white"),
panel.grid=element_blank())
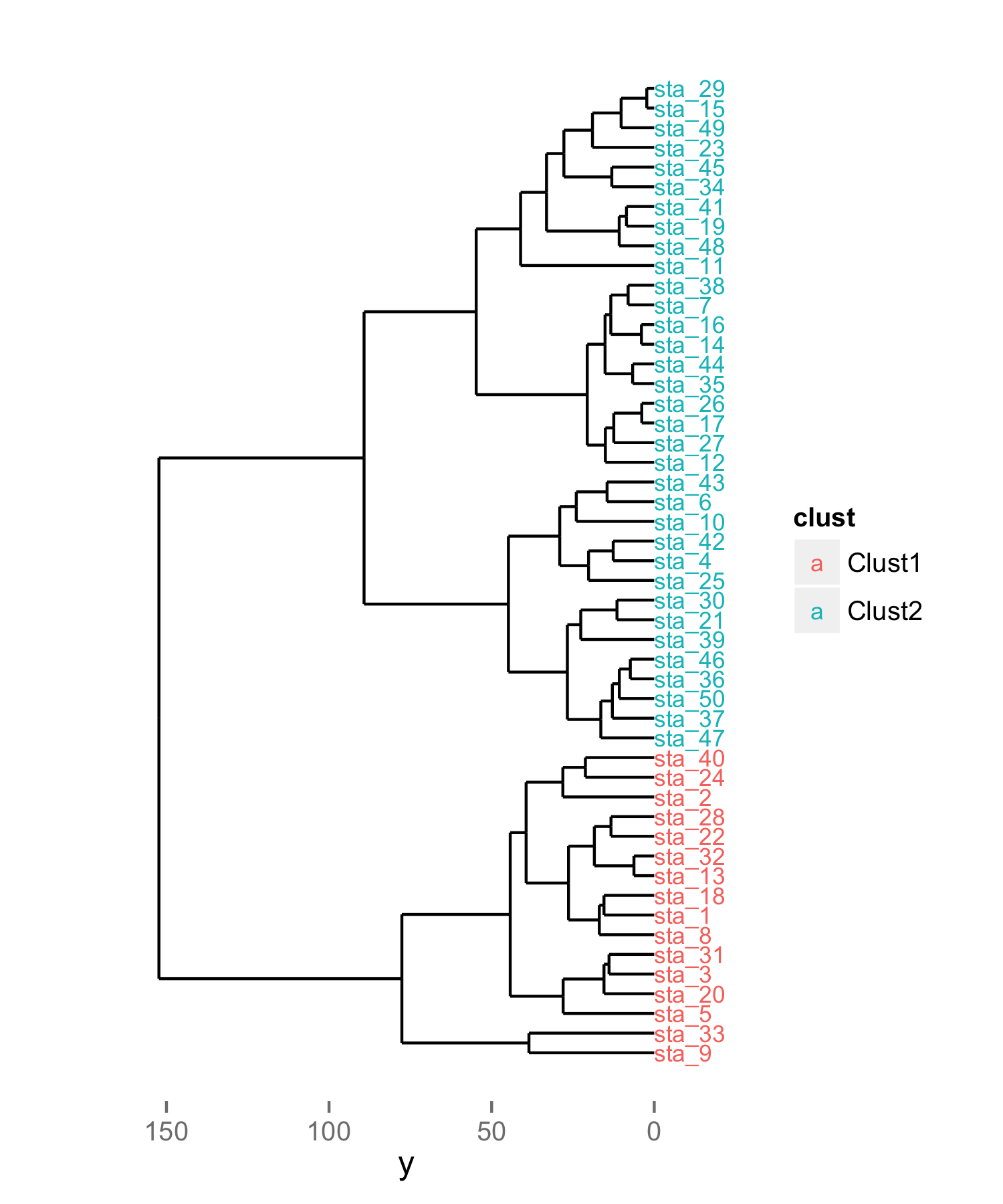
cette approche est très similaire à celle de @DidzisElferts, juste un peu plus simple.
df <- USArrests # really bad idea to muck up internal datasets
labs <- paste("sta_",1:50,sep="") # new labels
rownames(df) <- labs # set new row names
library(ggplot2)
library(ggdendro)
hc <- hclust(dist(df), "ave") # heirarchal clustering
dendr <- dendro_data(hc, type="rectangle") # convert for ggplot
clust <- cutree(hc,k=2) # find 2 clusters
clust.df <- data.frame(label=names(clust), cluster=factor(clust))
# dendr[["labels"]] has the labels, merge with clust.df based on label column
dendr[["labels"]] <- merge(dendr[["labels"]],clust.df, by="label")
# plot the dendrogram; note use of color=cluster in geom_text(...)
ggplot() +
geom_segment(data=segment(dendr), aes(x=x, y=y, xend=xend, yend=yend)) +
geom_text(data=label(dendr), aes(x, y, label=label, hjust=0, color=cluster),
size=3) +
coord_flip() + scale_y_reverse(expand=c(0.2, 0)) +
theme(axis.line.y=element_blank(),
axis.ticks.y=element_blank(),
axis.text.y=element_blank(),
axis.title.y=element_blank(),
panel.background=element_rect(fill="white"),
panel.grid=element_blank())
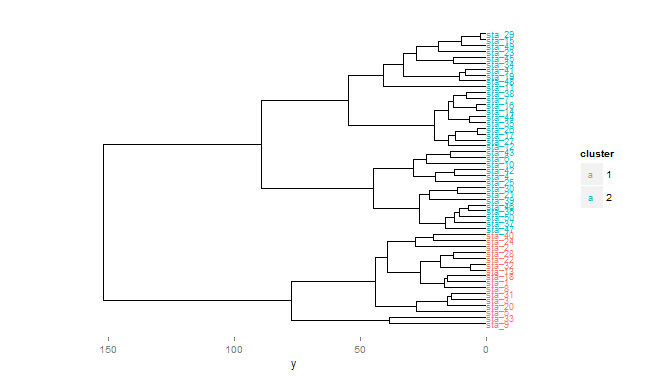
en ajoutant au code de @DidzisElferts et @jlhoward, le dendrogramme lui-même peut être coloré.
library(ggplot2)
library(ggdendro)
library(plyr)
library(zoo)
df <- USArrests # really bad idea to muck up internal datasets
labs <- paste("sta_", 1:50, sep = "") # new labels
rownames(df) <- labs # set new row names
cut <- 4 # Number of clusters
hc <- hclust(dist(df), "ave") # hierarchical clustering
dendr <- dendro_data(hc, type = "rectangle")
clust <- cutree(hc, k = cut) # find 'cut' clusters
clust.df <- data.frame(label = names(clust), cluster = clust)
# Split dendrogram into upper grey section and lower coloured section
height <- unique(dendr$segments$y)[order(unique(dendr$segments$y), decreasing = TRUE)]
cut.height <- mean(c(height[cut], height[cut-1]))
dendr$segments$line <- ifelse(dendr$segments$y == dendr$segments$yend &
dendr$segments$y > cut.height, 1, 2)
dendr$segments$line <- ifelse(dendr$segments$yend > cut.height, 1, dendr$segments$line)
# Number the clusters
dendr$segments$cluster <- c(-1, diff(dendr$segments$line))
change <- which(dendr$segments$cluster == 1)
for (i in 1:cut) dendr$segments$cluster[change[i]] = i + 1
dendr$segments$cluster <- ifelse(dendr$segments$line == 1, 1,
ifelse(dendr$segments$cluster == 0, NA, dendr$segments$cluster))
dendr$segments$cluster <- na.locf(dendr$segments$cluster)
# Consistent numbering between segment$cluster and label$cluster
clust.df$label <- factor(clust.df$label, levels = levels(dendr$labels$label))
clust.df <- arrange(clust.df, label)
clust.df$cluster <- factor((clust.df$cluster), levels = unique(clust.df$cluster), labels = (1:cut) + 1)
dendr[["labels"]] <- merge(dendr[["labels"]], clust.df, by = "label")
# Positions for cluster labels
n.rle <- rle(dendr$segments$cluster)
N <- cumsum(n.rle$lengths)
N <- N[seq(1, length(N), 2)] + 1
N.df <- dendr$segments[N, ]
N.df$cluster <- N.df$cluster - 1
# Plot the dendrogram
ggplot() +
geom_segment(data = segment(dendr),
aes(x=x, y=y, xend=xend, yend=yend, size=factor(line), colour=factor(cluster)),
lineend = "square", show.legend = FALSE) +
scale_colour_manual(values = c("grey60", rainbow(cut))) +
scale_size_manual(values = c(.1, 1)) +
geom_text(data = N.df, aes(x = x, y = y, label = factor(cluster), colour = factor(cluster + 1)),
hjust = 1.5, show.legend = FALSE) +
geom_text(data = label(dendr), aes(x, y, label = label, colour = factor(cluster)),
hjust = -0.2, size = 3, show.legend = FALSE) +
scale_y_reverse(expand = c(0.2, 0)) +
labs(x = NULL, y = NULL) +
coord_flip() +
theme(axis.line.y = element_blank(),
axis.ticks.y = element_blank(),
axis.text.y = element_blank(),
axis.title.y = element_blank(),
panel.background = element_rect(fill = "white"),
panel.grid = element_blank())
Le 2-cluster et 4-les solutions de cluster:
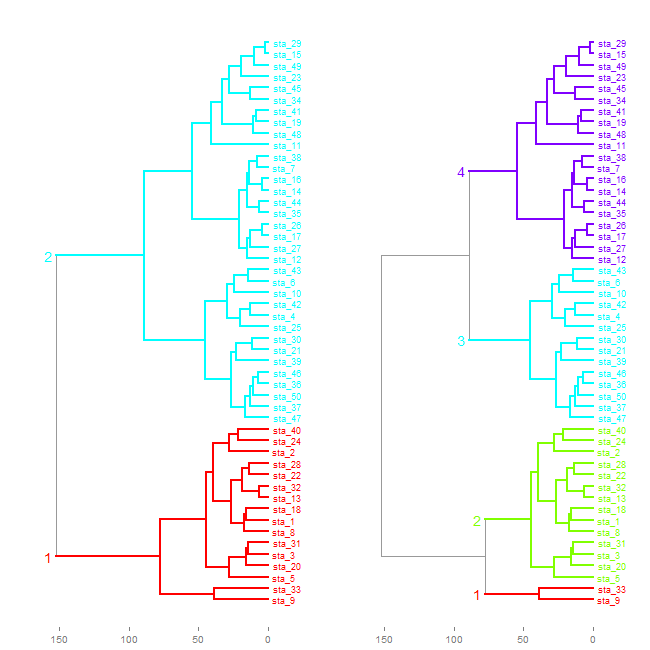
Un court chemin pour atteindre un résultat similaire est d'utiliser le paquet dendextend (dérivé de cette belle vue d'ensemble).
df <- USArrests # really bad idea to muck up internal datasets
labs <- paste("sta_",1:50,sep="") # new labels
rownames(df) <- labs # set new row names
require(magittr)
require(ggplot)
require(dendextend)
dend <- df %>% dist %>%
hclust %>% as.dendrogram %>%
set("branches_k_color", k = 4) %>% set("branches_lwd", 0.7) %>%
set("labels_cex", 0.6) %>% set("labels_colors", k = 4) %>%
set("leaves_pch", 19) %>% set("leaves_cex", 0.5)
ggd1 <- as.ggdend(dend)
ggplot(ggd1, horiz = TRUE)
Note: l'ordre des États est légèrement différent par rapport à ceux ci - dessus-pas vraiment changer l'interprétation cependant.
