Graphique de surface en 3d avec coordonnées xyz
j'espère que quelqu'un avec de l'expérience peut aider dans la façon dont on prépare les fichiers de forme à partir des données xyz. Un grand exemple d'un ensemble de données bien préparé peut être vu ici pour la comète Churyumov-Gerasimenko, bien que les étapes précédentes dans la création du fichier de forme ne sont pas fournis.
j'essaie de mieux comprendre comment appliquer une surface à un ensemble donné de coordonnées XYZ. Les coordonnées cartésiennes sont droites. en avant avec le paquet R "rgl", cependant les formes qui s'enroulent autour semblent plus difficiles. J'ai trouvé le paquet r geometry , qui fournit une interface aux fonctions QHULL . J'ai essayé d'utiliser ceci pour calculer les facettes triangulées de Delaunay, que je peux ensuite tracer dans rgl . Je suis incapable de comprendre certaines des options associées à la fonction delaunayn pour éventuellement contrôler les distances maximales que ces facettes sont calculées. J'espère que quelqu'un ici pourrait avoir quelques idées sur l'amélioration de la construction de la surface à partir des données xyz.
exemple utilisant L'ensemble de données" Stanford bunnny":
library(onion)
library(rgl)
library(geometry)
data(bunny)
#XYZ point plot
open3d()
points3d(bunny, col=8, size=0.1)
#rgl.snapshot("3d_bunny_points.png")
#Facets following Delaunay triangulation
tc.bunny <- delaunayn(bunny)
open3d()
tetramesh(tc.bunny, bunny, alpha=0.25, col=8)
#rgl.snapshot("3d_bunny_facets.png")

cette réponse me fait croire qu'il pourrait y avoir un problème avec la mise en œuvre de Qhull. En outre, j'ai maintenant essayé divers paramètres (par exemple delaunayn(bunny, options="Qt") ) avec peu d'effet. Les options de Qhull sont décrites ici
Edit:
voici un exemple Supplémentaire (plus simple) d'une sphère. Même ici, le calcul des facettes ne trouve pas toujours les sommets voisins les plus proches (si vous faites tourner la boule vous verrez quelques facettes traverser l'intérieur).
library(rgl)
library(geometry)
set.seed(1)
n <- 10
rho <- 1
theta <- seq(0, 2*pi,, n) # azimuthal coordinate running from 0 to 2*pi
phi <- seq(0, pi,, n) # polar coordinate running from 0 to pi (colatitude)
grd <- expand.grid(theta=theta, phi=phi)
x <- rho * cos(grd$theta) * sin(grd$phi)
y <- rho * sin(grd$theta) * sin(grd$phi)
z <- rho * cos(grd$phi)
set.seed(1)
xyz <- cbind(x,y,z)
tbr = t(surf.tri(xyz, delaunayn(xyz)))
open3d()
rgl.triangles(xyz[tbr,1], xyz[tbr,2], xyz[tbr,3], col = 5, alpha=0.5)
rgl.snapshot("ball.png")

3 réponses
Voici une approche utilisant l'estimation de la densité des grains et la fonction contour3d de misc3d . J'ai joué jusqu'à ce que je trouve une valeur pour levels qui a fonctionné décemment. Ce n'est pas parfaitement précis, mais vous pouvez être en mesure de modifier les choses pour obtenir une surface meilleure, plus précise. Si vous avez plus de 8 Go de mémoire, alors vous pouvez augmenter n au-delà de ce que j'ai fait ici.
library(rgl)
library(misc3d)
library(onion); data(bunny)
# the larger the n, the longer it takes, the more RAM you need
bunny.dens <- kde3d(bunny[,1],bunny[,2],bunny[,3], n=150,
lims=c(-.1,.2,-.1,.2,-.1,.2)) # I chose lim values manually
contour3d(bunny.dens$d, level = 600,
color = "pink", color2 = "green", smooth=500)
rgl.viewpoint(zoom=.75)

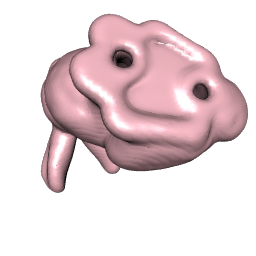
l'image de droite vient du bas, juste pour montrer une autre vue.
vous pouvez utiliser une plus grande valeur pour n dans kde3d mais cela prendra plus de temps, et vous pouvez manquer de mémoire vive si le tableau devient trop grand. Vous pouvez également essayer une bande passante différente (par défaut utilisé ici). J'ai pris cette approche de calcul et affichage Isosurfaces dans R-Feng & Tierney 2008 .
approche isosurface très similaire utilisant le paquet Rvcg :
library(Rvcg)
library(rgl)
library(misc3d)
library(onion); data(bunny)
bunny.dens <- kde3d(bunny[,1],bunny[,2],bunny[,3], n=150,
lims=c(-.1,.2,-.1,.2,-.1,.2)) # I chose lim values manually
bunny.mesh <- vcgIsosurface(bunny.dens$d, threshold=600)
shade3d(vcgSmooth(bunny.mesh,"HC",iteration=3),col="pink") # do a little smoothing

Puisqu'il s'agit d'une approche basée sur l'estimation de la densité, nous pouvons en tirer un peu plus en augmentant la densité du lapin. J'utilise aussi n=400 ici. Le coût est une augmentation significative du temps de calcul, mais la surface résultante est un lièvre mieux:
bunny.dens <- kde3d(rep(bunny[,1], 10), # increase density.
rep(bunny[,2], 10),
rep(bunny[,3], 10), n=400,
lims=c(-.1,.2,-.1,.2,-.1,.2))
bunny.mesh <- vcgIsosurface(bunny.dens$d, threshold=600)
shade3d(vcgSmooth(bunny.mesh,"HC",iteration=1), col="pink")

il existe des méthodes de reconstruction de surface meilleures et plus efficaces (p. ex. croûte de puissance, reconstruction de surface de Poisson, algorithme à pivot sphérique), mais je ne sais pas si elles ont été mises en œuvre en R.
voici un post de débordement de pile pertinent avec quelques bonnes informations et des liens à vérifier (y compris des liens vers le code): algorithme robuste pour la surface reconstruction à partir de 3D point cloud? .
je pense avoir trouvé une solution possible en utilisant le paquet alphashape3d . J'ai dû jouer un peu pour obtenir une valeur acceptable pour alpha , qui est liée aux distances dans l'ensemble de données donné (par exemple sd de bunny m'a donné un aperçu). Je suis toujours en train d'essayer de trouver comment mieux contrôler la largeur des lignes dans les sommets et les bords afin de ne pas dominer la parcelle, mais cela est probablement lié aux paramètres dans rgl .
Exemple:
library(onion)
library(rgl)
library(geometry)
library(alphashape3d)
data(bunny)
apply(bunny,2,sd)
alphabunny <- ashape3d(bunny, alpha = 0.003)
bg3d(1)
plot.ashape3d(alphabunny, col=c(5,5,5), lwd=0.001, size=0, transparency=rep(0.5,3), indexAlpha = "all")

Edit:
ce N'est qu'en ajustant la fonction plot.ashape3d que j'ai pu enlever les arêtes et les sommets:
plot.ashape3d.2 <- function (x, clear = TRUE, col = c(2, 2, 2), byComponents = FALSE,
indexAlpha = 1, transparency = 1, walpha = FALSE, ...)
{
as3d <- x
triangles <- as3d$triang
edges <- as3d$edge
vertex <- as3d$vertex
x <- as3d$x
if (class(indexAlpha) == "character")
if (indexAlpha == "ALL" | indexAlpha == "all")
indexAlpha = 1:length(as3d$alpha)
if (any(indexAlpha > length(as3d$alpha)) | any(indexAlpha <=
0)) {
if (max(indexAlpha) > length(as3d$alpha))
error = max(indexAlpha)
else error = min(indexAlpha)
stop(paste("indexAlpha out of bound : valid range = 1:",
length(as3d$alpha), ", problematic value = ", error,
sep = ""), call. = TRUE)
}
if (clear) {
rgl.clear()
}
if (byComponents) {
components = components_ashape3d(as3d, indexAlpha)
if (length(indexAlpha) == 1)
components = list(components)
indexComponents = 0
for (iAlpha in indexAlpha) {
if (iAlpha != indexAlpha[1])
rgl.open()
if (walpha)
title3d(main = paste("alpha =", as3d$alpha[iAlpha]))
cat("Device ", rgl.cur(), " : alpha = ", as3d$alpha[iAlpha],
"\n")
indexComponents = indexComponents + 1
components[[indexComponents]][components[[indexComponents]] ==
-1] = 0
colors = c("#000000", sample(rainbow(max(components[[indexComponents]]))))
tr <- t(triangles[triangles[, 8 + iAlpha] == 2 |
triangles[, 8 + iAlpha] == 3, c("tr1", "tr2",
"tr3")])
if (length(tr) != 0)
rgl.triangles(x[tr, 1], x[tr, 2], x[tr, 3], col = colors[1 +
components[[indexComponents]][tr]], alpha = transparency,
...)
}
}
else {
for (iAlpha in indexAlpha) {
if (iAlpha != indexAlpha[1])
rgl.open()
if (walpha)
title3d(main = paste("alpha =", as3d$alpha[iAlpha]))
cat("Device ", rgl.cur(), " : alpha = ", as3d$alpha[iAlpha],
"\n")
tr <- t(triangles[triangles[, 8 + iAlpha] == 2 |
triangles[, 8 + iAlpha] == 3, c("tr1", "tr2",
"tr3")])
if (length(tr) != 0)
rgl.triangles(x[tr, 1], x[tr, 2], x[tr, 3], col = col[1],
, alpha = transparency, ...)
}
}
}
alphabunny <- ashape3d(bunny, alpha = c(0.003))
plot.ashape3d.2(alphabunny, col=5, indexAlpha = "all", transparency=1)
bg3d(1)

le paquet Rvcg mis à jour à la version 0.14 en juillet 2016, et la reconstruction de surface à rotule a été ajoutée. La fonction est vcgBallPivoting :
library(Rvcg) # needs to be >= version 0.14
library(rgl)
library(onion); data(bunny)
# default parameters
bunnybp <- vcgBallPivoting(bunny, radius = 0.0022, clustering = 0.2, angle = pi/2)
shade3d(bunnybp, col = rainbow(1000), specular = "black")
shade3d(bunnybp, col = "pink", specular = "black") # easier to see problem areas.
le pivotement de la bille et les paramètres par défaut ne sont pas parfaits pour le lapin de Stanford (comme noté par cuttlefish44 dans les commentaires radius = 0.0022 does mieux que la valeur par défaut radius = 0 ), et vous êtes laissé avec quelques lacunes dans la surface. Le lapin actuel a 2 trous dans la base et quelques limitations de balayage contribuent à quelques autres trous (comme mentionné ici: https://graphics.stanford.edu/software/scanview/models/bunny.html ). Vous pouvez trouver de meilleurs paramètres, et il est assez rapide d'utiliser vcgBallPivoting (~0.5 secondes sur ma machine), mais des efforts / méthodes supplémentaires peuvent être nécessaires pour fermer les trous.

